На данной странице представлен практикум по созданию HMM-профиля семейства белков и проверке его работы.
1) Выбор домена:
Для выбора белка была создана таблица со всеми известными доменами (буквы A-Z и цифры) из базы данных Pfam. При помощи скрипта полученная таблица была отфильтрована по рекомендованным параметрам:
- число последовательностей в full менее 300, но более 40
- cредняя длина домена менее 150
- cреднее сходство (identity) более 40%
- Средний процент покрытия последовательности белка доменом (coverage) менее 40%
- Таблица
Из полученной отфильтрованной таблицы мной был выбран домен GAGA (GAGA factor), связывающийся с консенсусным сайтом 5'-GAGAG-3' на ДНК и содержащий ядро цинкового пальца: цистеин-гистидин. Характеристики домена:
- AC: PF09237
- число последовательностей в: seed – 2; full – 186; uniprot – 378
- cредняя длина домена – 46.1 а/к
- cреднее сходство (identity) – 42%
- Средний процент покрытия последовательности белка доменом (coverage) – 9.8%
Для данного домена известны 26 различных архитектур. Для изучения была выбрана двудоменная архитектура, состоящая из домена GAGA и BTB (сначала идет BTB, затем GAGA) и встречающаяся в 77 последовательностях из 186 известных. Ее схема на рисунке 1:

2) Получение последовательностей и выравнивания:
Все последовательности, содержащие домен GAGA, били скачаны в файл full.fasta. С помощью скрипта из полученного файла был составлен список их АС. Их получилось 170 из-за того, что в скрите использовались множества и дубликаты были удалены.
Из раздела "Domain organisation" были скопированы описания с AC последовательностей, соответствующих выбранной двудоменной архитектуре. При помощи скрипта из полученного файла был составлен список АС последовательностей, соответствующих выбранной архитектуре. Далее при помощи скрипта из файла full.fasta были выгружены последовательности, соответствующие выбранной архитектуре, по АС из списка. Результат в fasta формате.
Полученный fasta файл был выровнен в Jalview, после чего над выравниванием была произведена ревизия: удалены субъективно-лишние последовательности и фрагменты выравнивания до первого домена и после второго, также были удалены все полностью-идентичные последовательности. Длина профиля составила 382 а/к, взято 24 последовательности. Результат в файле, а также на рисунке 2:
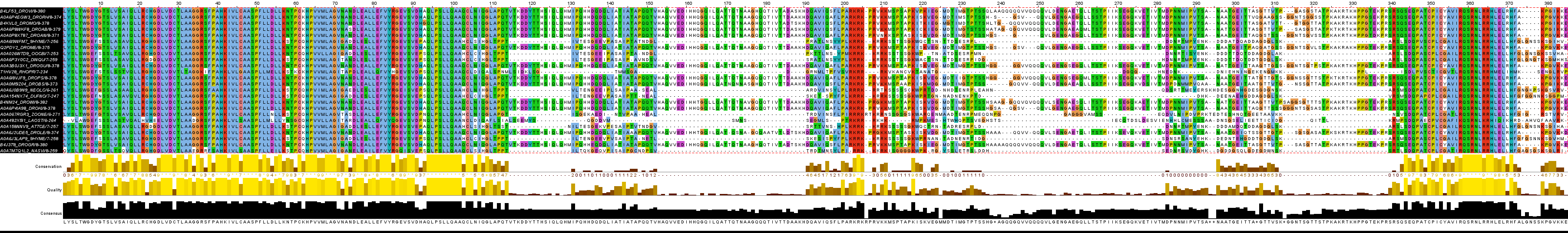
3) Создание HMM-профиля:
Для создания HMM-профиля полученный fasta файл с отредактированными последовательностями был загружен на сервер кодомо, после чего была выполнена следующая серия команд:
hmm2calibrate profile
hmmsearch --cpu=1 profile full.fasta &> log.txt
hmm2search --cpu=1 profile full.fasta &> log2.txt
hmm2build строит HMM-профиль, результат ее работы по ссылке. hmm2calibrate калибрует его. hmmsearch иhmm2search производит поиск изучаемой двудоменной архитектуры в переданных последовательностях с порогом E-value 0.01. Результат поиска в файлах: 1 и 2.
Из полученного log файла была выгружена таблица с результатами поиска. В переданных последовательностях было найдено 158 последовательностей с E-value менее 0.01, в которых можно предположить наличие изучаемой двудоменной стрктуры. В двух последовательностях E-value было меньше порога (они не внесены в таблицу). Еще в десяти последовательносях полученный профиль не был найден, возможно, это произошло из-за слишком жесткой ревизии.
С помощью скрипта информация из полученной таблицы, файла full.fasta с последовательностями всех белков с изучаемым доменом, файла с АС этих последовательностей, файла c АС белков с изучачаемой двудоменной архитектурой и файла c AC белков, использованных для построения учебного выравнивания была объединена в общую таблицу (tsv формат, xlsx формат). В колонках: АС белка, содержит ли изучаемую двудоменную архитектуру, исользовался ли для построения HMM профиля, был ли найден hmm2search, score находки, E-value находки, длина белка.
4) Анализ HMM-профиля:
Из файла, полученного командой hmm2search, с помощью скрипта была получена таблица, содержащая информацию о 145 находках: AC, Domain, seq-f, seq-t, hmm-f, hmm-t, score, E-value. Также был добавлен столбец true с ответами yes – если AC встретился в списке белков с данной архитектурой, или no в противном случае.
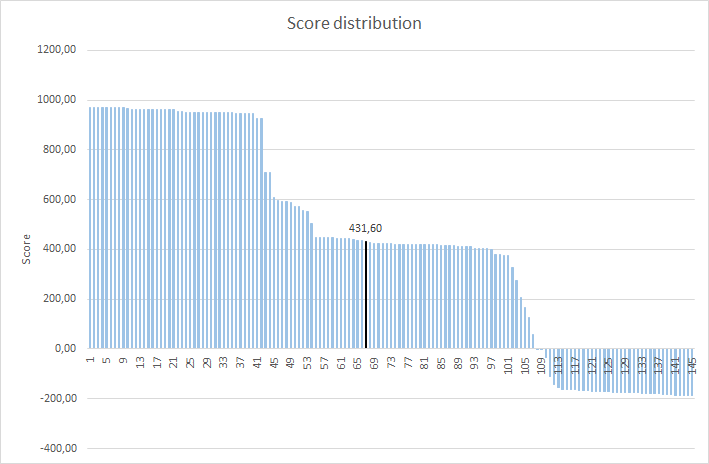
Данная таблица была экспортирована в Excel, после чего с помощью формул для нее были посчитаны значения specificity, 1 - sensitivity и F1. Таблица с данными, формулами и графиками доступна по ссылке. По полученным данным были построены следующие 3 графика:
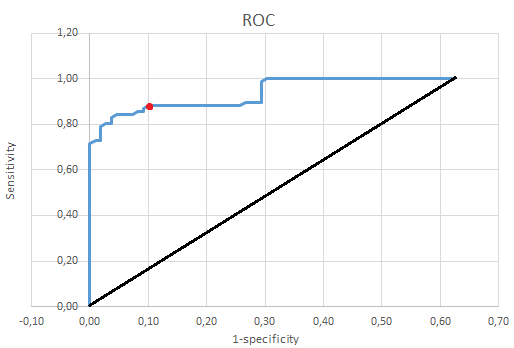
1) ROC-кривая. По полученным specificity и 1 - sensitivity была построена данная кривая. Для оценки наиболее подходящего порога была проведена прямая, соединящая конечные точки данной кривой и найден максимум длины от кривой до этой прямой. Координаты: 1 - sensitivity – 0.1; specificity – 0.88. По полученным значениям был найден порог по score последовательности для отнесения к изучаемой двудоменной архитектуре – 430.
2) Распределение весов последовательностей. Для визуализации полученного по ROC-кривой порога было построено распределение весов последовательностей. Полученный порог прошли 67 последовательностей из 77, которые обладают изучаемой двудоменной архитектурой по мнению Pfam. Это говорит о возможной слишком жесткой ревизии для построения профиля. Также хочется отметить визуальную неочевидность выбора данного порога.
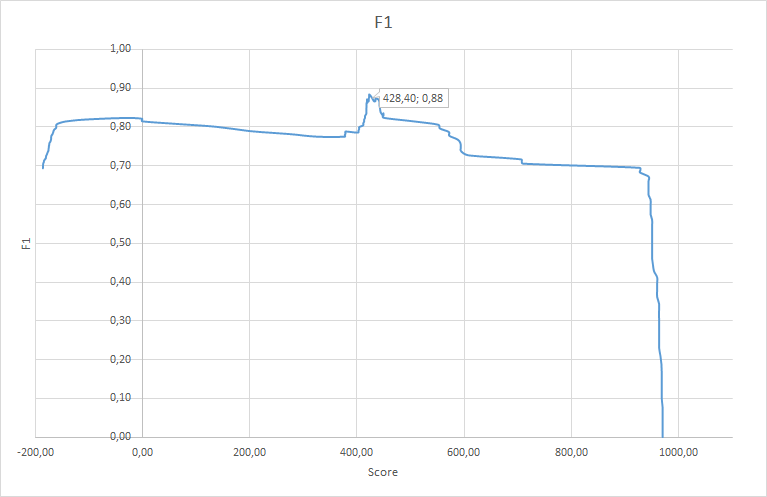
3) F1-кривая. Для визуализации полученного порога, а также подтверждения его значения по ROC-кривой была построена F1-кривая. Локальный максимум данного графика, соответствует оптимальному соотношению sensitivity и specificity. Значение порогового веса последовательнотси для отнесения к изучаемой двудоменной архитектуры незначительно отличается от такового полученного по ROC-кривой и составляет 428.4.