Поиск в нуклеотидных банках по аннотации. Выравнивание геномов
Для выполнения задания мне требовалось произвести сравнение blastn и megablast по построенным ими картам локального сходства. Для этого я выбрал две последовательности, найденные в категории референсных бактериальных геномов в NCBI.
Выбранные последовательности почвенных бактерий Bacillus licheniformis (NZ_CP140161.1) и Bacillus velezensis (NC_009725.2) имеют уровень полных геномов.
Параметры blastn и megablast были использованы по умолчанию.
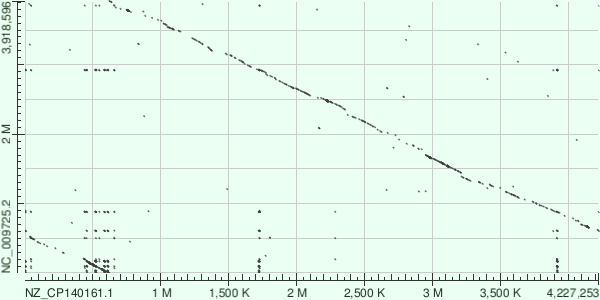
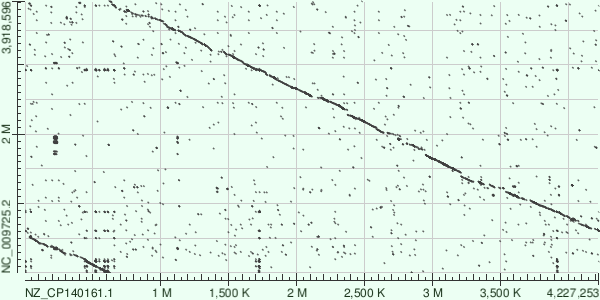
Выдача Megablast содержит меньше информации, в том числе на ней нет побочных выравниваний, не имеющих эволюционного смысла, однако теряется и часть полезной информации (например, на участке 2,6-2,9М на Megablast нельзя найти консервативные участки, а на участке 2,35М не видна дупликация), поэтому я решил привести анализ карты на примере blastn.
- Геном кольцевой, были выбраны разные места начала секвенирование и разные направления
- На участке 1,3-1,4М произошла крупная делеция/вставка
- На участке 2,1-2,15М наблюдается небольшая делеция/вставка, а на участке 2,35М небольшая дупликация
- На участке 2,75М, возможно, имела место небольшая транслокация
- Присутствует значительное количество повторяющихся участков, создающих шум
Различия в геномах двух бактерий Bacillus не очень существенны: возможно, если взять два других вида внутри данного рода можно получить значительно более различающиеся между собой геномы, содержащие заметные инверсии, дупликации и транслокации. Выдача программы Megablast удобнее для восприятия (отсутствие шума), но отличается низкой информативностью по сравнению с blastn, из-за чего для поиска гомологов выбирают последнюю.


