Нуклеотидный BLAST
Задание 1. Ген, кодирующий δ-субъединицу АТФ-синтазы
Для выполнения последующих заданий мне потребуется нуклеотидная последовательность гена, кодирующего δ-субъединицу АТФ-синтазы и некоторая информация об этой последовательности.
Идентификатор белка: XP_037238476.1
Идентификатор нуклеотидной записи: XM_037382579.1
XM_037382579.1.fasta - FASTA-файл, содержащий участок хромосомы, представленный искомым геном.
Последовательность аминокислот: файл
Задание 2. Разные варианты BLAST для фрагмента ДНК
Далее мне нужно было осуществить поиск BLAST по последовательностям геномов для последовательности гена δ-субъединицы АТФ-синтазы из таксона, представленного на выбор. Так как Falco rusticolus относится ко Вторичноротым, на выбор были семейства Кошачьи и Собачьи. Я решил выбрать первый таксон. В базе данных RefSeq Genome Database было 15 геномных сборок.
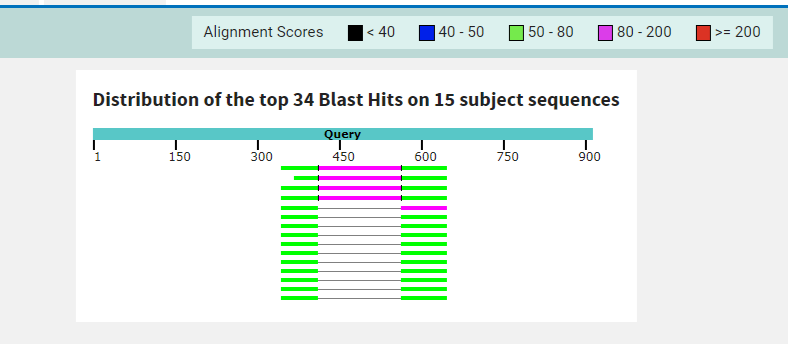
Алгоритм blastn – Уствновленные параметры: E-Value – 0.05, длина слова – 11. Алгоритм использовался, так как хорошо подходит для поиска нуклеотидных последовательностей небольшого размера среди неблизкородственных организмов. Поиск выдал 15 результатов. С данным порогом на E-Value я и не ожидал получить много находок. Результаты выдачи можно скачать в файле. Значение E-Value находок не превышало 3e-09 и процент идентичности был довольно высок (72.90% - 81.93%), хотя процент покрытия для локального выравнивания был довольно низок (обычно 16%, макс. 32%). Исходя из этих данных, можно предположить, что программа хорошо справилась со своей задачей. Если увеличить E-Value, например, до 10, можно ожидать значительное увеличение числа находок, но они также будут скорее всего иметь очень низкий процент покрытия и идентичности и сравнительно маленькую длину.
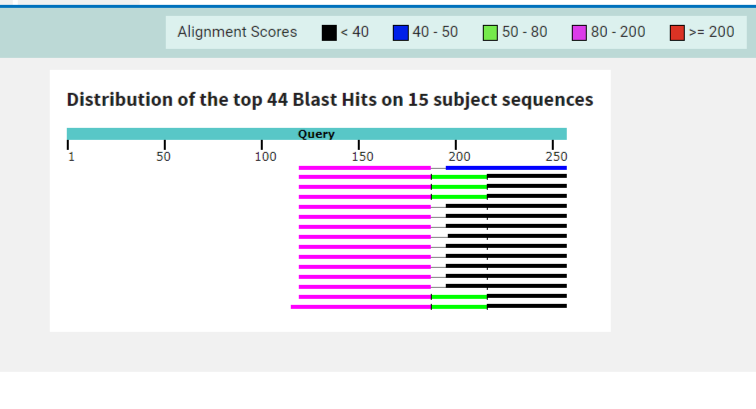
Алгоритм tblastn - Уствновленные параметры: E-Value – 0.05, длина слова – 5. На запрос подается последовательность белка. Белковая последовательность сопоставляется с транслированными последовательностями из RefSeq, такой тип алгоритма в теории должен давать значительно более точный результат. Поиск выдал те же 15 результатов. Их процент покрытия был уже значительно больше (50%-53%). Значение E-Value при этом не превышало 5e-18. Процент идентичности упал, но незначительно (почти у всех 67.65%). Алгоритм tblastn подтвердил, что 15 найденных blastn последовательностей имеют выраженные высококонсервативные участки.
Задание 3. Помск в геноме эукариота гены основных рибосомальных РНК по далекому гомологу
Для поиска гомологов 16S рРНК и 23S рРНК кишечной палочки мне потребовался файл с полным геномом кречета. Архив, скачанный напрямую с сайта NCBI, распаковывался с ошибкой. Чтобы получить файл с геномом, я воспользовался командой NCBI Datasets:
datasets download genome accession GCF_015220075.1
Далее я проиндексировал свой геном с помощью команды:
makeblastdb -in GCF_015220075.1_bFalRus1.pri_genomic.fna -dbtype nucl
Для каждой последовательности был запущен алгоритм blastn:
blastn -task blastn -query 16s.fasta -db GCF_015220075.1_bFalRus1.pri_genomic.fna -out 16s.out -outfmt 7
blastn -task blastn -query 23s.fasta -db GCF_015220075.1_bFalRus1.pri_genomic.fna -out 23s.out -outfmt 7
blastn -task blastn -query 16s.fasta -db GCF_015220075.1_bFalRus1.pri_genomic.fna -out 16st.out
blastn -task blastn -query 23s.fasta -db GCF_015220075.1_bFalRus1.pri_genomic.fna -out 23st.out
Я выбрал blastn, потому что сравнивал небольшие последовательности дальнородственных организмов.
Результаты выдачи:
рРНК требуется для синтеза белка и поддержания структуры рибосом. 16S рРНК необходима для связывания малой субъединицы рибосомы с мРНК во время процесса трансляции. 23S рРНК выполняет роль катализатора для роста пептидной цепи в ходе трансляции. Найденные гомологи в геноме кречета выполняют аналогичные функции.
Для 16S рРНК нашлось 6 находок, 0 относятся к гомологам, для 23S рРНК – 18 находок, гомологами являются 2 из них.


