Практикум 10. Мотивы в белках, паттерны и PSSM для их поиска
Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
В качестве домена для исследования я выбрал семейство – Домен Кунитца (бычий панкреатический ингибитор трипсина). Домен отвечает требованиям задачи: количество белков в SwissProt – 398, а выравнивание “вручную” состоит из 99 последовательностей. Характерен для ингибиторов протеаз, преимущественно ингибиторов сериновых протеаз. Его структура представляет собой α+β-складчатую архитектуру с высоким содержанием дисульфидных связей.
Модельный объект исследований: BPTI — широко изученная модельная структура, используемая для анализа механизмов ингибирования протеаз.
Родственные белки: Некоторые представители данного семейства демонстрируют структурное сходство с антикоагулянтным пептидом клещей (TAP, P17726) — высокоселективным ингибитором фактора Xa в системе свертывания крови
Загрузив выравнивание в Jalview, я постепенно понижал порог идентичности, чтобы найти мотив. За мотив было решено принять участок выравнивания 50-58: F.YG{2}C.GN На глаз IC предсказать было трудно: кажется, что мотив слишком мал, разнообразен по аминокислотам и раздроблен колонками, чтобы можно было однозначно отринуть случайное совпадение, однако поиск в Jalview выдал 34 результата, что может указывать на консервативность участка.

Переведём паттерн в формат, воспринимаемый просайтом: F-x-Y-G(2)-C-x-G-N Поиск по SwissProt через Prosite выдал 349 находок среди 294 последовательностей. Последовательности я скачал в фаста-формате по ссылке на UniProt. Большинство из них действительно принадлежат домену Кунитца, остальные же, вероятно, сходным по структуре и свойствам белкам. В списке находок было много предшественников бета-амилоида среди разных животных.
Задание 2. Построение филогенетического дерева в Jalview
Построим дерево методом NJ, опираясь на BLOSUM62. Выбранная клада отмечена на рисунке серым цветом. За консервативный для клады мотив примем [TS].[QKN]CE[EGR] c 45-49 букву. Проведя поиск последовательностей, получим 7 находок, которые и составляют выбранную кладу, то есть паттерн действительно специфичен для данной клады белков.
Рис. 3. Разделение по кладам, выбранная горит чёрным цветом Рис. 4. Клада в отдельном окне Рис. 5. Мотив, специфичный для кладыЗадание 3. PSI-Blast
Недолго думая, я решил выбрать последний белок – Q67XL4. В UniProt находим, что это хлоропластный белок резушки Arabidopsis thaliana (функция, увы, не описана), содержащий РНК-связывающий CRM-домен At3g25440. Запустим PSI-Blast, приняв за порог 0,005. Выдача стабилизировалась на четвертой итерации, как видно из таблицы, значит белки семейства обладают сходной структурой. Функция (связывание РНК) также совпадает для всех находок, из чего можно сделать вывод о консервативности этого свойства внутри домена.
Табл. 1. Итерации PSI-BLASTЗадание 4. Ищем мотив de nuvo
Из UniProt был получен файл с последовательностями 398 аннотированных белков. В Jalview средствами Muscle построим выравнивание, далее избавимся от избыточности. Выравнивание строилось более 10 минут, но с учётом количества сиквенсов и их длин это не удивительно. Уменьшение порога всего на 1% оставило лишь 64 последовательности, они были перевыровнены средствами Mafft (чтобы оценить на глаз, нашлось два достаточно консервативных участка). Результат сохранил в fasta формате. С помощью команды sed убрал гэпы из выравнивания.Команды:
meme t.fa -o memres -minw 4 -maxw 10 -nmotifs 2
fimo meme.txt ../fullfa.fa
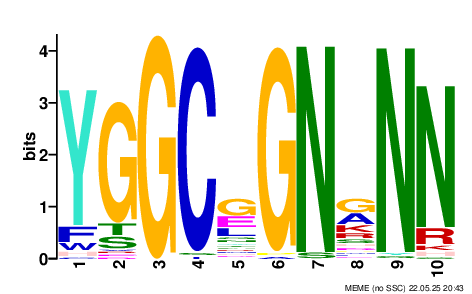
Задание 5. Представленность GATC в геноме галофильной археи
Геном моей археи Haloprofundus salinisoli был изучен на представленность нуклеотидов. На вход алгоритму нужно дать также всевозможные перестановки (без повторения) из четырёх нуклеотидов.
cbcalc -s permutations.txt -M -o results.tsv GCF_020097815.1_ASM2009781v1_genomic.fna
На выходе получаем tsv файл, по которому можно построить гистограмму на Python. Из гистаграммы видно, что набор GATC недопредставлен в геноме археи.



