Практикум 7. Трансмембранные белки
Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Белок – S-layer deinoxanthin binding complex (Белковый комплекс, связывающий дейноксантин поверхностного слоя)
Type: Transmembrane (Трансмембранный)
Class: Beta-barrel transmembrane (белок, имеющий β-листы в трансмембранной части)
Superfamily: S-layer homology domain
Family: S-layer homology domain
Species: Deinococcus radiodurans
Localization: Bacterial Gram-negative outer membrane (внешняя мембрана грамм-отрицательной бактерии)
PDB: 7zgx
Uniprot: SLPA_DEIRA
Кооринаты из OPM доступны здесь.
Играет важную роль в структурной организации и целостности клеточной оболочки, соединяя внешнюю мембрану с пептидогликановым слоем.
Является очень распространенной молекулой в клеточной оболочке D. radiodurans, хотя и не является основным компонентом S-слоя.
Связывает каротиноид дейноксантин, сильный защитный антиоксидант, специфичный для этой бактерии, и может быть частью первой линии защиты от ультрафиолетового излучения, особенно при высушивании.
Способен транспортировать заряженные аминокислоты, такие как Lys, Arg и Glu; большой размер пор указывает на физиологическую важность комплекса SDBC (S-layer deinoxanthin-binding complex), способствующего обмену веществами, включая питательные вещества, с окружающей средой.
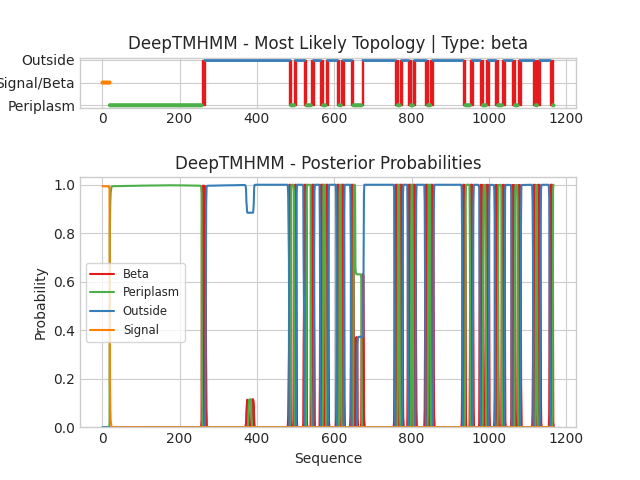
Возьмём белковую последовательность из UniProt и загрузим её в DeepTMHMM.
Выдача алгоритмаКоординаты из DeepTMHMM:
(1, '258-267'), (2, '484-488'), (3, '497-501'), (4, '523-528'), (5, '540-548'), (6, '564-571'), (7, '578-584'), (8, '607-613'), (9, '618-625'), (10, '644-649'), (11, '671-675'), (12, '757-764'), (13, '770-776'), (14, '791-798'), (15, '804-810'), (16, '836-842'), (17, '849-856'), (18, '932-939'), (19, '952-958'), (20, '976-984'), (21, '993-1000'), (22, '1016-1023'), (23, '1034-1041'), (24, '1059-1067'), (25, '1075-1082'), (26, '1115-1121'), (27, '1125-1133'), (28, '1157-1165')
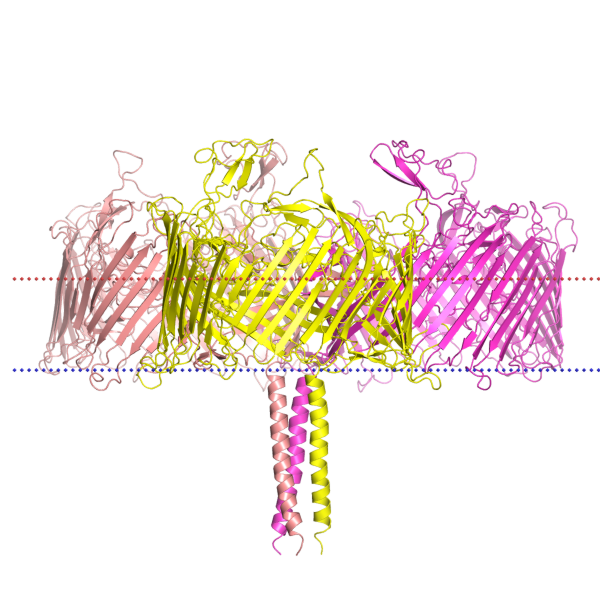
Cиние и красные шарики, ограничивающие изображение трансмембранных белков, обозначают границы мембраны:
Синие шарики – соответствуют внутриклеточной (цитоплазматической) стороне мембраны.
Красные шарики – соответствуют внеклеточной стороне мембраны.
C помощью скрипта на Python были посчитаны полные совпадения и условные (различаются не более, чем на две позиции в обе стороны).
Статистика
Вывод: результаты предсказаний программы не полностью точны. Количество трансмембранных участков отличается: программой предсказано 28, в то время как по экспериментальным данным их 30 в каждой субъединице. Координаты трех участков совпадают полностью, еще 16 условно (см. выше), многие предсказанные программой участки и участки из OPM перекрываются, но в точности не совпадают (отличат на несколько позиций). Два участка пропущены (Участки 2 и 3 из OPM не были предсказаны в выдаче). Можно сказать, что программа неплохо справилась со своей задачей, предсказав примерное (с ошибкой в один или несколько позиций) расположение трансмембранных участков. Подойдёт ли такой результат исследователю зависит от работы, которую он проводит: если ему нужно примерно оценить количество и расположение трансмембранных участков, то DeepTMHMM для этого подойдёт, если же нужна точность вплоть до конкретной позиции, то стоит рассмотреть иные методы.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
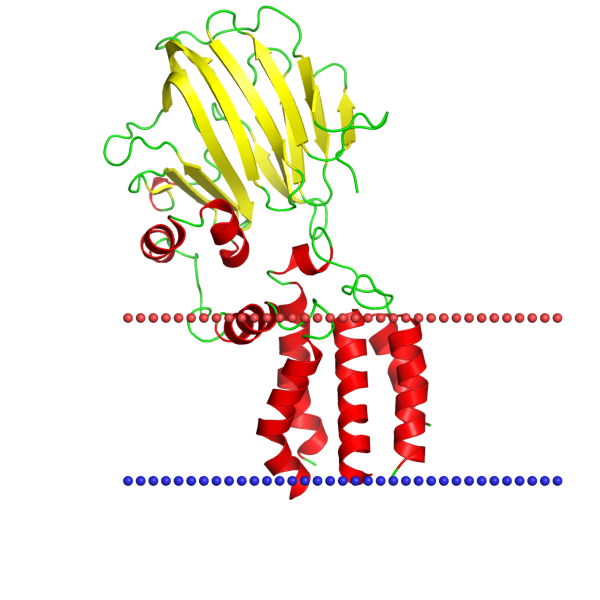
Мне был выдан белок 5y83 – Membrane protein insertase YidC (Мембранная инсертаза YidC).
Type: Transmembrane
Class: Alpha-helical polytopic
Superfamily: Cytochrome oxidase biogenesis family
Family: OxaA/YidC
Species: Thermotoga maritima
Localization: Bacterial Gram-negative inner membrane (внутренняя мембрана грамм-отрицательной бактерии)
UniProt: YIDC_THEMA
Необходим для встраивания и/или правильного сворачивания и/или формирования комплекса интегральных мембранных белков внутри мембраны. Участвует в интеграции мембранных белков, которые встраиваются как зависимо, так и независимо от Sec-транслоказы, а также, по крайней мере, некоторых липопротеидов. Способствует сворачиванию многопроходящих мембранных белков.
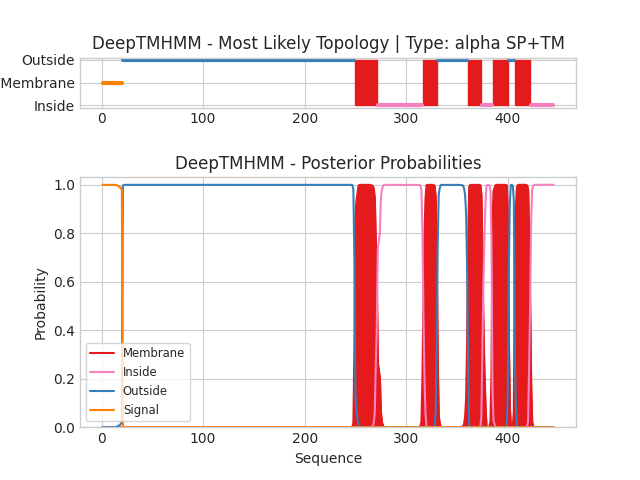
Координаты трансмембранных участков:
1( 249-268), 2( 316-331), 3( 358-372), 4( 390-402), 5( 404-420)
Аминокислотный сиквенс вновь возьмём из Uniprot.
Выдача доступна поссылке
Координаты трансмембранных участков, предсказанные моделью.
(1, '250-271'), (2, '317-330'), (3, '361-374'), (4, '385-400'), (5, '407-422')
Ни один из трансмембранных участков не совпал полностью, хотя они перекрываются почти по всем координатам. Количество трансмембранных участков не отличается. Предсказание получилось значительно лучше, чем при сравнении комплекса из 1-го задания, вероятно, дело в том, что структура нашего белка с альфа-спиралью проще.
Задание 3. База данных TCDB
Белки, исследуемые в практикуме, на нашлись в бд TCDB, поэтому я взял используемый в качестве примера для описания задания – P0A910. Поиск был осуществлен по AC.
Система TC
V (число) соответствует классу переносчика; W (буква) соответствует подклассу переносчика, который, в случае основных активных переносчиков, относится к источнику энергии, используемому для приведения в движение механизма транспорта; X (число) соответствует семейству переносчиков (иногда даже надсемейству); Y (число) соответствует подсемейству, в котором обнаружен переносчик, а Z (число) соответствует субстрату или диапазону транспортируемых субстратов.
TC находки: 1.B.6.1.1
1 – Channels/Pores
B – β-Barrel Porins
6 – The OmpA-OmpF Porin (OOP) Family
Имя белка: Внешний мембранный белок A OmpA
Длина: 346
Молекулярная масса: 37201.00
Вид: Escherichia coli [83333]
Число трансмембранных участков: 1
Местонахождение1 / Топология2 / Ориентация3: Cell outer membrane1 / Multi-pass membrane protein2
Субстрат: anion, resveratrol, (S)-naringenin, rutin
Порин OmpA слабо селективен к анионам. Может существовать в двух различных состояниях проводимости. Может участвовать в транспортировке фенилпропаноидов (ресвератрола, нарингенина и рутина). В ходе исследований фолдинга с использованием бислоев диолеоилфосфатидилхолина были обнаружены три мембраносвязанных промежуточных продукта OmpA. Был описан механизм образования вторичной и третичной структур, применимый к этому и другим мембранным белкам с β-бочками.


