Мне нужно было собрать геном бактерии Buchnera aphidicola из коротких чтений Illumina. Для начала я скачала эти самые чтения из проекта по секвенированию SRR4240381 и распаковала их в файл SRR4240381.fastq.
1. Подготовка чтений программой trimmomatic.
Чтобы удалить все адаптеры, я создала файл с ними:
echo /P/y15/term3/block4/adapters/NexteraPE-PE.fa >> list.txt echo /P/y15/term3/block4/adapters/TruSeq2-PE.fa >> list.txt echo /P/y15/term3/block4/adapters/TruSeq2-SE.fa >> list.txt echo /P/y15/term3/block4/adapters/TruSeq3-SE.fa >> list.txt echo /P/y15/term3/block4/adapters/TruSeq3-PE.fa >> list.txt echo /P/y15/term3/block4/adapters/TruSeq3-PE-2.fa >> list.txt seqret @list.txt ad.fastaПотом я проверила качество ридов:
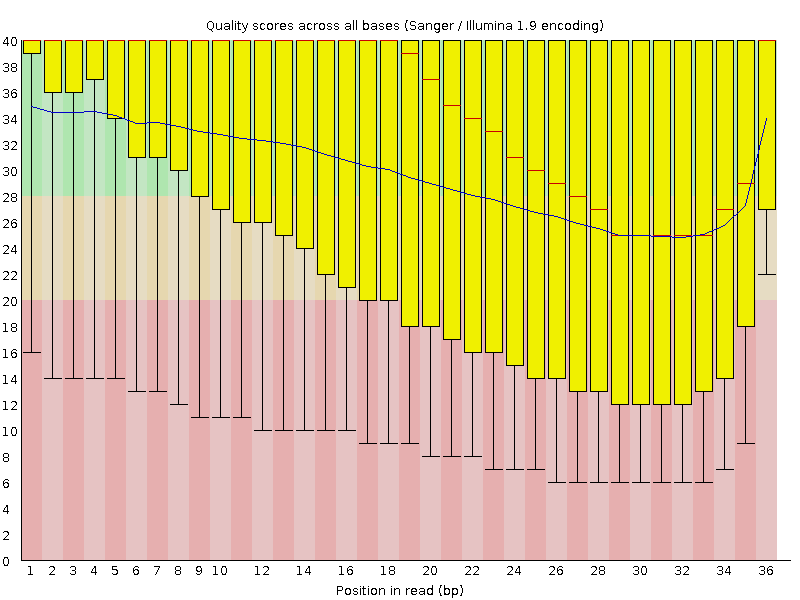
fastqc SRR4240381.fastqЗатем я удалила адаптеры и очистила риды:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240381.fastq clear.fastq ILLUMINACLIP:ad.fasta:2:7:7 TRAILING:20 MINLEN:30И проверила качество оставшихся ридов:
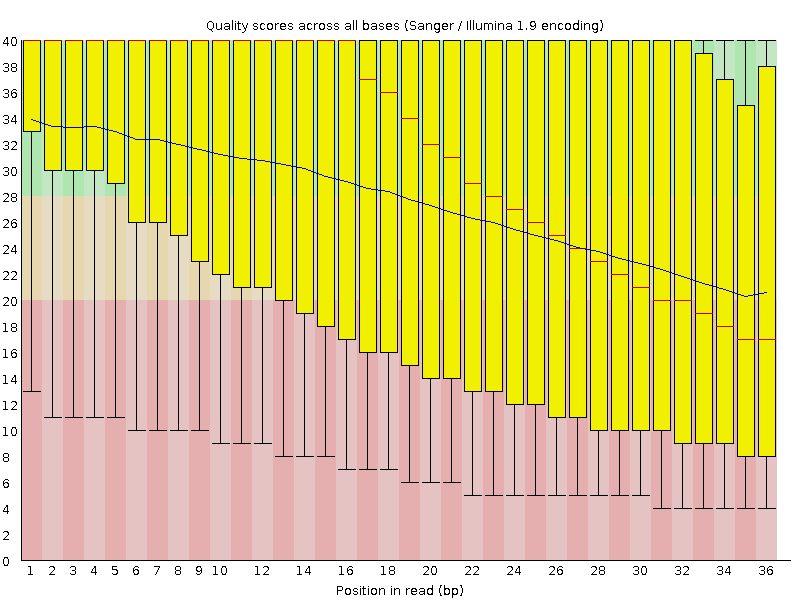
fastqc clear.fastqВот сравнение качества до и после чистки:
| До чистки | После чистки |
| SRR4240381_fastqc.html | clear_fastqc.html |
| 13710994 ридов | 11983833 ридов (87.4% от исходного количества) |
 |  |
2. Подготовка k-меров.
Я подготовила k-меры с помощью программы velveth. Длина к-мера 29. Команда:
velveth velveth29 29 -short -fastq clear.fastqПолучилась папка с k-мерами - velveth29.
3. Сборка генома.
Сборка производилась программой velvetg. Команда:
velvetg velveth29В полученной нами на предыдущем этапе папке velveth29 появилось 2 новых файла: contigs.fa (файл с контигами) и stats.txt (со статистикой по контигам). Вот что вышло (все вычисления производились с помощью Excel):
| Общая длина генома | Кол-во контигов | N50 | L50 |
| 1702250 | 9676 | 262 | 438 |
| ID | Длина | Покрытие | Последовательность |
| 9 | 50157 | 55.188927 | 9.fasta |
| 3 | 49915 | 50.676129 | 3.fasta |
| 6 | 47295 | 46.576298 | 6.fasta |
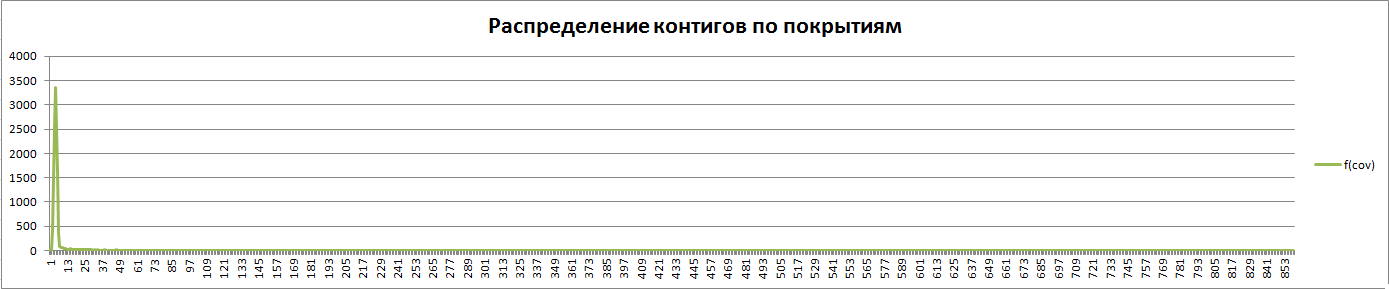
Рис.1. Распределение контигов по покрытиям. По горизонтали
покрытие, по вертикали кол-во контигов. Сюда не входит контиг с покрытием 74176.
4. Анализ.
Для трех самых длинных контигов был запущен megablast с хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253). Вот что получилось:
| ID | Max score | Total score | Query cover | E-value | Ident | Number of alignments | Coordinates in genome |
| 9 | 5760 | 23126 | 75% | 0.0 | 78% | 8 | 614190:620926, 621055:627104, 2004:11103, 13994:14465, 14727:17919, 17962:20171, 20358:22183, 23067:28363, 30028:32745 |
| 3 | 5421 | 21303 | 79% | 0.0 | 75% | 5 | 127825:140555, 144368:151796, 153752:161738, 161898:166701, 166750:173180 |
| 6 | 4050 | 14337 | 60% | 0.0 | 77% | 8 | 495107:495033, 494864:493487, 488106:48199, 481545:480874, 480660:474844, 474667:467412, 467421:4624967, 454069:451729 |
5. Другая сборка.
Я сделала сборку как в пунктах 2-3, но только с длиной к-мера 25:
velveth velveth25 25 -short -fastq clear.fastq velvetg velveth25Результаты:
| Общая длина генома | Кол-во контигов | N50 | L50 |
| 2468190 | 13507 | 615 | 857 |
| ID | Длина | Покрытие | Последовательность |
| 314 | 22177 | 44.406548 | 314.fasta |
| 97 | 14010 | 76.729408 | 97.fasta |
| 53 | 11450 | 88.82 | 53.fasta |