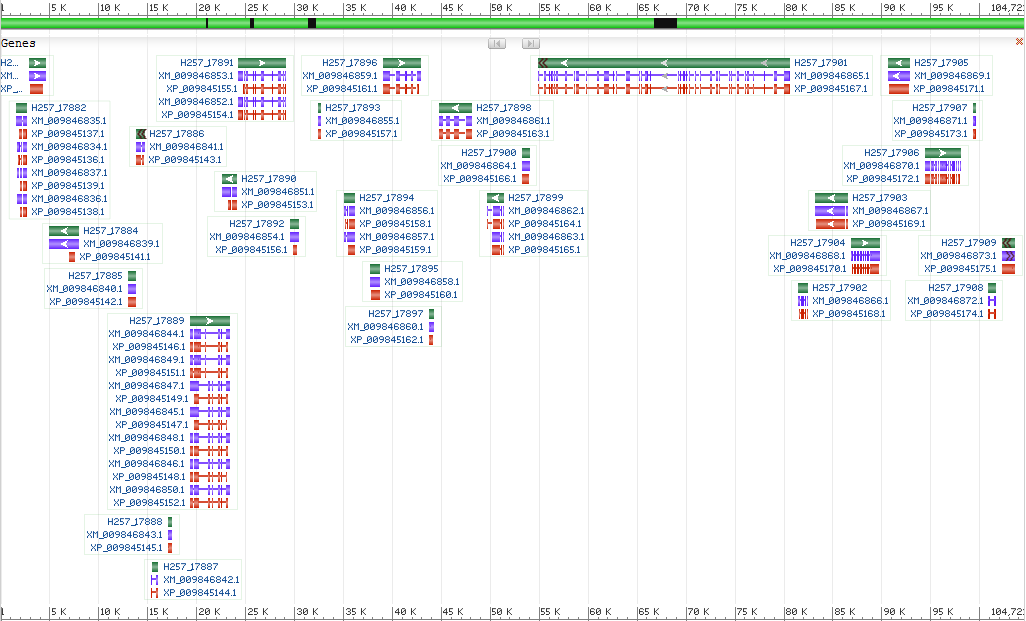
Описание выданного контига и одного из генов на нем, для которого предсказан альтернативный сплайсинг
Мне достался контиг NW_009798389, который на самом деле не контиг, а скэффолд (рис.1). Этот скэффолд принадлежит геному организма Aphanomyces astaci (strain APO3), который относится к классу Oomycetes, порядку Saprolegniales и является возбудителем так называемой чумы раков (рис.2). Размер скэффолда - 104721 bp. Кол-во генов - 28, кол-во CDS - 40. Надо сказать, что все CDS кодируют гипотетические белки, кроме двух (XM_009846864.1 и XP_009845170.1), одна из которых кодирует эукариотический фактор 6 инициации трансляции, а другая - TKL протеин киназу, причем эти CDS единственные в своих генах. Так как это скэффолд, в нем присутствуют нечитаемые участки - на рис.1 они показаны черным цветом сверху, а в последовательности они записаны буквами N. Также в скэффолде присутствуют гены или мРНК, обозначенные на рисунке несколькими темными стрелками, а в аннотации их координаты записаны с использованием знаков < и > . Это значит, что неизвестны точные границы гена.
Получен файл с последовательностью скэффолда в формате fasta - scaffold.fasta.
|
|
 Рис.2 Хвост рака, зараженного Aphanomyces astaci.
|
Можно заметить, что у некоторых генов существует 2 или более вариантов сплайсинга мРНК. Например, 2 варианта есть у гена H257_17891 (рис.3). В нем существует одновременно 2 мРНК, и, соответственно, может читатся 2 варианта белка: XP_009845155.1 и XP_009845154.1. В обоих белках найдено 5 доменов: TPR, TPR repeat, TPR_1, TPR_11 и TadD. Однако в варианте XP_009845154, который длиннее другого, обнаружился еще один домен ANAPC8 (все домены обозначены на рис.3 черным цветом).

Рис.3 Изображение гена H257_17891. Зеленым обозначен ген, фиолетовым мРНК, красным CDS,
черным домены.
Предсказание генов и белок-кодирующих областей в выданном скэффолде
С помощью программы AUGUSTUS я попыталась предсказать гены в моем скэффолде. Параметры запуска программы ниже.
Prediction job ID: predC92BqDYP AUGUSTUS parameter project identifier: tetrahymena Genome file: scaffold.fasta User set UTR prediction: true Server set UTR prediction: false [UTR parameters missing or conflict with allowed gene structure!] Report genes on: both strands Alternative transcripts: none Allowed gene structure: predict any number of (possibly partial) genes Ignore conflictes with other strand: false
Мой скэффолд принадлежит организму из царства Stramenopiles, представителей которого не было в списке организмов для параметров модели. Зато там были представители царства Alveolata, которые, как и Stramenopiles, входят в состав более крупного таксона Chromalveolata. Поэтому для параметров модели был взят представитель альвеолят Tetrahymena thermophila.
Полученный файл predictions.tar.gz был распакован командой
tar -xzvf *.tar.gz, и получились следующие файлы:
| augustus.aa | Последовательности трансляций предсказанных генов в формате fasta |
| augustus.cdexons | Предсказанные экзоны в формате fasta |
| augustus.codingseq | Предсказанные CDS в формате fasta |
| augustus.gbrowse | Координаты всех найденных особенностей в формате gtf |
| augustus.gff | Координаты всех найденных особенностей в формате gff |
| augustus.gtf | Координаты всех найденных особенностей в формате gtf |
Всего AUGUSTUS предсказал 66 генов. Но ни один из них не совпадал с аннотированными полностью. Только один (20819889) совпал по 5' концу. И это был не тот ген, который описывала я. Вдобавок, ему присуще такое явление как partial start, то есть границы гена не известны точно и определяются исходя из его мРНК или даже CDS (как и было в данном случае). Что касается CDS, то всего их нашлось 156, из них 14 совпадают с аннотированными по обоим концам, 11 только по 3' и 27 только по 5'. Тут стоит заметить, что CDS в данном случае (случае gtf файла) это не последовательность, с которой читается один белок, а кусок такой последовательности, не разделенный интронами в исходной последовательности, то есть одна запись в gtf файле, помеченная как CDS. Неизвестно, почему все предсказалось так плохо. Хотя этого следовало ожидать, ведь эукариотические гены очень сложно устроены.