Сравнение предсказаний генов в базе данных GenBank и по данным Prodigal для плазмиды
Мне была дана плазмида CP011023. Это плазмида pSHB9, принадлежащая почвенной бактерии Bacillus pumilus штамма SH-B9.
В ней 91229 н.о.; 104 гена, из них 92 кодирующих.
Я получила файл моей плазмиды в формате gff с особенностями с помощью команды:
seqret embl:CP011023 gff:plasmid.gff -featureПолученный файл: plasmid.gff
Для того чтобы извлечь информацию о генах плазмиды, я скачала последовательность fasta:
seqret embl:CP011023 fasta:plasmid.fastaПолученный файл: plasmid.fasta
С помощью Prodigal я извлекла информацию о генах:
prodigal -i plasmid.fasta -o plasmid.sco -f scoПолученный файл: plasmid.sco
Для того чтобы посчитать число и процент совпадений генов, аннотированных Genbank, с генами, найденными Prodigal, я создала следующий скрипт: script.py. На вход скрипту подаются названия файлов: из Genbank в формате gff и полученный с помощью Prodigal в формате sco. Скрипт создает 6 списков, по три на каждый файл - начало гена, конец гена и цепь. Потом он их сравнивает и выводит следующую информацию: число генов, аннотированных Genbank; число и процент генов Genbank, совпадающих с генами, предсказанными Prodigal по обоим концам; только по C-концу; только по N-концу; не совпадающих по обоим концам. Я запустила скрипт так:
python script.py plasmid.gff plasmid.sco
Выдачa:Whole number of genes by Genbank - 92 Number of genes matching by both ends - 75 Percent of genes matching by both ends - 81.52% Number of genes matching only by C-end - 14 Percent of genes matching only by C-end - 15.22% Number of genes matching only by N-end - 0 Percent of genes matching only by N-end - 0.0% Number of genes not matching - 3 Percent of genes not matching - 3.26% |
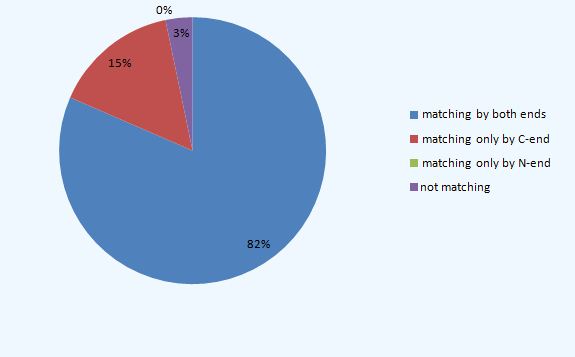 |
|
К тому же, в этой плазмиде
используется нуклеотидный код 11 (The Bacterial, Archaeal and Plant Plastid Code), особенностью которого является то,
что старт-кодонов в нем несколько: TTG, CTG, ATT, ATC, ATA, GTG (и ATG, который является старт-кодоном у всех организмов).
На самом деле эти кодоны кодируют алифатические аминокислоты, но в некоторых случаях они становятся старт-кодонами и
кодируют метионин. Prodigal учел этот момент, но это еще больше осложнило поиск генов и привело к большему количеству
ошибок. Что касается отсутствия несовпадений только по C-концу, то это легко объяснимо, так как стоп-кодоны не имеют
других функций кроме остановки трансляции, поэтому разночтений здесь быть не может.
|
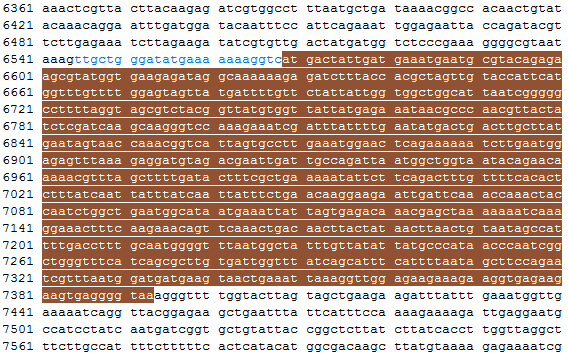 Рис.2 Участок, совпадающий только по С-концу. Синим шрифтом выделена область, содержащаяся в аннотированном гене, но не включенная в него Prodigal. |
Сравнение предсказаний генов в базе данных GenBank и по данным Prodigal для геномной записи
В первом семестре я работала с организмом Bacillus pumilus SAFR-032. Вот ссылка на его хромосому в Genbank: CP000813. С помощью команд (см. задание 1) я получила аннотации его генов bacpu.gff и их предсказания в Prodigal bacpu.sco. То же самое я получила и для Escherichia coli str. K-12 substr. MG165 (U00096.3) - ecoli.gff и ecoli.sco (fasta файлы я не стала сюда помещать). После этого к обоим геномам был применен вышеуказанный скрипт. Результаты:
| Bacillus pumilus | Escherichia coli |
Whole number of genes by Genbank - 3715 Number of genes matching by both ends - 3275 Percent of genes matching by both ends - 88.16% Number of genes matching only by C-end - 312 Percent of genes matching only by C-end - 8.4% Number of genes matching only by N-end - 1 Percent of genes matching only by N-end - 0.03% Number of genes not matching - 127 Percent of genes not matching - 3.42% | Whole number of genes by Genbank - 4386 Number of genes matching by both ends - 3831 Percent of genes matching by both ends - 87.35% Number of genes matching only by C-end - 318 Percent of genes matching only by C-end - 7.25% Number of genes matching only by N-end - 75 Percent of genes matching only by N-end - 1.71% Number of genes not matching - 162 Percent of genes not matching - 3.69% |
Видно, что данные для двух бактерий почти совсем не отличаются (в процентах). Единственное, что обращает на себя внимание - большое количество несовпадений по С-концам у ECOLI - 75, тогда как у BACPU только 1 (притом что их геномы довольно близки по размерам, во всяком случае, одного порядка). Как я уже объясняла в первом задании, несовпадение С-концов - явление маловероятное. Было замечено, что гены с несовпадением С-концов в ECOLI часто оказываются псевдогенами, которые представляют собой гены, утратившие функции. В данном случае, если эти псевдогены образовались в результате сдвига рамки считывания или точечной мутации, вполне возможно, что стоп-кодон исчез, и из-за этого Prodigal не смог найти С-конец гена. Единственное, что вызывает удивление - это практическoe отсутствие таких генов у BACPU. Возможно, это объясняется тем, что ECOLI обитает в агрессивной мутагенной среде в отличие от BACPU, но это не точно.
