1.Чтение последовательности ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру.
Мне дано два файла в формате .ab1, соответствующие прочтению прямой и обратной цепочки секвенируемой ДНК:
Для обратной цепи была получена комлементарная последовательность с помощью "reverse complement". В последовательностях были найденны и удалены нечитаемые участки: в прямой цепи 1-20 и 675-718, а в обратной 1-21 и 673-720 (если выровнять ее с прямой, то участки (-37)-(-18) и 636-683 по нумерации прямой цепи). Странно то, что длины начальных нечитаемых участков в цепях примерно одинаковы, как и длины конечных, хотя все должно было быть наоборот, ведь из-за применения "reverse complement" начало чтения обратной цепи оказалось в конце. На рис. 1 (а-г) изображены нечитаемые концы обоих последовательностей (эти и другие рисунки получены с помощью Paint).
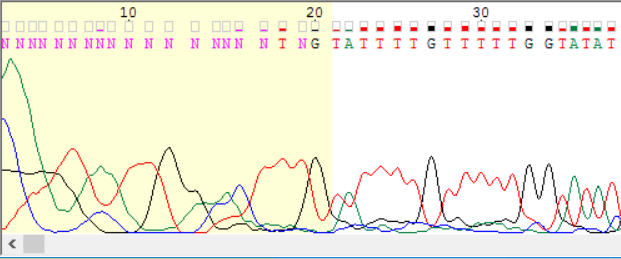 Рис.1а. 5'-конец прямой последовательности Рис.1а. 5'-конец прямой последовательности |
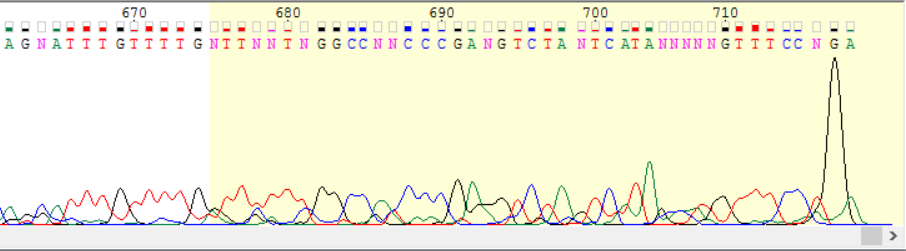 Рис.1б. 3'-конец прямой последовательности Рис.1б. 3'-конец прямой последовательности |
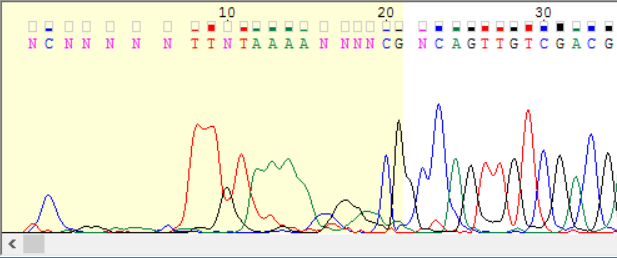 Рис.1в. 5'-конец обратной последовательности Рис.1в. 5'-конец обратной последовательности |
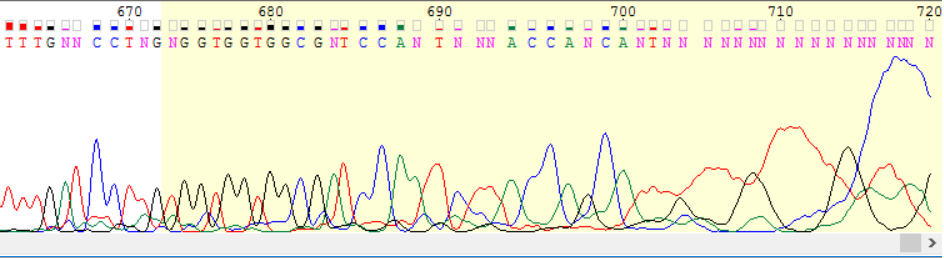 Рис.1г. 3'-конец обратной последовательности Рис.1г. 3'-конец обратной последовательности |
В начале чтения цепи уровень шума низкий, но пики сливаются друг с другом, поэтому хроматограмма не читается, а в конце цепи увеличивается уровень шума. По всей цепи уровень шума составляет около 15% от сигнала.
Далее были отредактированы сложные участки прямой и обратной цепей (рис.2 а-д). На рис. 2а и 2в показаны 2 пика, находящихся на слишком маленьком расстоянии друг от друга, так, что непонятно, то ли это 2 нуклеотида, то ли полиморфизм, то ли шум. На рис. 2д между соседними пиками слишком большое расстояние, на рис. 2б и 2в высокий уровень шума. В этих случаях истину легко установить по комплементарной цепи. Но в одном случае (рис. 2г) это невозможно, так как на прямой цепи хроматограмма не читается (это самое начало цепи). В этом случае проблемный нуклеотид был обозначен как полиморфизм, потому что адениновый и гуаниновый пики почти одинаковы.
 Рис. 2а Рис. 2а |
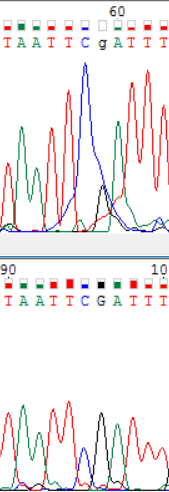 Рис. 2б Рис. 2б |
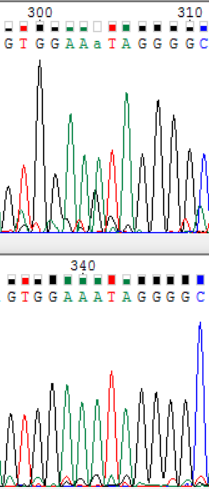 Рис. 2в Рис. 2в |
 Рис. 2г Рис. 2г |
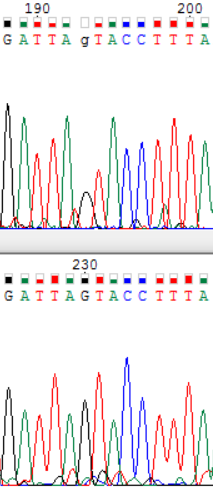 Рис. 2д Рис. 2д |
Обе отредактированные последовательности были сохранены в fasta формате ( прямая цепь, обратная цепь (reverse comlement)) и выровнены вручную в программе Jalview (проект).
2.Нечитаемые хроматограммы.
Я выбрала 2 файла из папки bad (прямая цепь, обратная цепь) и попыталась восстановить последовательность для этих файлов. Нельзя сказать, что это совсем невозможно, однако точность подобного секвенирования сомнительна. В обоих этих файлах нечитаемые области занимают около 60% всей хроматограммы (рис.3 а-б).
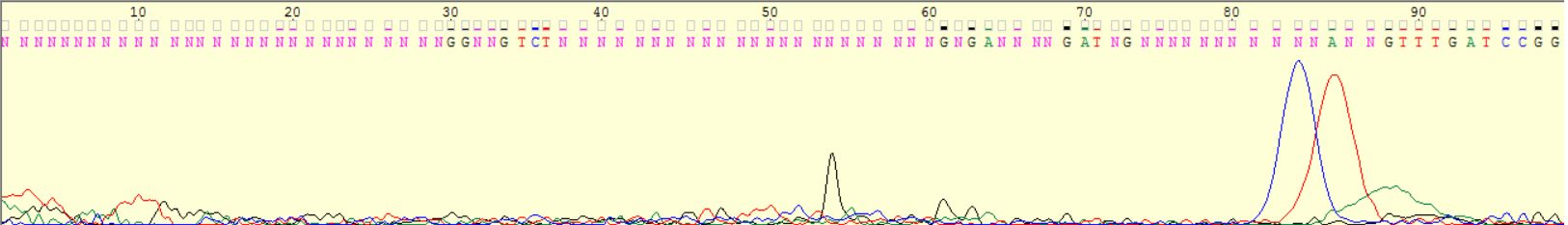 Рис.3а. фрагмент нечитаемой области в начале прямой последовательности
Рис.3а. фрагмент нечитаемой области в начале прямой последовательности
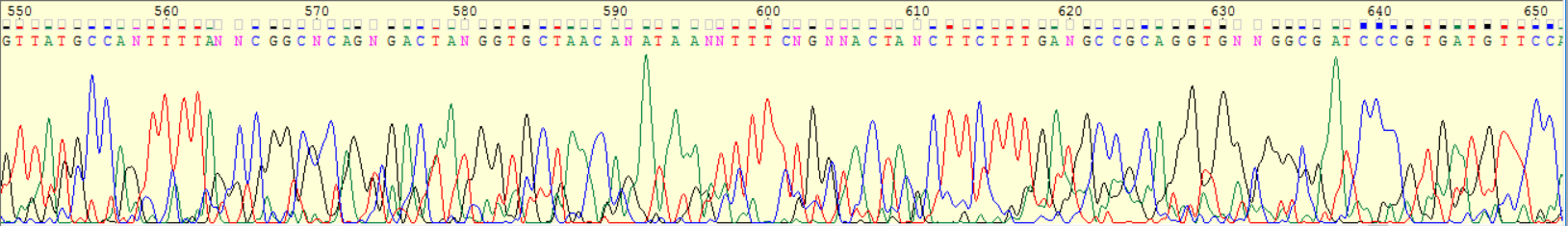 Рис.1б. Фрагмент нечитаемой области в конце прямой последовательности
Рис.1б. Фрагмент нечитаемой области в конце прямой последовательности
Вдобавок шум на хроматограмме составляет до 50% сигнала. К тому же встречаются полиморфизмы. На рис.4 изображен фрагмент прямой и комплементарной цепи с моими попытками восстановления. Можно увидеть полиморфизмы и шум.
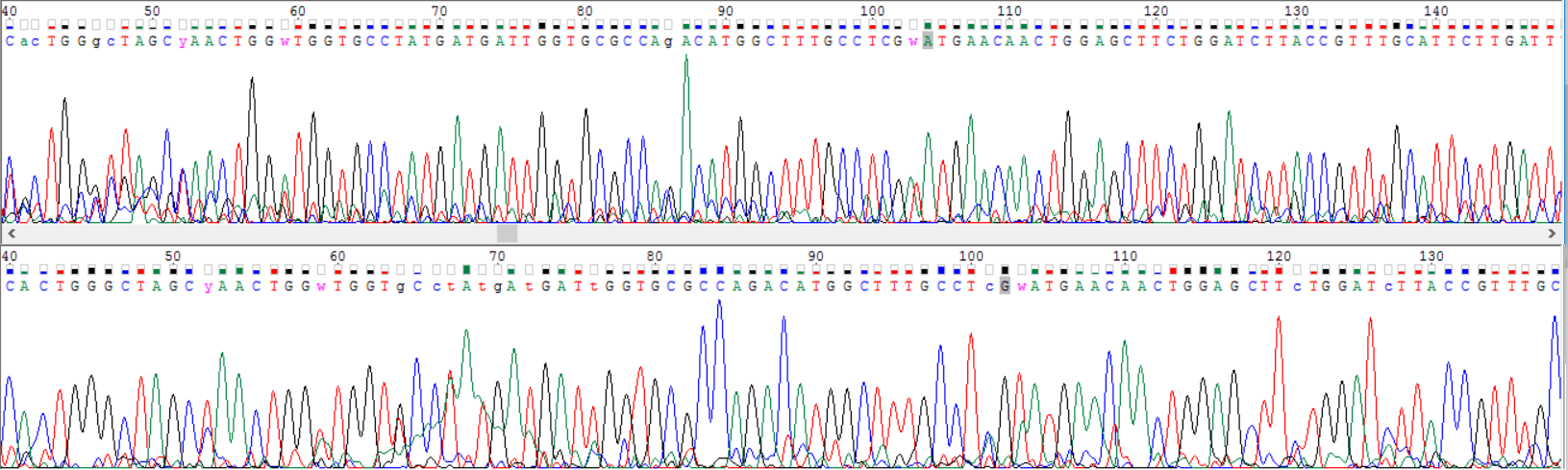 Рис.4
Рис.4
Тем не менее я смогла восстановить часть последовательности прямой цепи (читаемую область).