Поиск мотива связывания.
Для бактерии Mycobacterium leprae TN ищем сайт связывания транскрипционного фактора LexA_Myco c помощью программы MEME.
Параметры MEME:
- Длина мотива: 8-20 п.н. (сайты связывания представляют собой короткие сегменты ДНК, длиной от 8—10 до 16—20 пар оснований)
- 0 или 1 мотив на последовательность
- Мотив может располагаться на любой цепи ДНК
- Ищем вначале 3 наилучших мотива для сравнения E-value
В исходном файле было много дупликаций, пришлось оставить из дупликаций только последовательности с наилучшим скором (новый файл).
Результат работы программы - meme.txt. Программа выдала только 1 мотив, т.к. не могла найти больше стартовых точек для EM.
E-value = 3.4e-408
 Рис.1. Лого мотива. |
PWM. letter-probability matrix: alength= 4 w= 16 nsites= 67 E= 3.4e-408 0.000000 0.000000 0.000000 1.000000 0.029851 0.955224 0.014925 0.000000 0.014925 0.000000 0.985075 0.000000 1.000000 0.000000 0.000000 0.000000 1.000000 0.000000 0.000000 0.000000 0.104478 0.865672 0.000000 0.029851 0.522388 0.014925 0.358209 0.104478 0.089552 0.373134 0.253731 0.283582 0.671642 0.074627 0.134328 0.119403 0.000000 0.000000 0.014925 0.985075 0.000000 0.000000 1.000000 0.000000 0.000000 0.000000 0.000000 1.000000 0.000000 0.000000 0.149254 0.850746 0.000000 0.865672 0.000000 0.134328 0.000000 0.000000 0.925373 0.074627 0.776119 0.074627 0.119403 0.029851 |
Затем мотив TCGAACRBATGTTCGA, найденный MEME, был подан на вход сервису tomtom, чтобы найти похожий мотив (т.е. мотив с похожей PWM), предсказанный для этого ТФ в БД RegTransBase. tomtom нашел 19 похожих мотивов. Информация о лучшей находке представлена в таблице 1. PWM представлен в таблице 2 . Для того, чтобы получить не только html-выдачу tomtom, а еще текстовую и т.д., нужно снять галочку быстрого поиска. Результат представлен в xml-файле.
Таблица 1.Информация о лучшей находке
| Название мотива | E-value | Название транскрипционного фактора | Ориентация |
| PsrA_Proteobacteria | 7.65e-03 | Transcriptional regulator PsrA | Комплементарная |
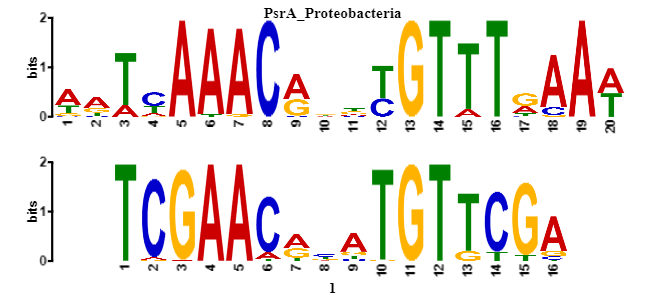 Рис.2. Logo двух мотивов | PWM лучшей находки
0.486639 0.00057289 0.00057289 0.512215 0.000705882 0.00057289 0.00057289 0.998148 0.000705882 0.0261483 0.12845 0.844696 0.128583 0.537658 0.0261483 0.307611 0.998148 0.00057289 0.00057289 0.000705882 0.895847 0.00057289 0.00057289 0.103008 0.998148 0.00057289 0.00057289 0.000705882 0.000705882 0.998015 0.00057289 0.000705882 0.640092 0.00057289 0.358629 0.000705882 0.384337 0.358629 0.12845 0.128583 0.230885 0.358629 0.230752 0.179734 0.000705882 0.384204 0.0261483 0.588942 0.000705882 0.00057289 0.998015 0.000705882 0.000705882 0.0261483 0.00057289 0.972573 0.0262813 0.00057289 0.00057289 0.972573 0.000705882 0.00057289 0.00057289 0.998148 0.282036 0.0261483 0.563233 0.128583 0.81912 0.00057289 0.00057289 0.179734 0.154158 0.230752 0.0517237 0.563366 0.282036 0.102875 0.0261483 0.588942 |
|
FIMO Затем с помощью программы FIMO был найден мотив в геноме бактерии. Для того, чтобы искать в upstream region (ищем там, т.к. большинство ТФ связываются до последовательности гена), была задан параметр Upstream Sequences: Prokaryotic. p-value вначале было ограничено значением 0.001, но при этом программа нашла очень много мотивов, так что ограничили значение p-value цифрой 1E-4. В качестве организма был выбран заданный организм Mycobacterium leprae TN. Результат был сохранен в файле fimo1.txt. Всего 335 находок. Ссылка на страницу генома из NCBI, также геном из NCBI представлен в данной таблице ncbi Были выбраны 7 лучших находок с более-менее приемлемым q-value. Информация по ним представлена в таблице 3. Координаты генов были определены при помощи соответствующей записи NCBI с помощью поиска в таблице ncbi. На всякий случай была скачан файл формата .gbff, и к нему была применена команда featcopy. Результат тот же - out1.gff. В базе данных STRING нашлись все три гена лучших находок: lexA, recA и ML1004 (строго говоря, ML1004 - это locus tag, названия гена для него нет). Геномное окружение для них представлено на рисунке 3.Таблица 3.Параметры находок FIMO
lexA кодирует белок SOS-системы репарации ДНК (репрессируют гены SOS-системы в нормальных условиях). В присутствии одноцепочечной ДНК белок RecA взаимодействует с репрессором LexA, вызывая его автокаталитическое расщепление, которое разрушает ДНК-связывающую часть репрессора и инактивирует его. После этого гены SOS-регулона активируются, происходит репарация ДНК. Является гидролазой [1],[2], [4] lexA и recA принадлежат одному и тому же регулону LexA. [3] Может катализировать гидролиз АТФ в присутствии одноцепочечной ДНК, зависимое от АТФ поглощение одноцепочечной ДНК дуплексной ДНК и АТФ-зависимую гибридизацию гомологичных одноцепочечных ДНК. Он взаимодействует с LexA, вызывая его активацию и приводя к его автокаталитическому расщеплению. Так как для ML1004 известен только locus_tag, мы не смогли найти исчерпывающей информации по нему. |
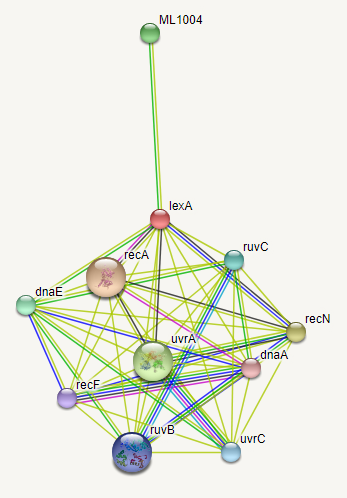
Рис.3. Геномное окружение |
fuzznuc
Используя fuzznuc из пакета EMBOSS,нужно было найти все сайты, пересекающиеся с предсказанными мною сайтами связывания ТФ, и похожие на сайты связывания метилтрансфераз. Для выполнения данного задания я запускала поочередно: script.py (для его работы нужны файлы ncbi.txt с информацией о генах и fimo1.txt с выдачей fimo) , команду seqret @list.txt out.fasta, и затем скрипт script1.py. В результате получился файл motif.fasta, содержащий мотивы из FIMO и их окрестности в 50 нуклеотидов. Этот файл подаем на вход команде fuzznuc -sequence motif.fasta -pattern @MT.pat -outfile out.txt (MT.pat - файл с сайтами связывания метилтрансфераз) и получаем файл out.txt, содержащий информацию о пересечениях мотивов и сайтов связывания метилтрансфераз. На самом деле программа выдала не пересечения, а сайты связывания, расположенные не более чем за 50 нуклеотидов от нашего мотива (в том числе пересекающие его). Это необходимо для того чтобы не потерять пересечения с длинными сайтами, которые, даже пересекаясь с мотивом, выходят за его границы.
REBASE
В базе данных REBASE был найден геном нашей бактерии, а также закодированные в нем метилтрансферазы. Результат можно посмотреть по ссылке. Всего было найдено три типа метилтрансфераз, для одного из них была указана специфичность: AGAAGATTGGGGTGATGATGTTCGGGTCTCCCGAAAC (pattern833 в fuzznuc). Данный сайт не был найден в участках пересечения мотивов и метилтрансфераз, полученных в выдаче fuzznuc, поэтому, скорее всего, метилирование не влияет на наличие сайтов связывания транскрипционных факторов.
Ссылки
[1] -microbesonline[2] - БД KEGG Enzyme
[3] - Regprecise
[4] - Uniprot