Чтение последовательностей по Сэнгеру
Справочная информация
В ходе работы использовались программы Chromas и Jalview.
Для удобства сразу прилагаю все файлы для скачивания в одном месте:
Исходные файлы (хромотограммы) я буду называть просто - 1 и 2
Фаста файл с результатом хромотограммы 1
Фаста файл с результатом хромотограммы 2
Качество хромотограммы
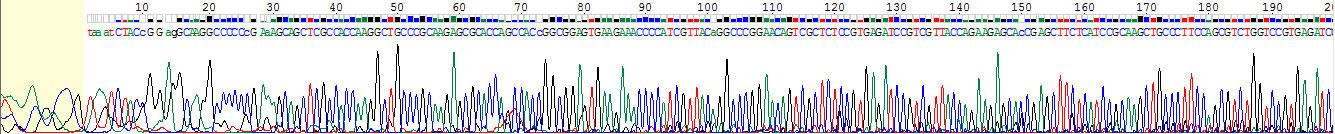
Качество хромотограммы изменяется от начала к концу: сначала сигнал слабее и он больше размыт, потом сигнал становится более разборчивым и сильным. Шум при этом изменяется не сильно.
Границы читаемости хромотограмм
Я установил следующие границы, подлежащие чтению:
1) [1-14]U[380-382]
2) [379-380]
При выборе границ я опирался на комплементарную хромотограмму, что позволило убедиться в том, что часть хромотограммы в начале и конце, которая имеет сама по себе очень плохое качество, может быть уточнена комплементарной цепью, в которой этот участок попадает в "хорошую" область.
Проблемные нуклеотиды
В первой хромотограмме обнаружилось довольно много проблемных нуклеотидов, в отличие от второй.

Внимание, нумерация используется новая, вырезанные участки я не считаю!
В первой хромотограмме:
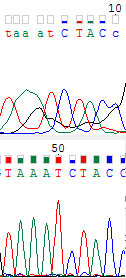
Былы заменены первые пять нуклеотидов. Хотя на самой хромотограмме сложно разобрать где шу, а где сигнал, но на комплементарной можно видеть пять ровных пиков.
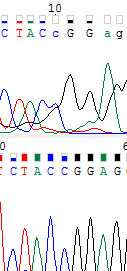
Аналогично, уточнен десятый. Тут можно было пойти и от одной хромотограммы - видно, что зеленый пик, соответствующий A лежит на оптимальной дистанции от соседних, тогда как черный пик (G) и слабее, и явно выбивается из схемы расположения пиков.
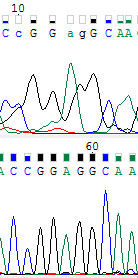
А так же тринадцатый и четырнадцатый. Так как качество хромотограммы еще не успело сильно возрасти, часто приходится прибегать к помощи второй хромотограммы, у которой этот участок расположен в оптимальном диапазоне качества.
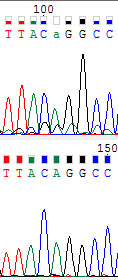
101 нуклеотид. Пик приближается к ровню шума, но по комплементарной хромотограмме видно, что это все же пик.
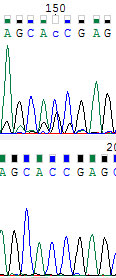
150 нуклеотид. Ситуация похожа на 101-ый, но тут уже поднимается сам уровень шума.
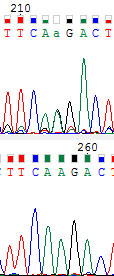
213-ый. Интересная ситуация - если бы не комплементарная хромотограмма, то я бы отнес это к полиморфизму, т.к. сигналы лежат в одном месте, и имеют одинаковую силу.
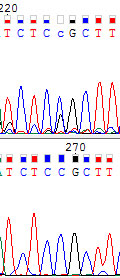
224-ый. Уточнен цитозин.
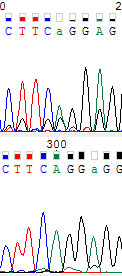
255-ый. Уровень сигнала упал к уровню шума, хотя все остается читаемо.
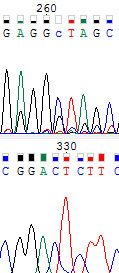
261-ый. У комплементарной хромотограммы это участок начала, на котором качество понижается. Однако все еще можно уточнить, что здесь точно не гуанин.
Во второй хромотограмме:
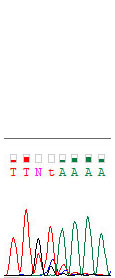
3-ий нуклеотид, если считать от начала комплементарной хромотограммы (с конца, если говорить о 2-ой). Хотя тут и нет комплементарной цепи, но я взял на себя смелость назвать все остальные пики шумом.
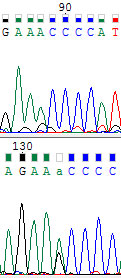
133-ий. Если бы пик гуанина на первой не был бы размытым, то я бы сказал, что тут полиморфизм. Однако я в смятении.
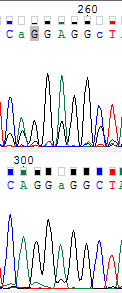
303-ий. Тут, вроде, все понятно.
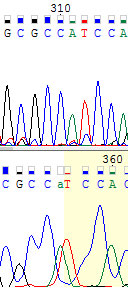
356-ой. Приближается самое начало хромотограммы, а значит и очень низкое качество, так что уточняем по комплементарной.
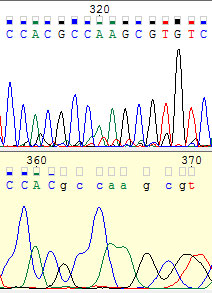
С 362-ой по 370. Так же, как и в предыдущем пункте.
Полиморфные нуклеотиды
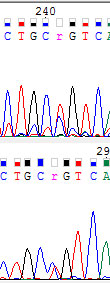
241-ый на 1 или 286-ой на 2. Единственный нуклеотид, чья полиморфность прослеживается на обоих хромотограммах (хотя, возможно, что 133-ий по 2 может к нему присоединиться).
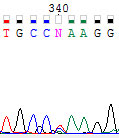
340-ой на 2. Тут может быть все что угодно.
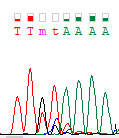
3-ий на 2. Либо T, либо G. Без комплементарной цепи не понятно, шум ли это, или полиморфизм. Однако, все-же отнесу его к последнему.
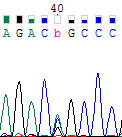
40-ой на 2. Та же история. Возможны А, C и G.
Пример нечитаемой хроматограммы
Что-бы внести ясность в понятие качества, приведу пример совершенно не читаемой хромотограммы.
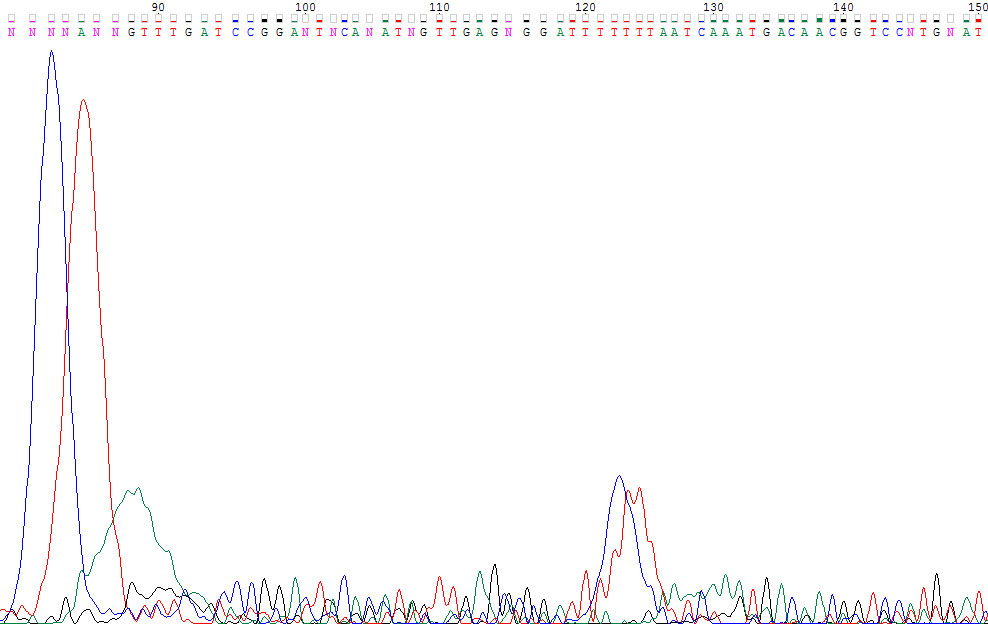
Видно, что нельзя сказать где сигнал, а где шум, несут ли пики сигнал от двух нуклеотидов, или от одного и т.д и т.п.
Так что я бы оценил качество своей хромотограммы как хорошее.
| на главную |
© Гавриш Глеб 2016 |