EMBOSS и выравнивания геномов
Упражнения по EMBOSS
Я буду использовать последовательности из своей курсовой. Вы можете скачать все файлы по этой ссылке
1. Объединение нескольких файлов fasta в один
Команда: seqret "kti?.fasta" ktis.fasta
При этом в директории лежат файлы kti1.fasta,kti2.fasta и т.д.
2. Разделение одного файла на несколько
Команда: seqretsplit kti12.fasta
Команда разделяет файл на несколько, создавая индивидуальный файл для каждой последовательности, которую найдет. При этом он дает им названия, исходя из их ID.
3. Вырезание последовательностей по координатам (от и до, а так же ориентация)
Команда: seqret seqret -sask kti1.fasta
После этой команды, нужно будет ответить на все нужные вопросы.
5. Трансляция данной нуклеотидной последовательности в 6 рамках.
Команда: transeq -frame 6 kti1.fasta kti1_prots_in_6_frames.fasta
Транслирует последовательность по всем 6 рамкам считывания.
10. Перемешать буквы в последовательности.
Команда: shuffle kti2.fasta > kti2_shuffle.fasta
Команда перемешивает нуклеотиды в файле kti2.fasta, при этом стандартно stdout отправляется на вывод в консоль.
Выравнивание геномов и построение карты локального сходства
Для начала я решил посмотреть род своей бактерии из первого семестра - Methylomicrobium,- однако геномы либо слишком разные, либо идентичные. Так что рассматривать я решил Pseudomonas stutzeri A1501 и Pseudomonas stutzeri strain SLG510A3-8. Искал по таблице организмов с известными геномами до хромосом, запуская megablast.
ID: NC_009434.1 и NZ_CP011854.1
Карта локального сходства получалась через blast для двух нуклеотидных последовательностей.
Query cover: 86%; E-value: 0.0; Ident: 98%; Search Strategies
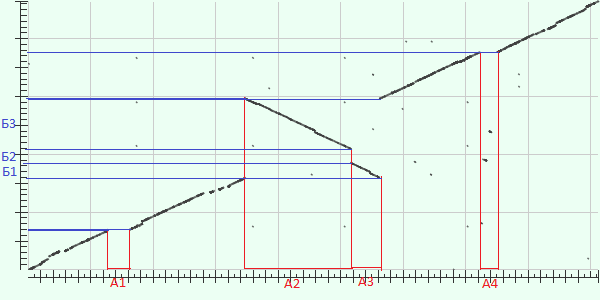
По оси X-NC_009434.1, по оси Y-NZ_CP011854.1
Крупные эволюционные события:
А1,А4- делеция по Y, или вставка по X. Б2- делеция по X, или вставка по Y.
А2,А3 (Б3,Б1) - инверсия. Скорее всего, сначала произошла инверсия, а потом вставка/делеция.
| на главную |
© Гавриш Глеб 2016 |