Предсказание генов у прокариот
Описание плазмиды
Eubacteria
Отдел Firmicutes
Класс Bacilli
Порядок Lactobacillales
Семейство Streptococcaceae
Lactococcus lactis
from https://www.ncbi.nlm.nih.gov/sutils/static/GP_IMAGE/Lactococcus.jpg
Ход работы
Для начала, я получил два файла с помощью команды seqret:
seqret embl:lt599053 fasta::lt599053.fasta # Подключение к библиотеке embl и поиск id lt599053. Скачивание файла fasta. # На самом деле, файл fasta можно не качать, так как gff его уже содержит. Но так просто проще работать. seqret embl:lt599053 gff::lt599053.gff -feature # Тоже самое, но скачивается файл gff. # При этом используется boolean параметр -feature, который позволяет скачать дополнительную информацию.
После этого я прогнал файл fasta через prodigital
prodigal_windows.exe -i lt599053.fasta -o prodigal.fasta -f sco
Результат - prodigal.fasta
После этого, я сделал два скрипта, для перевода двух форматов в один - таблица с табуляцией (программы from_gff_to_table.py и from_prodigal_to_table.py).
После того, как файлы были приведены к одинаковому формату, я запустил другой скрипт, производящий нужные вычисления (main_program.py). Для упрощения, он сразу выдает информацию в html формате (если его об этом попросить).
Результат
| Количество полностью совпавших генов | 56 ( 65.88% ) |
| Количество совпадений по N концу | 0 ( 0.0% ) |
| Количество совпадений по C концу | 21 ( 24.71% ) |
| Количество полностью несовпавших генов | 8 ( 9.41% ) |
| Количество совпадений по одному из концов, при условии не совпадения направлений | 0 ( 0.0% ) |
| Общее количество генов | 85 ( 100% ) |
В ходе отладки программы я столкнулся с интересной вещью - при условии, что направление совпадает не хватает одного гена - то есть критерии не исчерпывающие. Так что я добавил еще два условия - Совпадение на N или С конце, но при этом не совпадение по направлению. Позже выяснилось, что помехи внес полный геном в аннотации (он имеет такое же оформление как и кодирующие участки). И хоть после его удаления из списка все вернулось в норму, я не стал добавлять их к полному несовпадению - мне они показались интересными.
Ссылка на архив - programs.rar. Краткое описание кода = небольшие комментарии внутри программ.
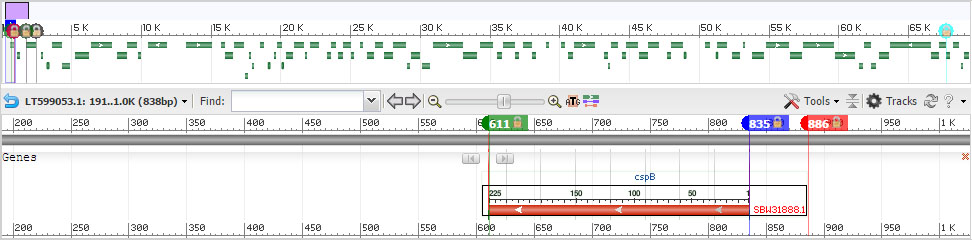
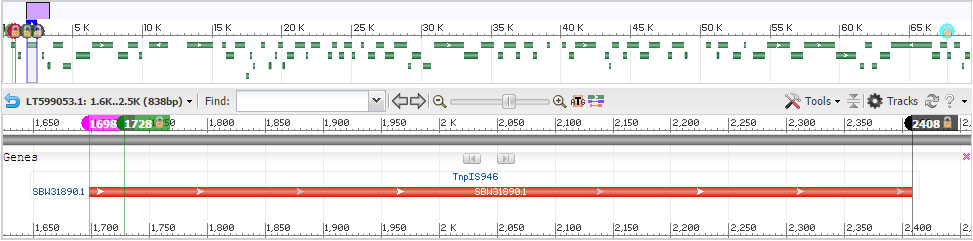
Предсказанные prodigal - 886 и 1728.
Так, как у меня нет примеров на разные концы генов, то в обоих случаях различается начало рамки считывания. В одном случае оно раньше, в другом позже. Оно и понятно - начать можно в совершенно разных местах, и все равно прочитать нужное, но вот дальше стоп-кодона прочесть не получиться.
| на главную |
© Гавриш Глеб 2016 |