Деревья
Из списка я выбрал следующие организмы:
| Мнемоника | Полное название |
| BACAN | Bacillus anthracis |
| CLOBA | Clostridium botulinum |
| FINM2 | Finegoldia magna |
| GEOKA | Geobacillus kanstophilus |
| LACAC | Lactobacillus acidophilus |
| STAAR | Staphylococcus aureus |
| STAES | Staphylococcus epidermidis |
По изображению дерева я составил следущую формулу:
((CLOBA,FINM2),(LACAC,((BACAN,GEOKA),(STAAR,STAES))));
По этой формуле в программе MEGA получилось изображение:
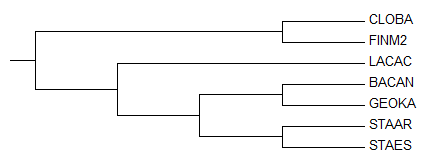
Из этого я делаю вывод, что дерево имеет следующие нетривиальные ветви:
1){STAAR,STAES} vs {BACAN,CLOBA,FINM2,GEOKA,LACAC}
2){BACAN,GEOKA} vs {CLOBA,FINM2,LACAC,STAAR,STAES}
3){CLOBA,FINM2} vs {BACAN,GEOKA,LACAC,STAAR,STAES}
4){BACAN,GEOKA,STAAR,STAES} vs {CLOBA,FINM2,LACAC}
Из нетревиальных ветвей я могу выделить следующие, показывающие разделение на таксоны:
1)Staphylococcus (genus)
2)Bacillaceae (family)
4)Bacillales (order)
Рассматривать я решил белок RF1 - фактор высвобождения пептидной цепи 1.
Я сформировал следующий запрос в jalveiw (очень удобно!):
RF1_BACAN;RF1_CLOBA;RF1_FINM2;RF1_GEOKA;RF1_LACAC;RF1_STAAR;RF1_STAES
Для использования этого запроса использовалось следующее окно:
Fetch Sequences... → Uniprot → окно сверху
С помощью алгоритма Muscle я посторил следующее выравнивание (в названиях оставил только мнемонику вида).
Используя метод Neighbor "Joining Using % Identity" было построено дерево:
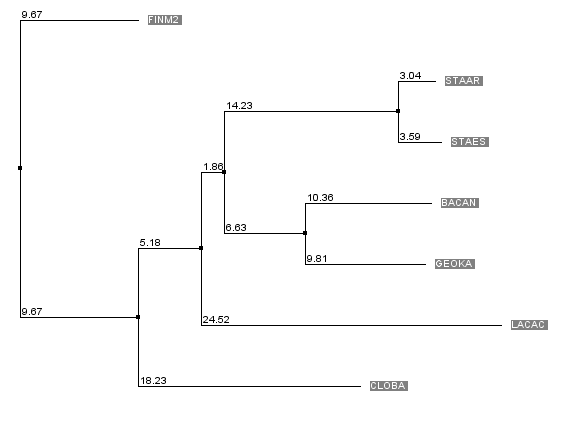
Ссылки: дерево (Newick), проект jalveiw (с выравниванием и деревом)
| на главную |
© Гавриш Глеб 2017 |