Реконструкция филогении по РНК
Построение дерева по нуклеотидным последовательностям
Цель - построить построить филогенетическое дерево "моих" бактерий по 16S rRNA.
Для этого я скачал файлы .frn из ftp://ftp.ncbi.nlm.nih.gov/genomes/archive/old_refseq/Bacteria/, из которых составил файл содержащий только последовательности гена 16S rRNA - 16s.fasta.
После этого я построил выравнивание (использовал Muscul через Jalview) и в MEGA построил дерево (Neighbor-Joining):
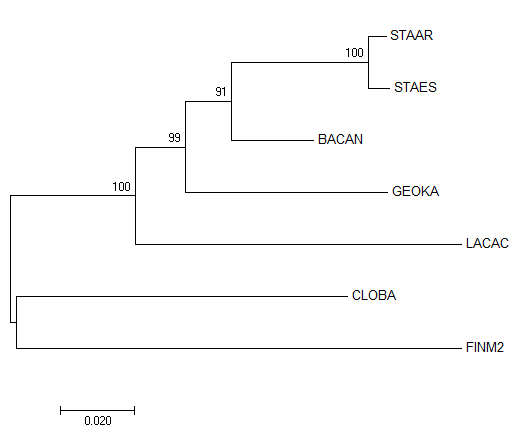
Рис.1 Дерево по 16s rRNA
По сравнению с правильным деревом, в данном отсуствует нетриваиальная ветвь {BACAN,GEOKA} vs {CLOBA,FINM2,LACAC,STAAR,STAES}, однако это не сильно искажает родственную картину.
Построение и анализ дерева, содержащего паралоги
Для того, что бы найти гомологи белка "ATP-dependent Clp protease ATP-binding subunit ClpX OS=Bacillus subtilis" в геномах моих бактерий, я использовал Blastp по протеомам соответствующих организмов. Гомологи были выравнены (Muscle), и на основе выравниваиня было посторенно дерево (Neighbor-Joining):
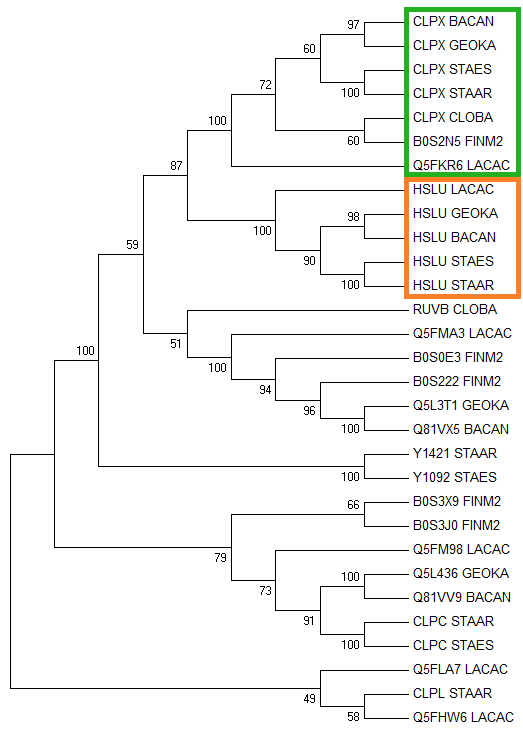
Рис.2 Дерево гомологов белка P50866
В дереве можно выделить две группы: с мнемоникой CLPX (в зеленой рамке) и HSLU (в оранжевой рамке). Эти две группы показывают наличие паралогов почти во всех бактериях (во второй группе не представленны FINM2 и CLOBA).
| на главную |
© Гавриш Глеб 2017 |