Работа с KEGG ORTHOLOGY
Выбор пары ортологических рядов для дальнейшей работы
| Название пути на английском | Nicotinate and nicotinamide metabolism |
| Название пути на русском | Метаболизм никотината и никотинамида |
| Выбранная реакция | Nicotinamide ↔ NAD⁺ |
| Кол-во EC для данной реакции | 3 |
| Выбранный EC | 3.2.2.5 |
| Число ортологических рядов | 2 |
| ID этих рядов | K18153, K19980 |
| Число генов выбранных ортологических рядов, представленных в базах данных | K18153 - 26, K19980 - 16 |
| Число белков выбранных ортологических рядов, представленных в базах данных | K18153 - 13 (13 в Uniprot), K19980 - 6 (3 в Uniprot) |
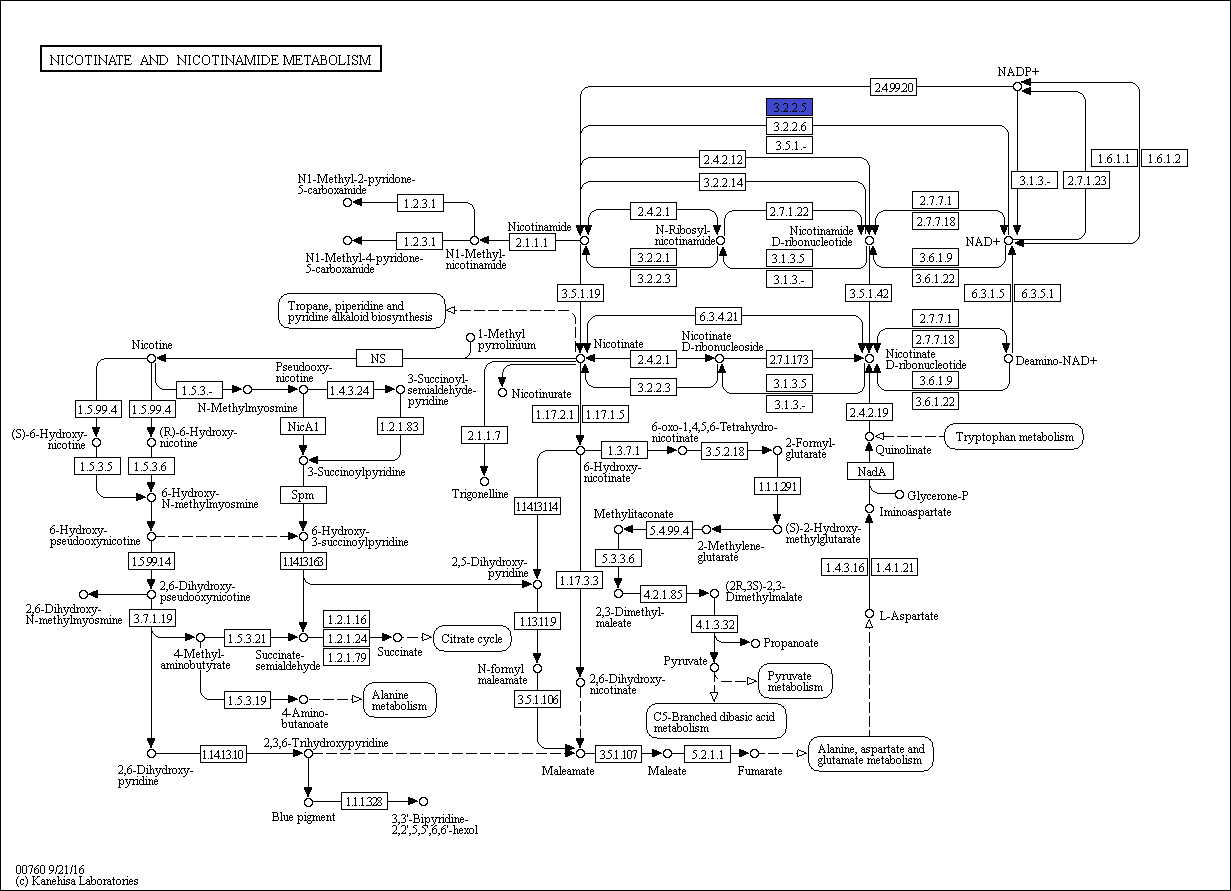
Рис. 1. Схема метаболического пути. Синим отмечен выбранный EC (3.2.2.5).
Получение совместного множественного выравнивания
Со страницы ортологических рядов, по ссылке Uniprot (справа, в доп. колонке All links), я перешел на страницу со всеми ID моих белков. Далее через сервис Retrieve/ID mapping я, по своим ID, получил последовательности моих белков в fasta формате (параметры: UniProtKB AC/ID - UniProtKB), скачав их всех в одном файле. В итоге я получил два разных файла для двух рядов (K18153.fasta и K19980.fasta).
С помощью сервиса Muscle (через Jalview) построено выравнивание.

Рис. 2. Выравнивание Muscle со стандартными параметрами.
Проверка гомологичности белков в выравнивании
Для начала я слегка улучшил выравнивание, произведя следующие манипуляции:
1) Вручную выровнял между собой группу K19980
2) Из группы K18153 сильно выбивается C0M7T1, так что я его выкинул из рассмотрения (много гэпов и замен).
По итогу я работал уже с таким выравниванием:

Рис. 3. Улучшенное выравнивание Muscle.
Посмотрев на это выравнивание, я пришел к следующим выводам:
1) K18153 довольно-таки близки к друг-другу. 2)Хоть и мало известных представителей K19980, однако между собой они также похожи (это видно на картинке внизу). 3) Возникают сомнения в ортологичности K18153 и K19980. Однако, K19980 - млекопитающие(мыши и крыса), а K18153 - грамположительные бактерии (стрептококки). Так что различие этих двух групп довольно предсказуемы.
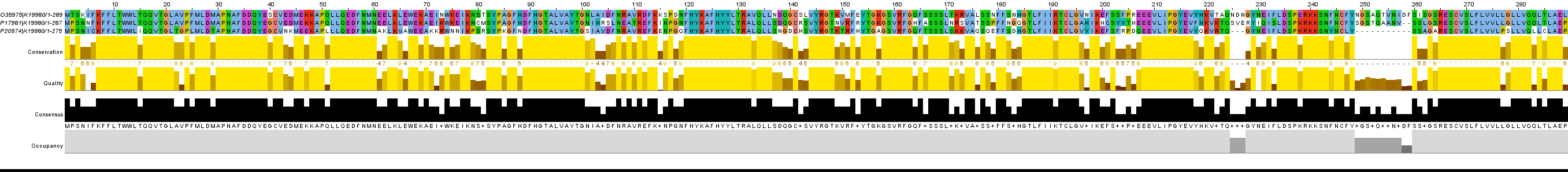
Рис. 4. Выравнивание K19980.
Рисунок 4 подтверждает, что между собой последовательности K19980 похожи.
Дерево
Из выравнивания следует, что скорее всего эти ряды не являются ортологами, исходя из чего дерево я строить не стал.
Так как один ряд представлен только прокариотами, а второй - эукариотами (грызунами), то можно предположить, что эти белки просто приобрели общую функцию в ходе эволюции, но общего предка-белка не имеют. В принципе, это довольно обычное дело для про- и эу-кариот.
| на главную |
© Гавриш Глеб 2017 |