Поиск сигналов. Chip-seq
Цель: Определите сайты связывания данного транскрипционного фактора в данном участке хромосомы человека
Контроль качества чтений
Для анализа мне достался один из многих участков хромосомы, полученных после ChiP-seq эксперимента. Он находится в файле chipseq_chunk23.fastq.
Я проанализировал его с помощью программы fastqc (установлена на kodomo):
fastqc chipseq_chunk23.fastq
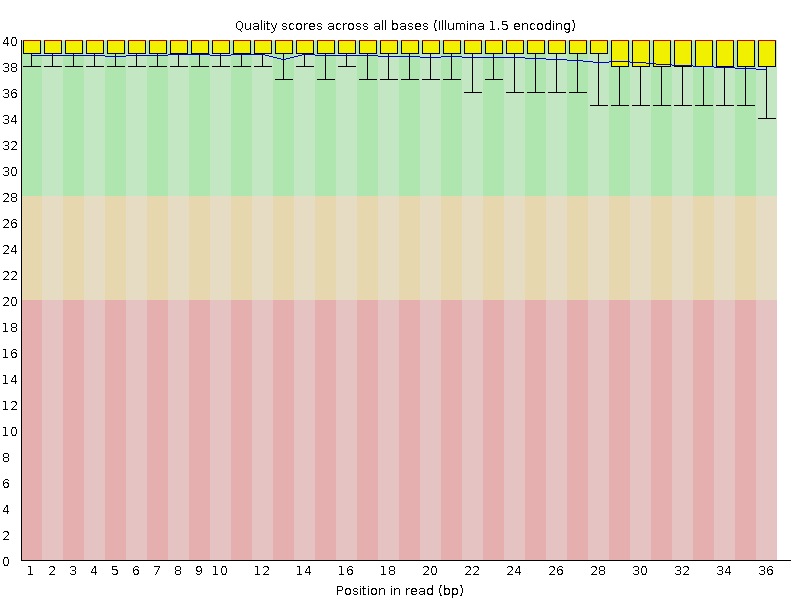
Рис. 1 результат анализа моего файла (качество рида)
Результат показал, что чтения чистить не надо, т.к. значения находятся в пределах приемлемых значений (зеленая зона). Полный анализ в html формате можно найти тут.
Картирование прочтений на геном человека hg19
Картирование прочтений на геноме человека hg19 осуществлялось с помощью программы BWA.
bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk23.fastq > chipseq_chunk23.sam
Далее рядом команд данные были преобразованы в вид, пригодный для понимания.
samtools view chipseq_chunk23.sam -bSo chipseq_chunk23.bam - файл с выравниванием переводится в бинарный формат .bam samtools sort chipseq_chunk23.bam -T chip_temp -o chipseq_chunk23_sort.bam - сортирует выравнивание ридов и референса по координате рида в референсе samtools index chipseq_chunk23_sort.bam - полученный файл сортируется samtools idxstats chipseq_chunk23_sort.bam > count.out - считает, сколько ридов откартировалось
В результате 7095 прочтений откартировались на 10-ую хромосому (? процентов?), что говорит о том, что изучать мне дали именно её.
Поиск пиков (Peak calling)
Для поиска пиков будет использовать программы MACS
macs2 callpeak -n chipseq_chunk23 -t chipseq_chunk23_sort.bam --nomodel
В результате было получено 3 файла: chipseq_chunk23_summits.bed, chipseq_chunk23_peaks.xls, chipseq_chunk23_peaks.narrowPeak.
Всего было найдено 10 пиков. Далее я визуализировал пики сервисом UCSC Genome Browser, загрузив в него следующий модифицированный файл: в файле chipseq_chunk23_peaks.narrowPeak первая строка была дополнена строкой:
track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk 23" browser position chr10:80733322-81288266
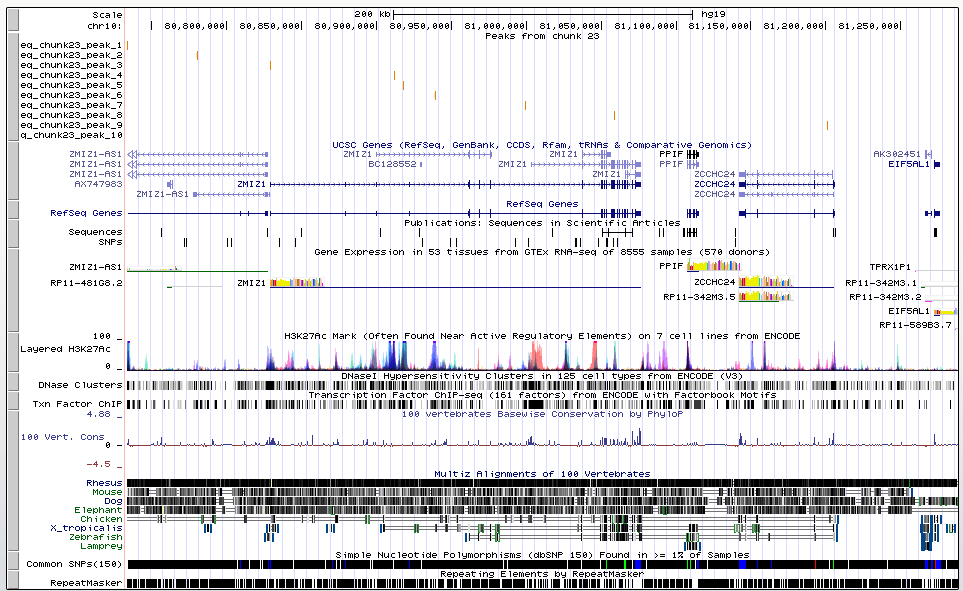
Рис.2. Визуалиция в UCSC Genome Browser пиков
Таблица 1. Характеристики пиков
| Номер | Координаты | Длина | Вершина пика | -log10(p-value) |
| 1 | 80733322 - 80733608 | 287 | 80733453 | 41.741 |
| 2 | 80779694 - 80780016 | 323 | 80779851 | 21.833 |
| 3 | 80828619 - 80828838 | 220 | 80828658 | 12.007 |
| 4 | 80911552 - 80911752 | 201 | 80911602 | 14.750 |
| 5 | 80917337 - 80917634 | 298 | 80917470 | 37.393 |
| 6 | 80938644 - 80938950 | 307 | 80938790 | 15.354 |
| 7 | 80999134 - 80999334 | 201 | 80999261 | 17.219 |
| 8 | 81058548 - 81058762 | 215 | 81058666 | 16.277 |
| 9 | 81200463 - 81200769 | 307 | 81200598 | 14.729 |
| 10 | 81288047 - 81288266 | 220 | 81288123 | 18.122 |
Каждому найденному пику соответствует число -log10(p-value), по которому можно оценить достоверность находки. Более высокие значения этого показателя означают более низкий p-value, а значит более высокую достоверность находки. Рассмотрим в более крупном масштабе пики с наименьшим p-value (номера 3 и 7 в Таблице 1):

Рис. 3. Пик 3 в более крупном масштабе
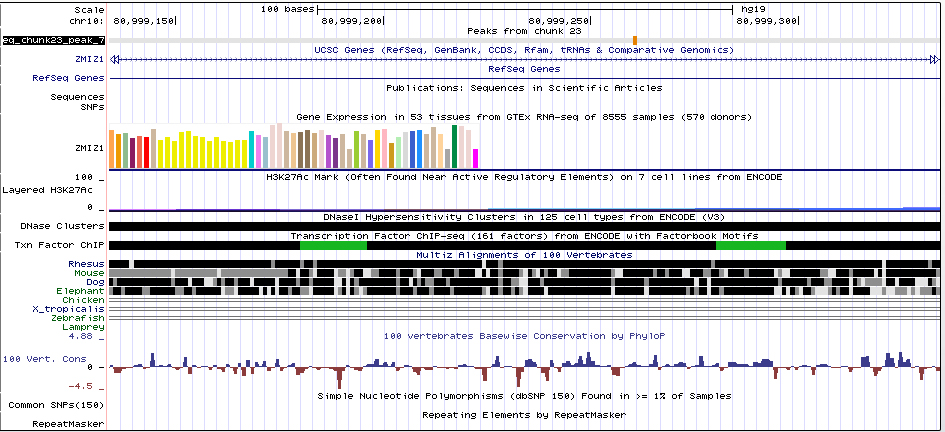
Рис. 4. Пик 7 в более крупном масштабе
Ген ZMIZ1 кодирует белок содержащий домен с цинковым палецем.
| на главную |
© Гавриш Глеб 2017 |