Поиск сигналов. Теория
Определение биологической роли определенного транскрипционного фактора в бактерии
Для изученя я взял бактерию Bifidobacterium dentium Bd1. Из файла LexA_Bifidobacteriaceae я нашел последовательности, относящиеся к моей бактерии, и записал их в файл Bd1.fasta. Его я и подал для поиска MEME. Результат:
E-value = 2.1e-039 ;PWM:
0.000000 0.000000 0.000000 1.000000 0.111111 0.777778 0.111111 0.000000 0.111111 0.000000 0.888889 0.000000 1.000000 0.000000 0.000000 0.000000 1.000000 0.000000 0.000000 0.000000 0.000000 1.000000 0.000000 0.000000 0.555556 0.000000 0.444444 0.000000 0.000000 0.000000 0.666667 0.333333 0.777778 0.000000 0.222222 0.000000 0.000000 0.000000 0.000000 1.000000 0.000000 0.000000 1.000000 0.000000 0.000000 0.000000 0.000000 1.000000 0.111111 0.000000 0.000000 0.888889 0.000000 0.888889 0.000000 0.111111 0.000000 0.000000 0.777778 0.222222 0.888889 0.000000 0.000000 0.111111

Рис. 1. Logo сайта связывания транскрипционного фактора, полученное с помощью программы MEME.
Результат работы MEME я перенаправил в Tomtom:
Таблица 1. Характеристика находок/лучшей находки Tomtom
| Число значительных находок мотивов с похожей PWM в последовательности | Название TФ находки с похожей PWM | E-value лучшей находки | ориентация |
| 14 | EXPREG_00000020 (LexA_C.glutamicum) | 1.95e-03 | обратная |
Выдачу MEME я так же использовал для програмы FIMO. Мотив нужно искать только в upstream region, потому что большинство транскрипционных факторов связываются с последовательностью до гена по направлению транскрипции (т.е. в upstream region). Результат:
fimo.txt - таблица (если вставлять на страницу, то все равно ничего не понятно т.к. данных много).
Проверка, может ли метилирование повлиять на связывание исследуемого ТФ со своим сайтом
Используя программу fuzznuc (пакет EMBOSS) я провел поиск сайтов митилирования, по upstream региону (т.к. метилирование может регулировать эксперссию генов, вполне логично искать именно там).
fuzznuc -sequence Bd1.fasta -pattern @MT_sites.txt -outfile fuzznucX.out
Результат храниться в файле fuzznucX.out.
Всего было проверенно 9 последовательностей, число нахдок - 49. Разброс от 4 до 8 находок на одну последовательность.
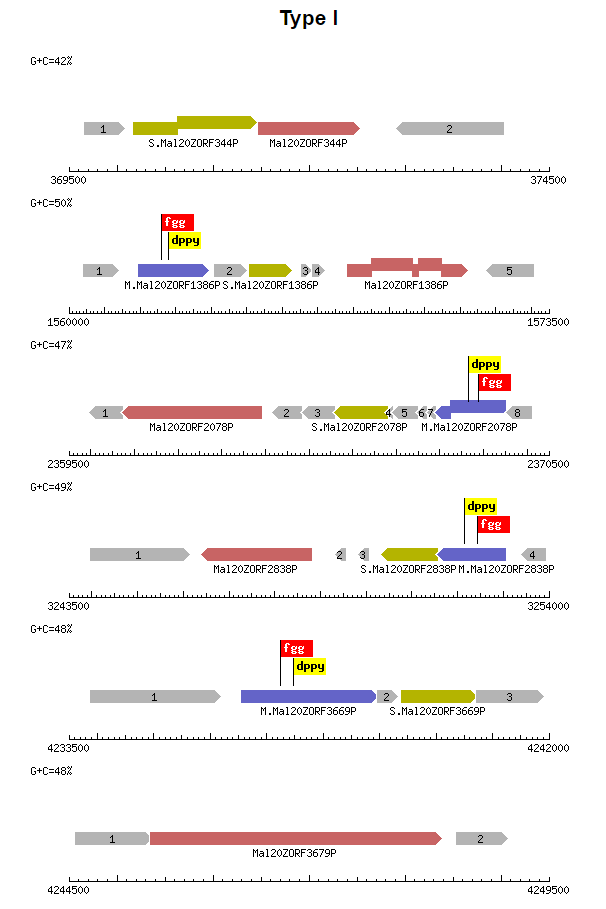
Рис. 2. Сайты метитрансфераз генома Methylomicrobium alcaliphilum 20Z, Тип I.

Рис. 3. Сайты метитрансфераз генома Methylomicrobium alcaliphilum 20Z, Тип II.

Рис. 4. Сайты метитрансфераз плазмиды Methylomicrobium alcaliphilum 20Z.
Рис. 5. Тоже, но в виде окружности.
Так как у моей бактерии неизвестен нуклеотидный состав сайтов метелирования, то сравнение с предыдущим предсказанием представляется невозможным.
| на главную |
© Гавриш Глеб 2017 |