Реконструкция эволюции доменной архитектуры
Цель: Реконструировать эволюцию доменной архитектуры белков, содержащих один и тот же домен Pfam
Задача 1. Построить выравнивание представителей домена Pfam белков с разной доменной архитектурой
Для дальнейшей реконструкции эволюции доменной архитектуры белков был выбран N-концевой домен ГТФ-связывающей ГТФазы (GTP-binding GTPase N-terminal).
| Pfam ID | Pfam AC | Описание | Доменные архитектуры |
| GTP-bdg_N | PF13167 | N-концевой домен ГТФ-связывающей ГТФазы | 22 архитектуры, 4191 последовательность; 8 структур |
Загруженное с помощью JalView выравнивание из Pfam (File > Fetch Sequences по идентификатору PF00134) было раскрашено по консервативности (ClustalX) -> all_aligned.jvp (ссылка на проект Jalview)
Выбор архитектур и таксонов
Для работы я выбрал следующие две архитектуры, содержащие исследуемый домен:
| ID | V4Y7K7_9ARCH | X8ATW7_MYCXE |
| название(кто включен) | GTP-bdg_N, MMR_HSR1 | GTP-bdg_N |
| кол-во последовательностей | 57 | 41 |
| схема |  |  |
Далее с помощью скрипта swisspfam_to_xls.py были отобраны последовательности с исследуемым доменом из файла /srv/databases/pfam/swisspfam.gz с информацией об архитектуре всех последовательностей:
python swisspfam-to-xls.py -z -i /srv/databases/pfam/swisspfam.gz -p PF13167 -o arc.xls
В результате я получил файл arc.xls, после чего сохранил в файл arc_my.txt только свой домен.
Сервисом Uniprot я поднял последовательности для моих ID.
была получения таксономия с помощью скрипта uniprot_to_taxonomy.py:
python uniprot-to-taxonomy.py -i uniprot.txt -o tax.xls
В результате был получен файл с таксономией - tax.xls.
Я выбрал таксон Ecdysozoa и два его подтаксона: Arthropoda и Nematoda
python filter-alignment.py -i align.fasta -m id.txt -o align_selected.fa -a
Анализ выравнивания и таксономии
Для данных таксонов я посторил выравнивание (Muscle), а также филогенетическое дерево (NJ):
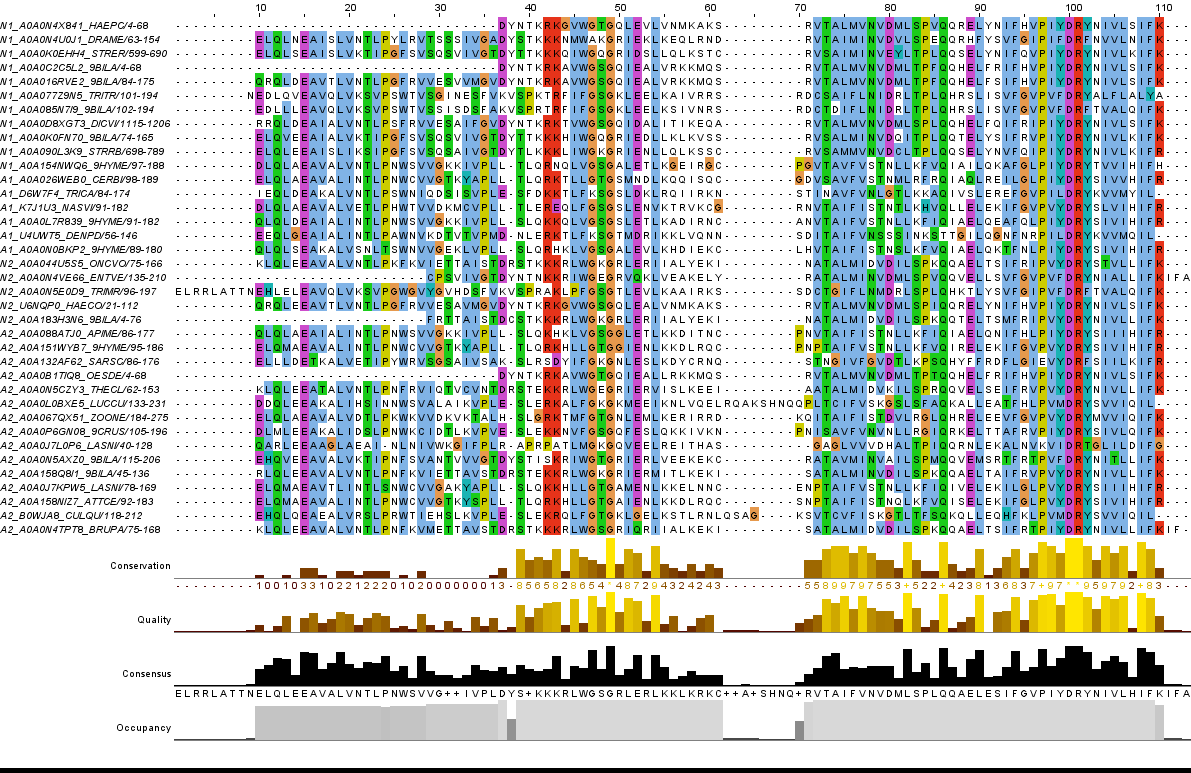
Рис. 1. Выравнивание выбранных последовательностей с разделением на группы по архитектурам
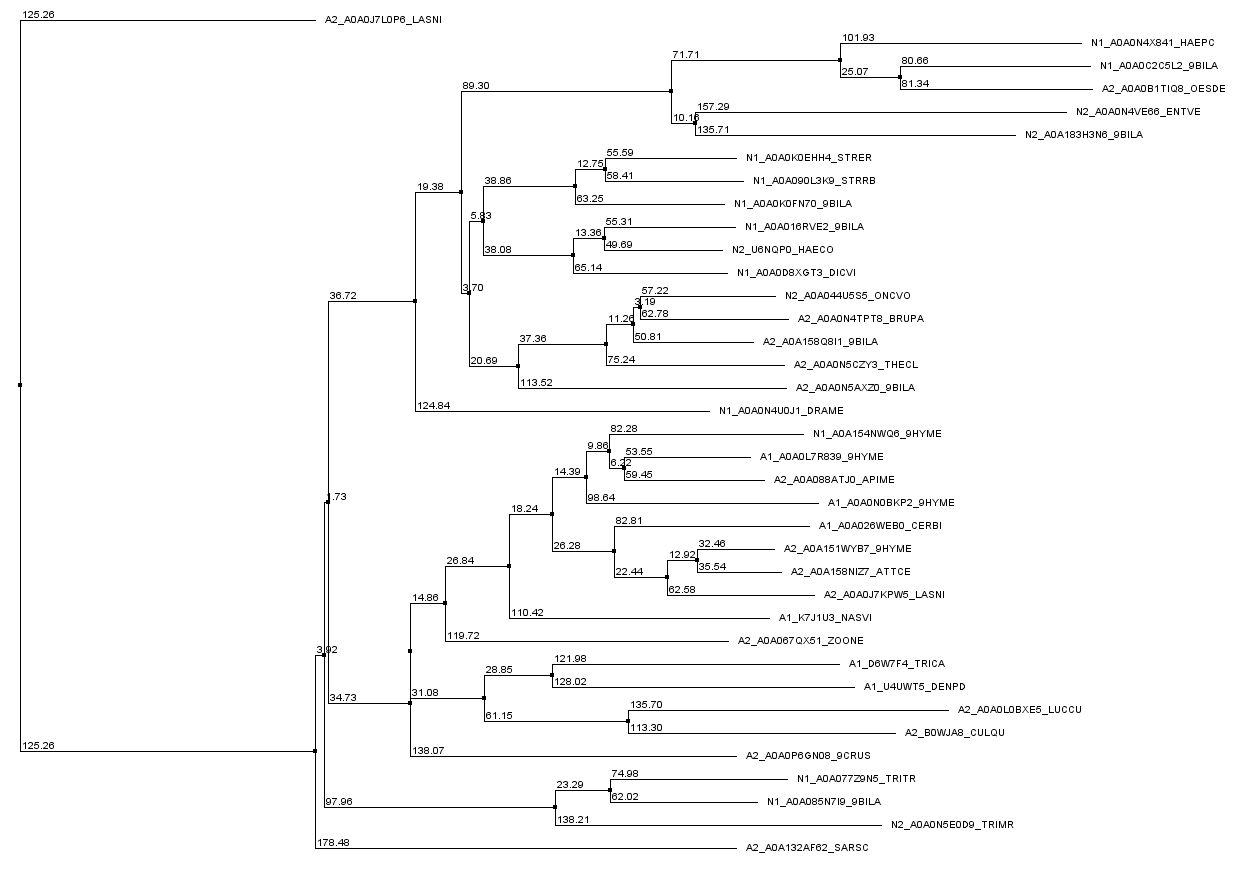
Рис. 2. Дерево, построенное по выравниванию выбранных последовательностей методом NJ
По выравниванию видно, что последовательности имеют общее, однако меня такое выравнивание не сильно убеждает. Я все же решил построить дерево, и вот по нему уже видно, что не происходит разделение ни по таксонам, ни по архитектурам.
| на главную |
© Гавриш Глеб 2017 |