Реконструкция филогенетического дерева с помощью 4-х разных методов.
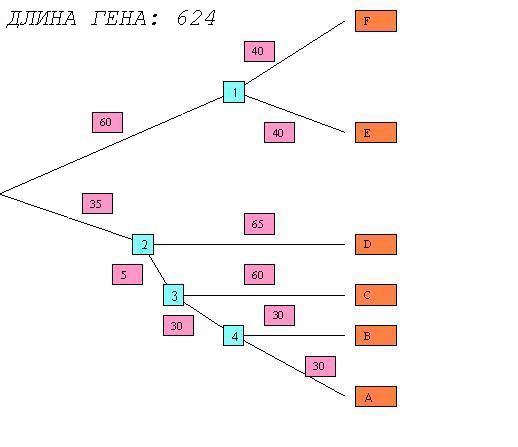
Дерево, востановленное по скобочной формуле.
Матрица попарных расстояний, выданная ednadist.
a 0.0000 0.4237 0.9782 1.1281 1.4250 1.6925
b 0.4237 0.0000 0.9665 1.1209 1.5282 1.4922
c 0.9782 0.9665 0.0000 1.1500 1.7399 1.4143
d 1.1281 1.1209 1.1500 0.0000 1.5660 1.4358
e 1.4250 1.5282 1.7399 1.5660 0.0000 0.5939
f 1.6925 1.4922 1.4143 1.4358 0.5939 0.0000
Дерево, построенное по методу UGPMA с помощью программы eneighbor.
+------------a
+---------------1
+----3 +------------b
! !
+-----------4 +----------------------------c
! !
--5 +---------------------------------d
!
! +-----------------e
+---------------------------2
+-----------------f
((((a:0.21185,b:0.21185):0.27433,c:0.48618):0.08032,d:0.56650):0.20187,
(e:0.29695,f:0.29695):0.47142); |
Дерево, построенное по методу ближайших соседей (Neighbor-Joining) с помощью программы eneighbor.
+-------------------------------d
!
! +------------------e
--4---------------------------------------1
! +---------------f
!
! +------------a
! +--------------2
+------3 +-----------b
!
+-----------------------------c
(d:0.54118,(e:0.32499,f:0.26891):0.66277,((a:0.22309,b:0.20061):0.25729,
c:0.50321):0.10989); |
Дерево, построенное по методу наибольшего правдоподобия (Maximum Likelihood)с помощью программы ednaml.
+-----b
!
! +----------f
! +----------------------4
! +------3 +--------e
! ! !
--1--------2 +-----------------d
! !
! +-----------------c
!
+------a
(b:0.19911,(((f:0.35260,e:0.28225):0.76648,d:0.59020):0.21329,
c:0.59317):0.29733,a:0.23427);
|
Дерево, построенное по методу максимальной экономии (Parsimony) с помощью программы ednapars.
+--f
+--5
+--4 +--e
! !
+--3 +-----d
! !
+--2 +--------c
! !
--1 +-----------b
!
+--------------a
(((((f,e),d),c),b),a);
|
Cравнение топологии исходного филогенетического дерева модели и 4-х вариантов его реконструкции
| Ветвь |
Исходное дерево
модели |
UPGMA |
NJ |
ML |
Parsimony |
| ABCDEF |
| 110000 |
+ |
+ |
+ |
+ |
+ |
| 111000 |
+ |
+ |
+ |
+ |
+ |
| 111100 |
+ |
+ |
+ |
+ |
+ |
КАК ВИДНО ИЗ ТАБЛИЦЫ, ЕСЛИ РАССМАТРИВАТЬ ТОЛЬКО
ТОПОЛОГИЮ ДЕРЕВЬЕВ, ТО ВСЕ ДЕРЕВЬЯ ЯВЛЯЮТСЯ
ПРАВИЛЬНЫМИ, НО ЕСЛИ УЧИТЫВАТЬ ОСТАЛЬНЫЕ
ОСОБЕННОСТИ, ТАКИЕ КАК ОТНОСИТЕЛЬНЫЕ РАССТОЯНИЯ
МЕЖДУ ПОСЛЕДОВАТЕЛЬНОСТЯМИ, ТО БОЛЕЕ
ПРЕДПОЧТИТЕЛЬНО ДЕРЕВО, ПОСТРОЕННОЕ ПО МЕТОДУ
UGPMA. В ЭТОМ СЛУЧАИ МЫ ПОЛУЧАЕМ НАИБОЛЕЕ БЛИЗКОЕ
К ИСТИННОМУ ДЕРЕВО.
©
Бабаян Тигран, 2005