Анализ множественных выравниваний
Для выполнения данной работы я использовала построенное c помощью программы JalView в предыдущем практикуме выравнивание последовательностей Uniprot со следующими идентификаторами:
- I4C028_DESTA
- D3V4H4_XENBS
- E3HYT5_RHOVT
- V2VXE3_9GAMM
- F4LVQ3_TEPAE
Задание 1. Поиск блоков в выравнивании
Используемое техническое определение
Вертикальный блок - участок множественного выравнивания не менее чем 5 последовательностей, обладающий следующими свойствами:
- не содержит гэпов;
- длина - не менее 4 колонок;
- первая и последняя колонки - абсолютно консервативны или абсолютно функционально консервативны;
- не содержит более 3 колонок, не являющихся абсолютно консервативными или абсолютно функционально консервативными, подряд;
- не может быть расширен без нарушения предыдущих свойств.
1а. Выделение вертикальных блоков
Используя вышеприведенное определение, я выделила в своём выравнивании 6 вертикальных блоков, создала для них разметку Vertical blocks, где каждая колонка обозначена
буквой B.

1b. Один блок из части последовательностей
Три последовательности (с идентификаторами GCSH_BURXL, Q8II35_PLAF7, D7CE49_STRBB) показались мне более схожими, и я объединила их в группу. Внутри этой группы был выделен
блок (21-28 позиции, отмечен символом 'H'). Важно отметить тот факт, что по биологическому
опопределению блока, мы нашли гомологичные аминокислоты, но согласно определению техническому мы не можем судить о гомологичности аминокислот, так как блок мы нашли в группе,
которая включает лишь 3 последовательности из 5, а значит, в техническом смысле это блоком не является.
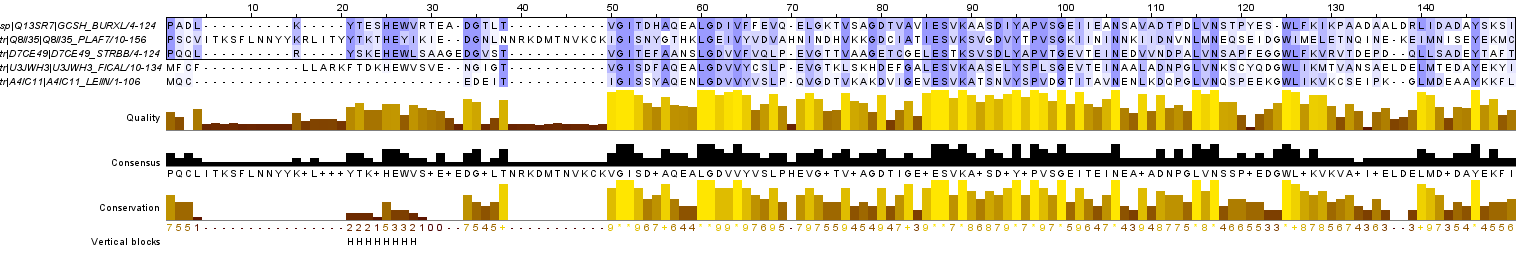
Задание 1 в проекте JalView
Задание 2. Статистика
2a. Число и процент консервативных позиций
Данные приведены для одного вертикального блока с координатами 60-68 (9 колонок).

| Позиции | Число | Процент |
|---|---|---|
| Абсолютно консервативные | 3 | 33,(3)% |
| Абсолютно функционально консервативные | 5 | 55,(5)% |
2b. Число и процент позиций с гепами для самого длинного участка, не входящего в состав блоков
Данный участок состоит из 49 колонок (координаты 1-49). Число позиций с гэпами - 41, что составляет 83,7%.
Задание 2 в проекте JalView
Задание 3. Консенсусная последовательность и LOGO одного блока
Консенсусная последовательность (consensus/canonical sequence) - некая усредненная последовательность
(возможны небольшие вариации в аминокислотных или нуклеотидных остатках). Обычно характерна для генов, кодирующих один и тот же белок у разных организмов, т.к. ее
составляют наиболее часто встречающиеся нуклеотиды/аминокислоты. Консенсусная последовательность создается на основе массива выравнивания, часто является эталонной
в анализе.[1]
С помощью программы cons на сервере я построила консенсусную последовательность для блока 60-68. На входе программы - выравнивание
последовательностей, на выходе - нужная консенсусная последовательность в одном из распространённых форматов (FASTA, Genbank, SwissProt...).
LOGO блока можно получить, используя следующий сервис, который принимает на вход совокупность последовательностей
из выравнивания и выдает построенное по ней изображение в разрешении по выбору. По умолчанию выбрана раскраска по гидрофобности: для гидрофобных аминокислот она черная,
для гидрофильных - синяя, для нейтральных - зеленая.
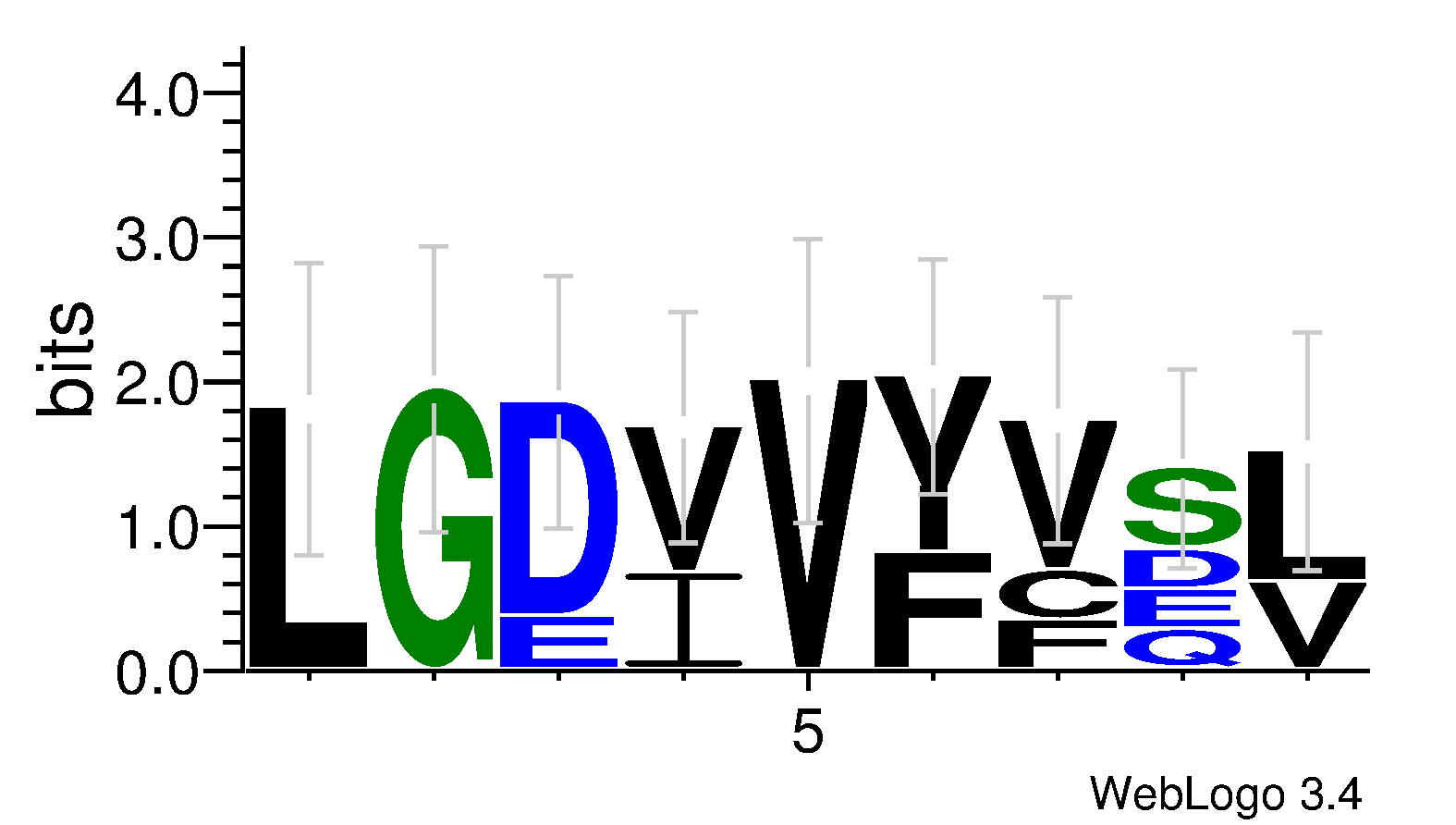
Задание 4. Паттерн для выбранного блока
Паттерн для блока с координатами 60-68: L-G-[ED]-[IV]-V-[YF]-[VFC]-[DQSE]-[VL].
Задание 5. Выравнивание с заведомо негомологичной последовательностью белка NP_809341.1
Построение искусственного выравнивания данных последовательностей с полседовательностью белка бактероида показало, что максимальное число совпадений негомологичной последовательности с консервативными позициями в блоках равно 14. В пересче в проценты - около 29%, что все же свидетельствует о малой вероятности гомологичности белков, а следовательно, о дальних систематических связях организмов, у которых они встречаются. Совпадения в разметке False coincidence обозначены буквой F.

Задание 6. "Выравнивание" заведомо негомологичных белков
Попытка построить выравнивание для неродственных белков с указаннами идентификаторами (NP_820762.2; NP_603408.1; NP_354235.1; NP_354235.1; NP_274428.1; NP_228195.1) оказалось неудачной: получилось слишком много гэпов, не удалось найти ни одной абсолютно консервативной колонки, количество абсолютно функциональных колонок тоже было недостаточным для построения хорошего выравнивания. Количество полученных блоков - 3. Таким образом, можно убедиться в том, что метод выравнивания последовательностей действительно отражает гомологичность в биологическом понимании термина.

Две наиболее похожие последовательности мне удалось объединить в группу, для этого в блоке с координатам 397-400 я выбрала подблок из 2 последовательностей, опираясь на то, что в них есть 3 абсолютно консервативные позиции, но при рассмотрении этих же колонок для совокупности всех последовательностей, то исчезают абсолютно консервативные колонки, 398 колонка (для 6 последовательностей) не является даже абсолютно функционально консервативной, то есть, чем меньше последовательностей в "выравнивании", тем легче найти "гомологичные учаски".
Задание 6a в проекте JalView
Задание 6b в проекте JalView

Для выполнения этой части задания я рассмотрела блок с координатами 397-400 (из 4 колонок).
| Позиции | Число | Процент |
|---|---|---|
| Абсолютно консервативные | 0 | 0% |
| Абсолютно функционально консервативные | 3 | 50% |
Максимальный участок, не образующий блоков, состоит из 385 колонок (координаты 12 - 396)
Число позиций с гэпами - 64, что составляет 16.62%, данный показатель значительно меньше аналогичного в хорошем выравнивании.
Задание 2 в проекте JalView

Источники:
[1]База знаний по биологии человека[2] Руководство к JalView
[3] www.bioinformatics.nl/emboss-explorer/
[4] http://weblogo.threeplusone.com/
[5] UniProt