Сборка геномов de novo.
В данном практикуме мне был выдан код доступа к проекту по секвенированию генома Buchnera aphidicola (
SRR4240382).
Файл .fastq c чтениями (секвенирование Illumina) был обработан программой Trimmomatic. Таким образом были удалены адаптеры. Сначала был создан файл adapters.fasta с собранием всех известных адаптеров.
Выдача программы:
Подготовка k-меров была произведена в программе velveth. Она предназначена для создания набора данных для последующей обработки программой velvetg. Velveth принимает на вход несколько последовательностей, строит хэш-таблицу, в результате получаются файлы Sequences и Roadmaps (также Log с отчетом о работе) в отдельной директории. Длина k-мера (хэша) соответствует длине хэшируемого слова в п.о. В задании она была указана: k=29. Выбор данного параметра зависит от технической задачи:
Команда запуска:
Cборка на основе k-меров с помощью программы velvetg на вход получила данные из предыдущего этапа. Velvetg строит граф де Брёйна - ориентированный n-мерный граф из m символов, отражает пересечения между последовательностями символов. Он имеет mn вершин, состоящих из всех возможных последовательностей длины n из данных символов (т.е. k-меры являются вершинами), ориентированные рёбра соединяют так называемые префикс- и суффикс-последовательности. Один и тот же символ может несколько раз встречаться в последовательности. [4] При запуске программы с параметрами по умолчанию можно получить файл .fasta с контигами и статистические данные в указанной директории.
Команда запуска:
Для анализа были взяты три самых длинных контига (ID 1, ID 13, ID 8). Каждый из них был обработан по алгоритму blastn megablast в паре с хромосомой Buchnera aphidicola (GenBank AC: CP009253).
* В таблице приведены только участки выравниваний с максимальным Score.
Построенные для 3 максимальных по длине контигов выравнивания оказались вполне приемлемыми. Аналогичный анализ для двух контигов с аномально большим покрытием выполнить не удалось, так как длина исследуемых последовательностей оказалась абсурдно маленькой, для построения выравнивания (можно попробовать изменить Expect thershold, но мне это не помогло). По данным FAQ программы, возможной причиной ошибки может также являться фильтрация участков малой сложности. Поиск без фильтрации, тоже не дал результатов.
Buchnera aphidicola относится к гамма-протеобактериям, является первичным эндосимбионтом гороховой тли Acyrthosiphon pisum, питающейся соком растений. По мнению исследователей, предками данного вида могли быть свободноживущие грамм-отрицательные бактерии, близкие к современным энтеробактериям (таким, как E. coli). Анализ последовательностей 16S rRNA подтверждает родство с семейством Enterobacteriaceae. В диаметре достигают 3 мкм, компоненты клеточной стенки во многом схожи с таковыми у энтеробактерий. Однако, в отличие от большинства грам-отрицательных бактерий, у бухнеры нет генов, отвечающих за синтез LPS. Изучение данного вида позволит уточнить эволюционные механизмы и углубить знания о внутриклеточном эндосимбиозе. [1] 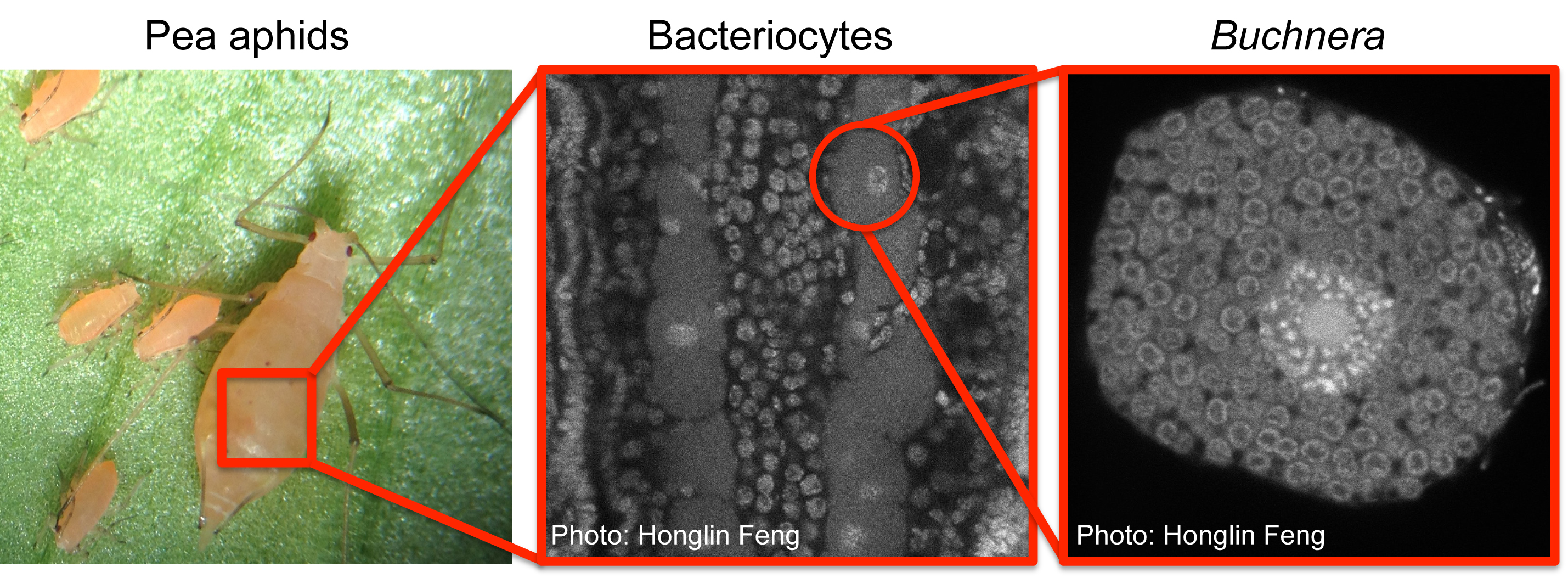 Buchnera проживает в специализированных клетках тлей - бактериоцитах, где синтезирует незаменимые аминокислоты для тли-хозяина. Бактериоциты организованы в парный орган - бактериом. [2] |
1. Подготовка чтений программой Trimmomatic.
Файл .fastq c чтениями (секвенирование Illumina) был обработан программой Trimmomatic. Таким образом были удалены адаптеры. Сначала был создан файл adapters.fasta с собранием всех известных адаптеров.
| Использованная команда | Описание | Результат |
java -jar /usr/share/java/ | Удаление остатков адаптеров, обрезка нуклеотидов с качеством ниже 20 и отбор чтений длины не менее 30 (где 2 - отдельные несовпадения, 7 - порог для палиндромной шпильки, 7 - порог для простой шпильки) | SRR4240382_trim.fastq |
Выдача программы:
Input Reads: 17756177 Surviving: 11919768 (67,13%) Dropped: 5836409 (32,87%)Размер файла уменьшился с 1808 Мб до 1173 Мб, что говорит о достаточно хорошем результате чистки. Однако, скорость работы программы не самая быстрая.
2. Подготовка k-меров.
Подготовка k-меров была произведена в программе velveth. Она предназначена для создания набора данных для последующей обработки программой velvetg. Velveth принимает на вход несколько последовательностей, строит хэш-таблицу, в результате получаются файлы Sequences и Roadmaps (также Log с отчетом о работе) в отдельной директории. Длина k-мера (хэша) соответствует длине хэшируемого слова в п.о. В задании она была указана: k=29. Выбор данного параметра зависит от технической задачи:
- k нечетно для избежания палиндромов
- k<=31 по дефолту (31 - max хэш-длина по умолчанию, может быть изменена)
- k меньше длины ридов (иначе отсутствуют перекрывания)
Команда запуска:
velveth velveth 29 -short -fastq SRR4240382_trim.fastq
3. Сборка на основе k-меров.
Cборка на основе k-меров с помощью программы velvetg на вход получила данные из предыдущего этапа. Velvetg строит граф де Брёйна - ориентированный n-мерный граф из m символов, отражает пересечения между последовательностями символов. Он имеет mn вершин, состоящих из всех возможных последовательностей длины n из данных символов (т.е. k-меры являются вершинами), ориентированные рёбра соединяют так называемые префикс- и суффикс-последовательности. Один и тот же символ может несколько раз встречаться в последовательности. [4] При запуске программы с параметрами по умолчанию можно получить файл .fasta с контигами и статистические данные в указанной директории.
Команда запуска:
velvetg velvethРезультаты работы velvetg:
- изначально 2430680 "пре"-вершин, в итоге — 3266 (количество вершин не обязательно соответствует количеству контигов, так как мы ограничили снизу контиги длиной 29, "нормальные" контиги описаны в файле contigs.fa);
- N50 = 13439;
- max длина контига — 67459;
- файлы с графами: PreGraph, LastGraph, Graph;
- файл с информацией о вершинах графа - stats.txt:
1) длина вершины в k-мерах (для расчёта длины в нуклеотидах добавляем k–1);
2) in — число входящих рёбер с 5'-конца;
3) out — число выходящих рёбер с 3'-конца;
4) колонки *_cov и *_Ocov содержат информацию о покрытии с разницей в методе подсчета (*_Ocov — более строгий) - файл с контигами в формате .fasta — contigs.fa (последовательности длины не менее 2k, можно установать специально ограничение на минимальную длину контига, чего мы не делали);
- встречаются контиги с аномально большими покрытиями (как, например, контиг с ID 3234 и покрытием 43986, однако его длина составляет 1 k-мер, чем и объясняется такое большое отклонение покрытия);
- достаточно сложно судитьо контигах с аномально маленьким покрытием, так как их слишком много (только контигов с покрытием меньше 5 несколько сотен)
Длины и покрытия самых больших контигов


4. Анализ.
Для анализа были взяты три самых длинных контига (ID 1, ID 13, ID 8). Каждый из них был обработан по алгоритму blastn megablast в паре с хромосомой Buchnera aphidicola (GenBank AC: CP009253).
Сравнение самых длинных контигов с хромосомой Buchnera aphidicola
| ID | Координаты в геноме | Max score (bits) | Total score | Query cover | E-value | Identities | Gaps |
| 1 | 532032-550219 | 12576 | 26469 | 64% | 0.0 | 14656/18457 (79%) | 488/18457 (2%) |
| 8 | 295935-303252 | 3947 | 13037 | 67% | 0.0 | 5696/7429 (77%) | 186/7429 (2%) |
| 13 | 37007-44693 | 6909 | 19815 | 72% | 0.0 | 6424/7741 (83%) | 97/7741 (1%) |
* В таблице приведены только участки выравниваний с максимальным Score.
Построенные для 3 максимальных по длине контигов выравнивания оказались вполне приемлемыми. Аналогичный анализ для двух контигов с аномально большим покрытием выполнить не удалось, так как длина исследуемых последовательностей оказалась абсурдно маленькой, для построения выравнивания (можно попробовать изменить Expect thershold, но мне это не помогло). По данным FAQ программы, возможной причиной ошибки может также являться фильтрация участков малой сложности. Поиск без фильтрации, тоже не дал результатов.