Комплексы ДНК-белок
Суть работы - сравнительный анализ канонической ДНК и стеблей тРНК.
Задание 1.
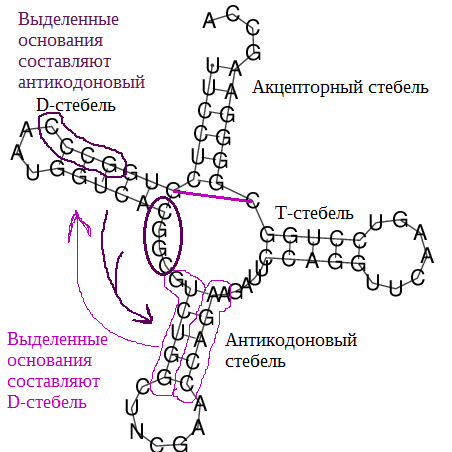
Предсказание вторичной структуры заданной тРНК.[1]
Упражнение 1.
Предсказание вторичной структуры тРНК путем поиска инвертированных повторов.
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных
последовательностях, на вход получая последовательность нуклеотидов (USA), как, например, в файле данной мне тРНК 1f7u.fasta. На выходе мы имеем два файла:
[sequence.fasta], с данными о полученных комплементарных участках, и [sequence.inv], показывющий водородные связи в пределах этих участков.
При попытке запуска программы со стандартными параметрами создавались пустые файлы, поэтому для получения предсказания наиболее близкого к реальной структуре параметр
"Minimum score threshold" был изменен с базового (50), на подобранный (15), при котором появился первый и, видимо, наиболее удачный результат. По результатам сравнения
с find_pair программа einverted смогла верно предсказать структуру T-стебля и сделала ошибку со сдвигом на 1 основание в структуре D-стебля.
Упражнение 2.
Предсказание вторичной структуры тРНК по алгоритму Зукера.
Другая программа RNAfold из пакета Vienna RNA Package, которая реализует алгоритм Зукера. RNAfold принимает на вход
последовательность, вторичную структуру РНК рассчитывает с учетом минимальной свободной энергией (mfe), на выходе дает файл в формате DBN (Dot-Bracket Notation). Точками
обозначаются нуклеотиды без пары, спаренные - круглыми скобками. Псевдоузлы отмечены круглыми и квадратными скобками. Для моей тРНК RNAfold сработала
недостаточно хорошо, что подтверждают данные онлайн-сервиса о низкой вероятности образования пар.
По его рассчету возможна отличная от клевера структура - РНК-центроид, вторичная структура с минимальным расстоянием пар оснований относительно остальной части молекулы
в ансамбле Больцмана. RNAfold почти идеально предсказала 2 из 4 стеблей, для двух других предсказание оказалось очень сильно отличающимся от других
программ.
В целом, результаты работы 3 программ получились достаточно неоднозначными, чтобы делать выводы об их эффективности, а для предсказания структуры тРНК с PDB ID 1f7u требуется
использование стороннего софта и дополнительное исследование.
| Реальная и предсказанная вторичные структуры тРНК из файла 1f7u.pdb | |||
|---|---|---|---|
| Участок структуры | Позиции в структуре (результаты find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5`-901-907-3` 5`-966-972-3` всего 7 пар | предсказано 0 пар из 7 реальных | предсказано 6 пар из 7 реальных, не хватает связи между парой оснований 5`-907-966-3` |
| D-стебель | 5`-939-944-3` 5`-926-931-3` всего 6 пар | предсказано 5 пар из 6 реальных,
отсутствует пара 5`-926-944-3` | предсказано 0 пар из 5 реальных |
| T-стебель | 5`-949-953-3` 5`-961-965-3` всего 5 пар | верно предсказаны все 5 из 5 пар,
начало и конец совпадают | верно предсказан весь стебель |
| Антикодоновый стебель | 5`-910-913-3` 5`-922-925-3` всего 4 пары | предсказано 0 из 4 пар | предсказано 0 из 4 пар |
| Общее число канонических пар нуклеотидов | 23 | 10 | 11 |
| Предсказанная вторичная структура тРНК с минимальной свободной энергией, RNAfold | Точечный график. Показывает вероятность образования пар оснований, RNAfold |
 |
 |
Задание 2.
Поиск ДНК-белковых контактов в заданной структуре
Упражнение 1.
Задание множества атомов с помощью команды define в программе JMol.
~ Множество атомов кислорода 2'-дезоксирибозы (o_in_dr). ~ Множество атомов кислорода в остатке фосфорной кислоты (o_in_ph).
~ Определите множество атомов азота в азотистых основаниях (n_in_base).
~ Cкрипт-файл, вызов которого в JMol даст последовательное (с паузами) изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов o_in_dr, затем o_in_ph и n_in_base.
Для загрузки апплета нажмите на изображение. Для запуска скрипта используйте кнопку Start script, для продолжения работы скрипта - Resume.
Упражнение 2.
Описание ДНК-белковых контактов в заданной структуре. Сравнение количества контактов разной природы.
Будем считать полярными атомы кислорода и азота, а неполярными - атомы углерода, фосфора и серы. Назовем полярным контактом ситуацию, в которой расстояние между
полярным атомом белка и полярным атомом ДНК меньше 3.5Å. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Å.
| Контакты разного типа в комплексе 1tro.pdb | |||
|---|---|---|---|
| Контакты атомов белка с: | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 0 | 57 | 57 |
| остатками фосфорной кислоты | 54 | 48 | 102 |
| остатками азотистых оснований со стороны большой бороздки | 11 | 44 | 55 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 4 | 4 |
Исследование ДНК-белковых контактов показало:
- Отсутствие полярных контактов белка с остатками дезоксирибозы. Возможное объяснение: трудная стерическая доступность O-атомов 2'-дезоксирибозы, повернутых внутрь двойной спирали.
- Большое количество неполярных контактов незаряженных аминоксилот (Q, N, S, T, G, A) с остатками декзоксирибозы.
- Наибольшее количество контактов обнаружено между белком и остатками фосфорной кислоты. К ним фотянутся положительно заряженные аминокислоты - K и R, в целом, фосфаты лучше всего контактируют с разнообразными аминокислотами, так хорошо доступны.
- Что интересно, полярные контакты большой бородки ДНК реализуются только за счет аргинина (R), потому как данная аминоксилота положительно заряжена и достаточно крупная.
- Многочисленные неполярные контакты азотистых оснований большой бороздки с аминокислотами.
- В малой бороздке, куда доступ молекул сильно ограничен удалось обнаружить 4 неполярных контакта с глицином (G). Из-за отсутствия заряда и маленького размера он единственный способен преодолеть пространственные преграды.
Упражнение 3.
Получение. популярной схемы ДНК-белковых контактов с помощью программы nucplot.
| ДНК-белковые контакты в комплексе 1TRO на схеме, полученной командой nucplot |
|---|
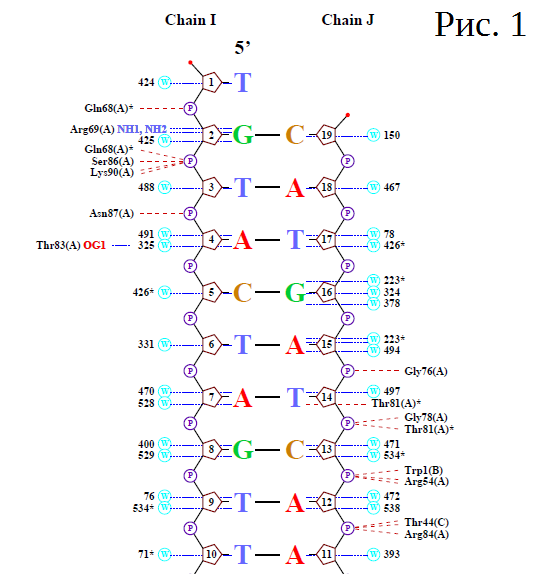 |
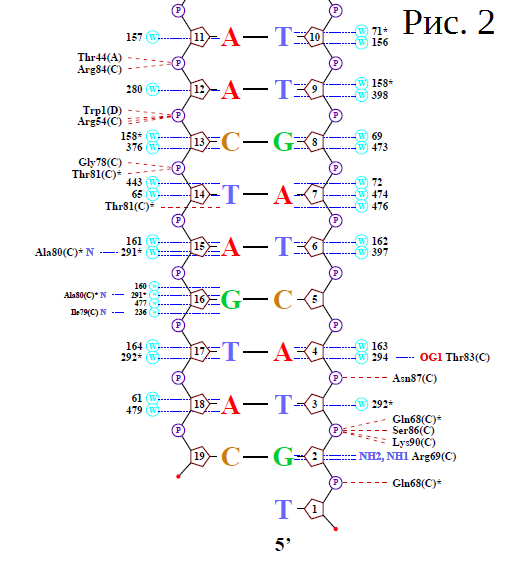 |
 Условные обозначения: пятиугольник - сахар остова с номером основания; P в кружке - фосфатная группа * - дополнительные контакты; синий штрих - водородные связи красный штрих - интересующие нас контакты; W - вода |
Программа nucplot результатом выдачи имеет популярную схему комплекса ДНК-белок в .ps формате, которая легко переводится в изображение. Она работает только со старым форматов .pdb, поэтому нужно конвертировать файл в программе remediator. К сожалению, при работе с nuplot возникла ошибка сегментации, которую я не смогла устранить. Я провела поиск нужной мне схемы в нескольких базах данных и в итоге нашла необходимое изображение в базе PDIdb.
Упражнение 4.
Из представленной схемы следует, что:
- Аминокислотные остатки с наибольшим числом указанных на схеме контактов с ДНК - Arg54(C) (3 контакта с одним фосфатом) и Arg54(A), Thr44(C), Lys90(C), Arg54(A) - у всех по 2 контакта с остатком фосфорной кислоты. (A)
- Белок 1TRO является репрессором trp, т.е. регулирует транскрипцию триптофана. Один из генов, регулируемых репрессором Trp, TRpr, кодирует
сам белок триптофанового репрессора. Это является одной из форм регулирования с обратной связью. [2] Его структура имеет обширую контактную
поверхность, образуя водородные связи и солевые мостики с ДНК. Непосредственных H-связей или неполярных контактов с основаниями, которые могли бы объяснить специфичность
репрессора к последовательности оператора, нет. Вероятнее всего, последовательность оператора распознается косвенным путем, благодаря эффектам, возникающим из геометрии
фосфатного остова, которые формируют стабильный интерфейс. Гидратационные полярные контакты с основаниями тоже, по предположению, влияют на специфичность.
[3]Как мы можем наблюдать на схеме, в комплексе на каждое азотистое основание (за исключением 4) приходится от одной до четырех связей с
молекулами воды.
Очевидно, что для распознавания последовательности ДНК наиболее важны аминокислотные остатки, специфически контактирующие непосредственно с остатками азотистых оснований. Не совсем очевидно, почему на схеме nucplot не указаны многочисленные неполярные контакты, рассмотренные мной в ходе работы с Упр.2. На популярной схеме, где не отображены непосредственные контакты аминокислотные остатков с основаниями, можно наблюдать связывание Thr81(A) и Thr81(С) с фосфатными остатками, что проиллюстировано на изображении ниже (показаны только группы в пределах региона контакта).Полярный ДНК-белковый контакт (Thr81(A) и фосфат, изображение получено с помощью JMol) 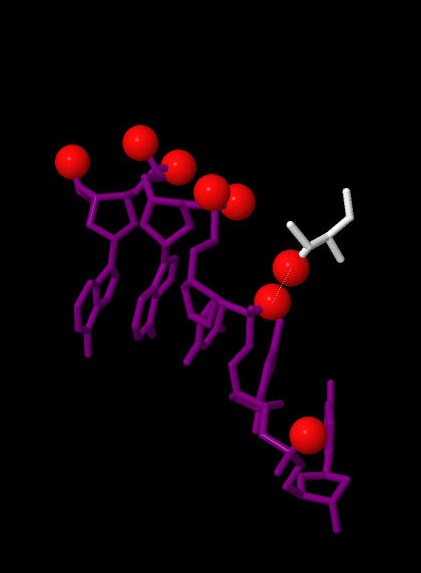
Источники
- [1] О. О. Фаворова, Строение транспортных РНК и их функция на первом (предрибосомном) этапе биосинтеза белков, Соровский образовательный журнал, №11 (1998): 71-77, рис. 2
- [2] Wikipedia: Tryptophan repressor
- [3] Pudmed: Crystal structure of trp repressor/operator complex at atomic resolution, Nature (1988).