Чтение последовательностей по Сэнгеру.
Задание 1. Прочтение последовательности ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру. Отчёт о проблемах при чтении хроматограмм.
Капиллярный секвенатор по Сангеру выдает файлы с хроматограммой и автоматически прочтенной последовательностью в
формате .ab1. Мне было дано два файла .ab1 (прямая и обратная
цепи), соответствующие прочтению прямой и обратной цепочки секвенируемой ДНК. Для просмотра хроматограмм и редактирования автоматического прочтения использована
программа Chromas (Lite).
- Список используемых терминов:
- Проблемный нуклеотид - тот, по которому Вы приняли решение, отличное от предложенного программой, или согласились с программой, но необходимо было проанализировать хроматограммы для принятия окончательного решения. Проблемные нуклеотиды в последовательности выделены строчными буквами.
- Полиморфизм - нуклеотид, про который Вы решили, что в секвенируемой ДНК встречаются два (или более) варианта. Полиморфизмы обозначены кодами вырожденных нуклеотидов (ambiguity codes) - W: A или T; S: G или C; и т.д.[1]
| Общая характеристика хроматограммы | ||
|---|---|---|
| Параметр | Прямая цепь | Обратная цепь |
| Начальный нечитаемый участок (5'-конец) | 1-35 (35 нуклеотидов) | 1-6 (6 нуклеотидов) |
| Конечный нечитаемый участок (3'-конец) | 704-717 (14 нуклеотидов) | (после замены на
комплементарную!) 688-718 (31 нуклеотид) |
| Примерное отношение уровня сигнала к шуму | ~1300/~160 (1/8) | ~1400/~160 (1/9) |
| Неравномерность силы сигнала и шума вдоль последовательности | Сигнал относительно равномерный | Аналогично |
| Другие особенности | Начиная с 470 номера очень часто идут повторяющиеся нуклеотиды, что вызывает образоване слившихся двойных, тройных и большей кратности пиков. | |
В cледующей таблице показаны 4 участка, содержащие проблемные нуклеотиды. Верхнее изображение из пары показывает прямую цепь, а нижнее - комплементарную обратной. После удаления нечитаемых участков 1-ой позиции прямой цепочки соответствует 68-ая комплементарной обратной.
| Анализ проблемных участков | |||
|---|---|---|---|
| Иллюстрация | Фактор, вызывающий проблему | Описание | |
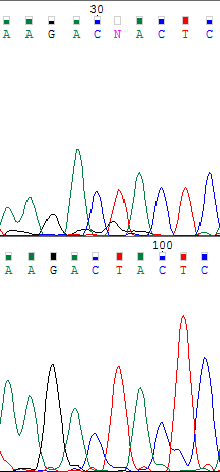 | Шум, затрудняющий идентицикацию нуклеотида | Из-за высокого уровня шума в хроматограмме прямой цепи не был распознан 31-ый нуклеотид - тимин, что можно восстановить по данным второй хроматограммы. В .fasta-файле на данной позиции проставлена t. | |
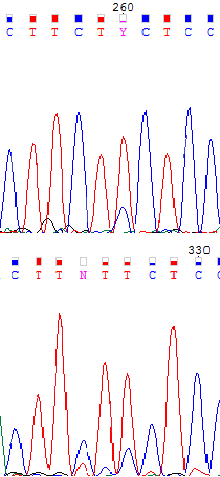 | Полиморфизм | В позиции 325 обратной цепи не может быть автоматически определен нуклеотид из-за примерно равного соотношения сигнал-шум, что, по моему мнению, может быть объяснено возможным полиморфизмом данного участка, так как на соответствующей 258-ой позиции прямой цепи стоит цитозин, а красный цвет накладывающегося пика на нижнем изображении говорит о возможном присутствии в цепи тимина. В .fasta была произведена соотвествующая замена на y. Кроме того, на 260-ом месте прямой цепи указан вырожденный кодY (обозначающий C или T), что также доказывает моё предположение. Соотвественно ему под 327-ым номером стоит тимин в обратной цепи. | |
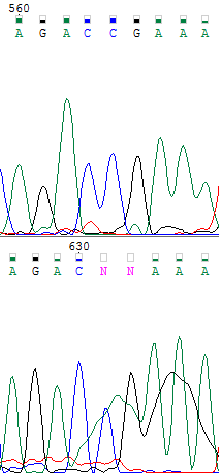 | Перекрывание размытых пиков | Нуклеотиды в позициях 631 и 632 обратной цепи не были распознаны. Сравнивая нижнее изображение с верхним, можно понять, что ошибки программы возникают из-за перекрывания размытых пиков гуанина и аденина, которые накладываются на сигнал цитозина, воспринимаемый в данном случае как шум. По данным последовательности прямой цепи можно установить, что 631-ому нуклеотиду снизу соответствует 564-ый цитозин сверху, а 632-ому - 565-ый гуанин. Соответствующие изменения внесены в .fasta-файл обратной цепи. | |
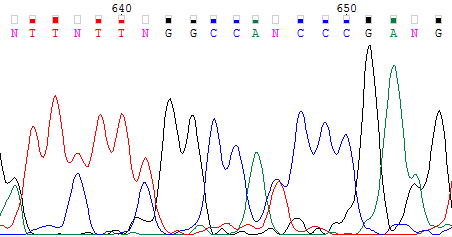 | Падение качества в конце хроматограммы | На данном изображении представлен концевой участок прямой цепи, где мы можем наблюдать множественные размытые пики, перекрывания и связанные с этим проблемы определения нуклеотидов (много N). | |
Обобщая вышесказанное, можно прийти к выводу, что добиться высокой точности при секвенировании позволяет параллельный анализ данных, полученных программой, по
прямой и комплементарной обратной последовательностям. Предложенные мне для изучения хроматограммы я бы оценила как достаточно качественные.
Далее приведены ссылки на материалы практикума. Прошу обратить Ваше внимание на то, что помимо указанных в таблице изменений в .fasta-файлах были удалены концы плохого
качества, как в последнем примере.
Задание 2. Пример не читаемого фрагмента хроматограммы.
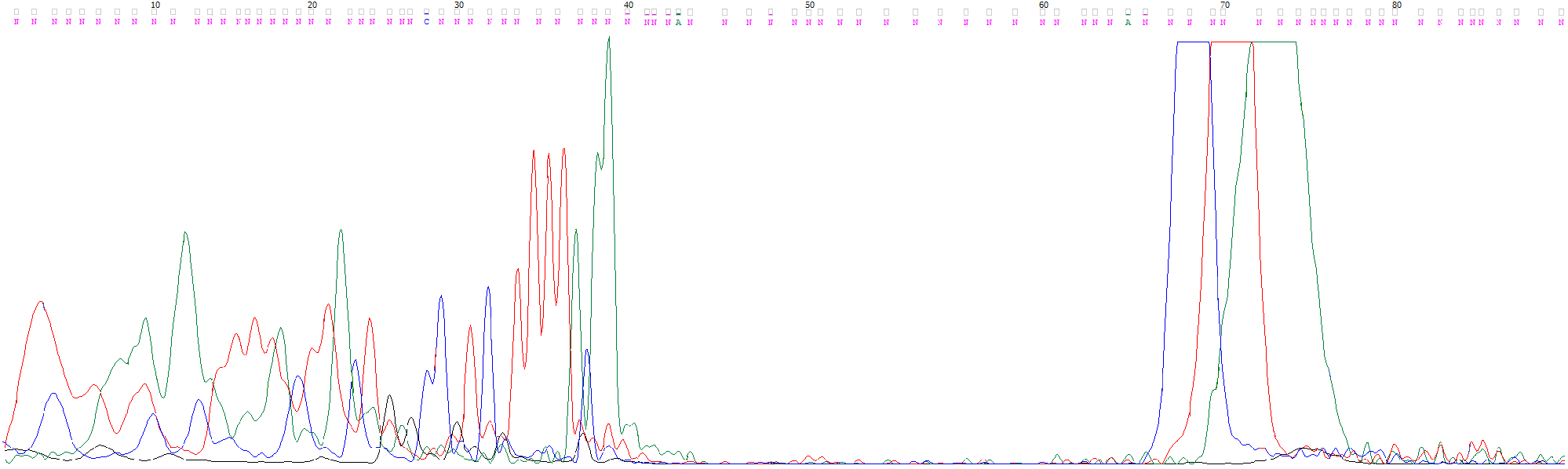
С 1-го по 40-ой нуклеотиды мы можем наблюдать на хроматограмме многочисленные перекрывания размытых пиков, сложным образом соединённые несколько пиков. Похожая картина наблюдается при полиморфизме, вызванном делецией нуклеотида, однако в таком случае пики идут равномерно и более схожи по высоте. Вероятно, причиной данного "узора" могут быть различные загрязнения, например, содержание примесных солей. После 40-го нуклеотида наблюдается резкий спад амплитуды пиков, который продолжается вплоть до 67-ого номера, что свидетельствует о наличии сложной структуры ДНК. В позициях 67-74 можно увидеть пятно красок, полученное наложением сразу трёх пиков.