Предсказание генов прокариот.
Задание 1. Сравнение предсказаний генов в базе данных GenBank и по данным Prodigal для плазмиды.
1. Подготовка файлов для анализа.
В этом задании с помощью EMBOSS был получен файл выданной мне плазмиды (CP014286) в формате gff. Из него была извлечена информация о координатах последовательностей, кодирующих белки (начало, конец и ориентация соотвестственно). Ниже приведены соответствующие команды и ссылки на полученные файлы.seqret embl:CP014286 CP014286.fasta
seqret embl:CP014286 -feature CP014286.gff
grep CDS CP014286.gff | awk '{print $4 " " $5 " " $7}' > CP014286_feat
Благодаря первой команде из базы данных EMBL была сохранена нужная мне последовательность в fasta-формате. Cледующая команда, используя квалификатор -feature,
перезаписала файл в табличный gff-формат особенностей. Далее командой grep были найдены все паттерны, содержащие "CDS", перенаправлены утилите awk для
извлечения данных столбцов с номерами 4, 5 и 7, где содержатся координаты. [1] Результаты направлены в файл CP014286_feat.| Указанный идентификатор относится к последовательности Bacillus thuringiensis strain
Bt185 plasmid pBT1850054 размером 54205 п.о. Имеет 72 гена, 71 из которых белок-кодирующий, 1 - псевдоген, определяемый как нефункциональная копия генетического
фрагмента, включенного в геном путем ретротранспозиции мРНК или дупликации геномной ДНК. [2] Bacillus thuringiensis (Bt) - грамположительная почвенная бактерия, зачастую используется в качестве биопестицида. Во время споруляции многие штаммы образуют кристализующиеся белковые включения, называемые δ-эндотоксинами, имеющими инсектицидное действие. [3]  Bacillus thuringiensis, покраска по Граму, увеличение 1000 X 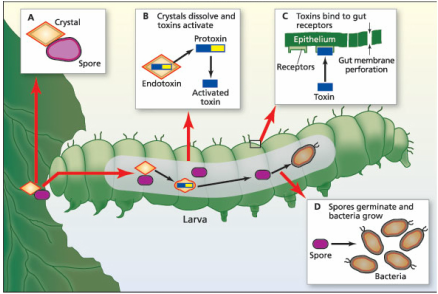 Показан процесс инфицирования гусеницы, которая съедает спору Bt. [4]
|
2. Предсказание генов с помощью Prodigal.
| Prodigal - программное обеспечение для предсказания генов, кодирующих белки
в геномах бактерий и архей. Работает из командной строки. Акроним был получен в результате слияния PROkaryotic DYnamic Programming Genefinding ALgorithm. Что может делать программа?
Normal Mode, в котором Prodigal изучает поданную на вход последовательность, узнает ее характеристики и делает на их основе предсказание. Используется для изучения полных геномов, геномных проектов хорошего качества и больших вирусах. Anonymous Mode, в котором Prodigal использует заранее сделанные калькуляции для исследуемой последовательности и предсказывает гены, основываясь на лучших результатах. Используется для изучения метагеномов, геномных проектов низкого качества, маленьких вирусах и плазмидах. Training Mode, принцип работы которого совпадает с нормальным режимом, но кроме этого сохраняются тестовые файлы для дальнейшего использования. |
prodigal.windows.exe -c -i CP014286.fasta -o CP014286_prodigal prodigal.windows.exe -c -i CP014286.fasta -o CP014286.pro -f scoПосле установки на ПК данная программа была запущена с представленными выше параметрами, а именно: -i (input_file) - последовательность на входе, -o (output_file) - последовательность на выходе. При запуске программы без квалификатора -c Prodigal начал предсказывать гены с 3-го основания, что противоречит и здравому смыслу, и данным аннотации GenBank. Квалификатор -s записывает все потенциально посчитанные гены вместе со scores, с помощью квалификатора -f был получен итоговый файл CP014286.pro в рекоммендуемом минималистичном формате .sco.
Для получения отформатированного файла CP014286.pro_feat, содержащего только координаты генов, был использован скрипт changeform1.py.
3. Сравнение и результаты.
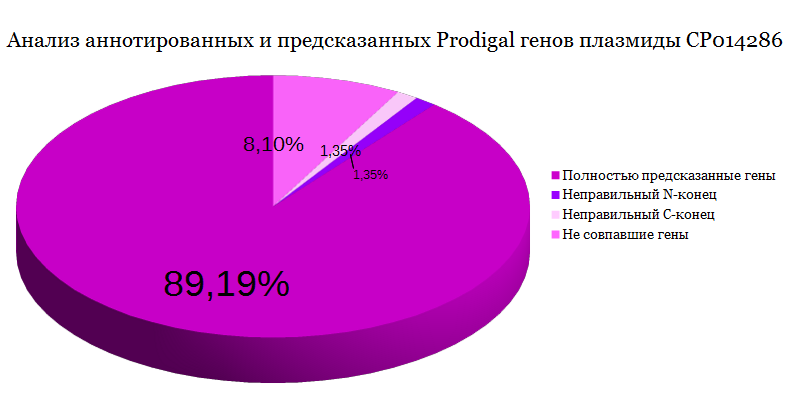
Для сравнения двух файлов (CP014286_feat и CP014286.pro_feat) был написан скрипт в Python comparison1.py. Подробное описание работы программы закомментировано, поэтому здесь лишь опишу основную идею: путем создания списков и сравнения в них элементов-координат (например, положительная ориентация означает, что координата N-конца - первая, в противном случае - вторая), удалось однозначно установить соответствие между списками генов, выявить гены с одним несовпадающим концом и вообще не аннотированные гены.| Результаты | |
|---|---|
| Число аннотированных в Genbank генов | 74 |
| Число предсказанных Prodigal генов | 77 |
| Число генов с двумя совпадающими концами | 66 |
| Процент генов, совпадающих обоими концами | 89,19% |
| Число генов с неправильно определенным N-концом | 1 |
| Процент генов с неправильно определенным N-концом | 1,35% |
| Число генов с неправильно определенным C-концом | 1 |
| Процент генов с неправильно определенным C-концом | 1,35% |
| Число не совпавших генов | 6 |
| Процент не совпавших генов | 8,1% |
 | |
4. Причины несовпадения.
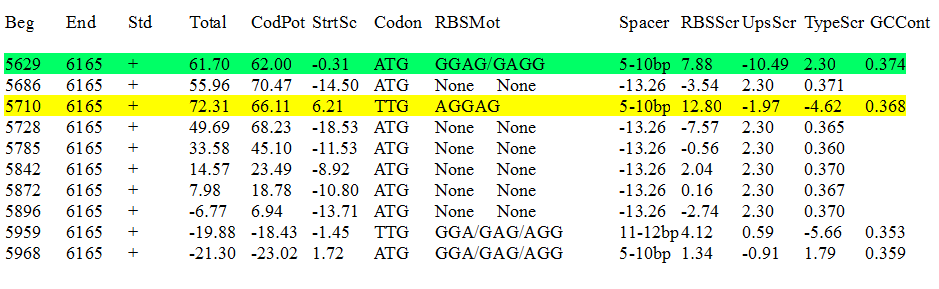
Вообще говоря, предсказания Prodigal для плазмиды CP014286 оказались достаточно хорошими (почти 90% верно определенных генов). Как в случае с C-, так и с N-концами неправильно предсказанных "на половину" генов оказалось по одному.¡К сожалению, при перепроверке вручную я обнаружила, что программа неправильно посчитала количество не совпавших N- и C-концов (по данным моей программы их по одному в каждом случае, но по моим подсчетам для С-концов ошибок не было вообще, для N-концов по результатам Prodigal не совпало 5 генов). Однако установить причину ошибки и найти логическую ошибку в скрипте мне не удалось. Буду очень признательна, если Вы поможете мне выяснить причину такого расхождения. Соотвественно, диаграмму и таблицу в предыдущем задании нельзя считать достоверными!В качестве такого примера для N-конца выступает ген AXW78_29345, расположенный на позициях 5629 - 6165, кодирующий ДНК-связывющий белок AMR06410. Prodigal предсказал его так: 5710 6165 +, с ошибкой на 21 нуклеотид. Причиной ошибки может быть определение программой в качестве стартового кодона триплета TTG (в отличие от аннотированного ATG). Оба эти варианта по данным трансляционной таблицы 11 (The Bacterial, Archaeal and Plant Plastid Code) могут являться стартовыми у бактерий. Если смотреть подробное описание со scores, то разница между предложенным алгоритмом вариантом (72.31) и реальным геном (61.70) составляет 10.61 балла.
Ген AXW78_29345 в последовательности Genbank
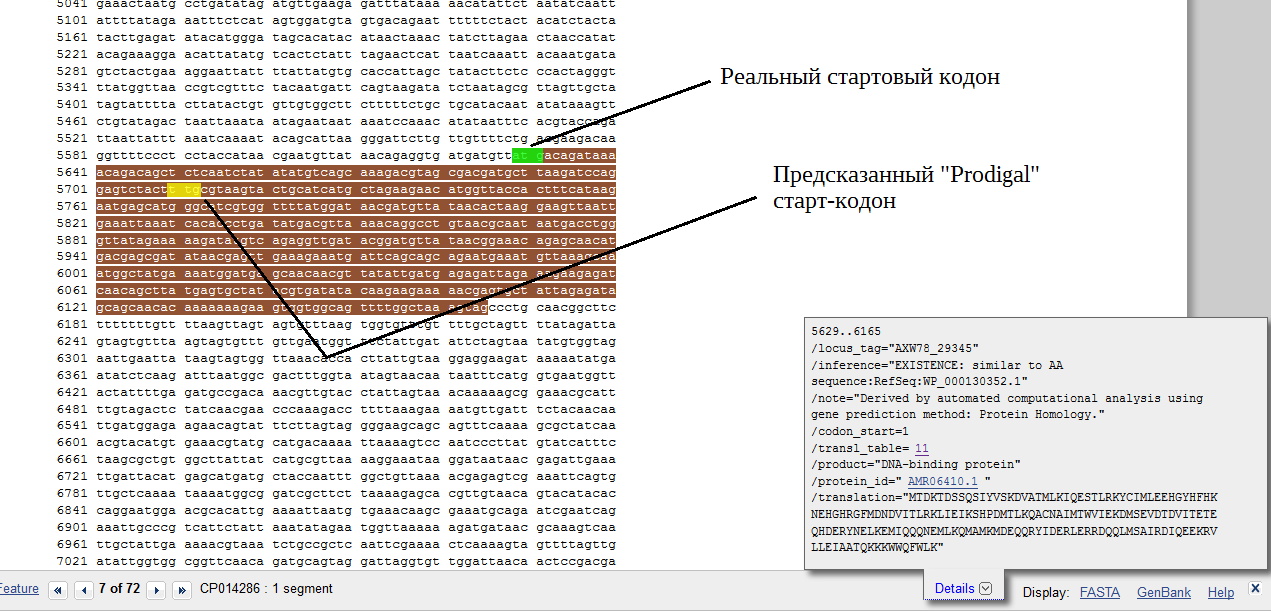
Результаты работы программы (из файла prodigal_potscores)
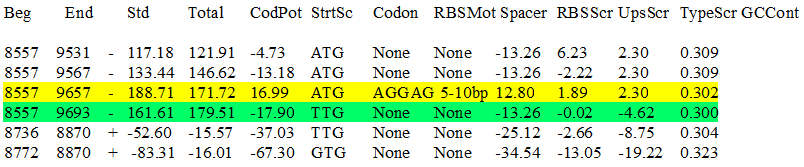
Другим примером несоответствия N-конца является рассчет для гена AXW78_29370, расположенного на позициях 8557 - 9693 (-), кодирующего аспартат-фосфатазу. Предсказание Prodigal оказалось короче - всего с 8557 по 9657 (-). По описанию, качественные баллы предсказанных и аннотированных координат различаются на почти 17 баллов. Но в данном случае, наоборот, программа посчитала наиболее вероятным стартовым кодоном тривиальный вариант ATG для метионина, нежели описанный в Genbank TTG.
Результаты работы программы (из файла prodigal_potscores)