Предсказание генов эукариот.
Задание 1. Описание выданного контига и одного из генов на нем, для которого предсказан альтернативный сплайсинг.
В данном практикуме был проанализирован контиг NW_004797386, принадлежащий морскому моллюску Aplysia californica. Его длина составляет 1527627 п.о., число кодирующих последовательностей при общем количестве генов (35) - 40.
Eukaryota Opisthokonta Metazoa Eumetazoa Bilateria Protostomia Lophotrochozoa Mollusca Gastropoda Heterobranchia Euthyneura Euopisthobranchia Aplysiomorpha Aplysioidea Aplysiidae Aplysia Aplysia californica - крупный заднежаберный моллюск, обитающий на тихоокеанском побережье Северной Америки и Мексики. Способен достигать 40 см в длине и весить до 2.3 кг, однако, большинство имеет средние размеры около 20 см длиной и весом 1 кг. Имеется защитная окраска, которая меняется от красновато-коричневого до каре-зеленого цвета. Она во многом зависит от типа водорослей, используемых в рационе (красные водоросли, ульва, зостера). Тело мясистое, на голове распологаются 2 пары щупалец: одна из них прямо за глазами, а вторая немного выше ротового отверстия. Из-за необычного внешнего вида его так же называют морским зайцем. На дорсальной стороне располагаются 2 крылоподобные складки. Гермафродит. Нерестится в летнее время в глубоких частях ареала. [1]  Aplysia californica выбрасывает чернильную жидкость. [2]
|
В качестве гена с предсказанным в NCBI альтернативным сплайсингом был выбран ген LOC101860970.
- Координаты: 624861..635454 complement
- Длина: 10594
- Число экзонов: 5
- Описание: trithorax group protein osa-like (кодирует osa-подобный белок группы Thrithorax)
Геномное окружение гена LOC101860970
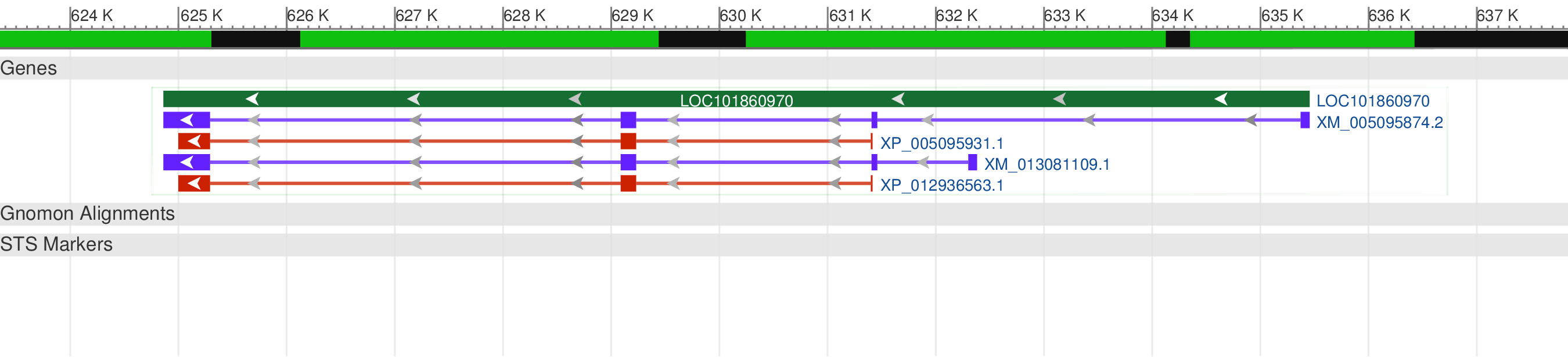
Один из вариантов транскрибируемых мРНК (transcript variant X1) - XM_005095874
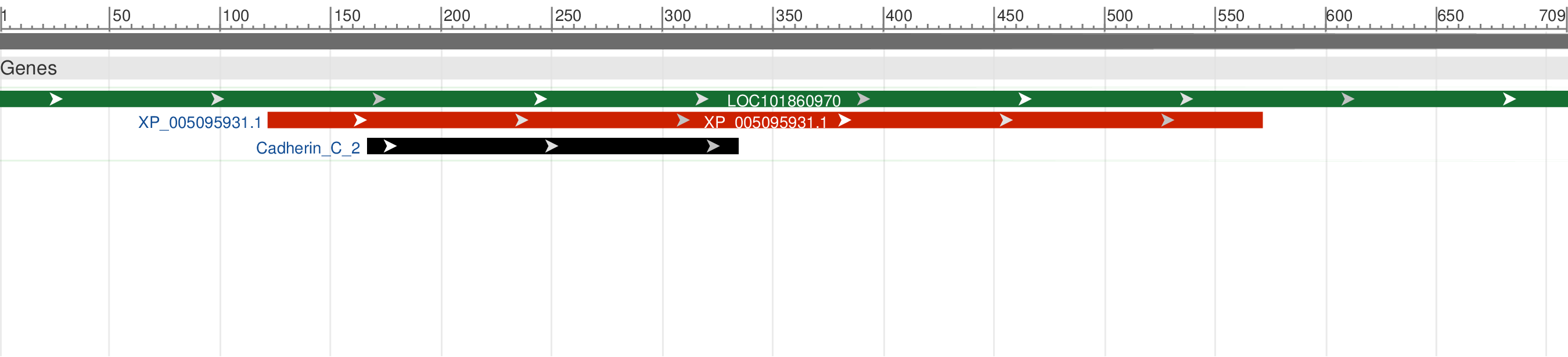
Соответсвующая мРНК транслированная изоформа белка группы Thrithorax - XP_005095931

Как показано на верхнем изображении, для данного гена возможны 2 альтернативных изоформы белка (красные), соответствующие 2 вариантам мРНК (синие). Из особенностей представленной на 3-ем рисунке изоформы osa можно выделить консервативный домен Cadherin_C_2, который является цитоплазматическим C-концевым доменом некоторых прото-кадгеринов. Указанный класс поверхностных рецепторов отвечает за клеточную адгезию и морфогенез в многоклеточных организмах.
Белки Thrithorax (TrxG) - гетерогенное семейство белков с основной функцией поддержания генетической экспрессии. Их можно поделить на 3 группы по механизму работы:
- гистон-модифицирующие
- хроматин-ремодулирующие
- ДНК-связывающие
Задание 2. Предсказание генов и белок-кодирующих областей в выданном контиге.
Выполнено с помощью сервера AUGUSTUS в режиме Prediction. Для сравнения необходимо было использовать в качестве модели наиболее близкий к изучаемому организм. Выбор оказался довольно сложным, так как не было представлено организмов из типаMollusca и надтипа Lophotrochozoa, поэтому я выбрала нематоду Caenorhabditis elegans (по современной классификации менее таксономически далекого от изучаемого объекта организма). Предсказание UTR для нетранслируемых областей делает предсказание более точным, однако эту опцию следует использовать при гарантированном соответствии файла с параметрами UTR исследуемому Вами организму, поэтому было оставлено дефолтное значение False. Значение few при подсчете альтернативных транскриптов было выбрано исходя из наличия небольшого количества транслируемых изоформ в изучаемом объекте (по умолчанию указывается none). Все оставшиеся параметры изменены не были.
Details of your job: Prediction job ID: predZauqe2ZH AUGUSTUS parameter project identifier: caenorhabditis Genome file: NW_004797386.fasta User set UTR prediction: false Report genes on: both strands Alternative transcripts: few Allowed gene structure: predict any number of (possibly partial) genes Ignore conflictes with other strand: false
Результатом работы программы стал архив tar.gz со следующими файлами:
| Формат | Описание |
| .aa | предсказание генов в формате fasta-последовательности белков |
| .cdsexons | предсказание экзонов в формате fasta-последовательности ДНК |
| .codingseq | предсказание генов в формате fasta-последовательности CDS ДНК |
| .gbrowse | трек-файл предсказания для GBrowse |
| .gff | предсказание генов в табличном формате gff (General Feature Format) |
| .gtf | предсказание генов в табличном формате gft (аналогичен gff) |
Предсказание AUGUSTUS, на мой взгляд, плохо совпадает с аннотацией в GenBank. Так, при наличии 35 аннотированных генов в базе программа предсказывает 129 таковых по данным таблицы .gff. Много вариантов предсказаний, в аннотации вообще не описанных. Что касается гена LOC101860970, то предсказание для него, по-моему, не особо удачное. Вместо него на близких позициях 624999 - 634845 предсказаны g44 и g45. Окружение гена предсказано с ошибками, начало и конец не совпадают. Возможно, причиной не очень качественного предсказания является выбор модельного объекта, слишком далекого по таксономии от изучаемого организма.