Evolutionary trees.
Задание 1. Укоренение в среднюю точку.
С помощью функции M программы retree пакета PHYLIP
дерево енолаз было переукоренено в среднюю точку. На выходе был получен файл "outtree", переименнованный в "ENO_after_retree_tree.tre". Ниже представлены соответствующие изображения
из MEGA.
Переукорененное дерево с указанием длины ветвей |
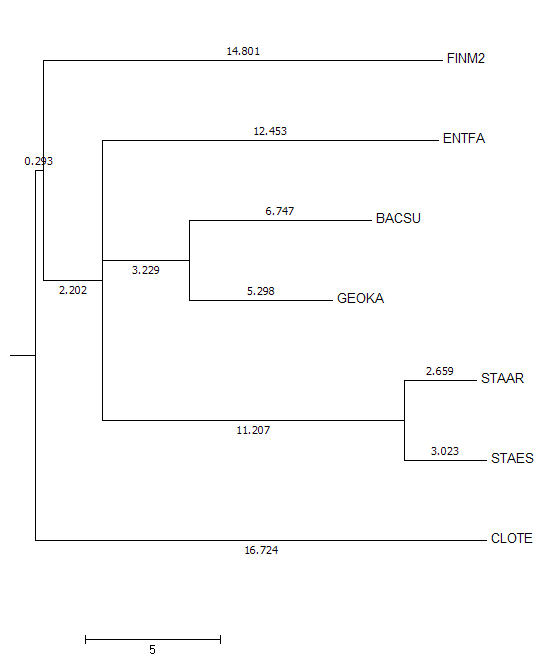 |
Переукорененное дерево с отражением только топологии |
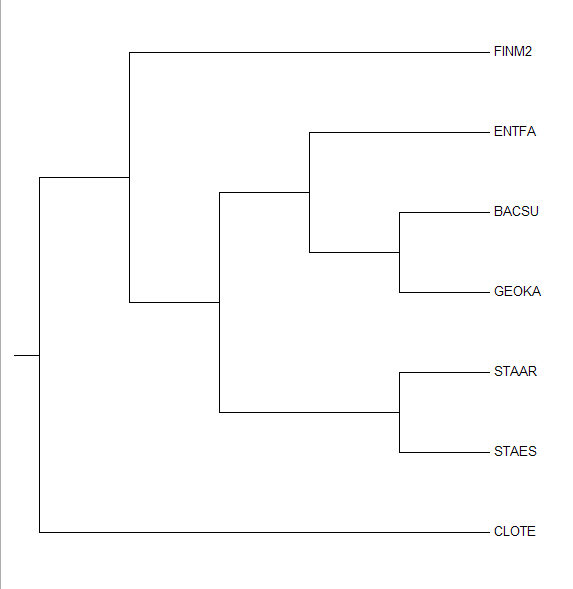 |
Следует отметить, что при построении деревьев (как до, так и после переукоренения) с учетом длин ветвей получаются небинарные деревья, чего можно
избежать при построении с учетом лишь топологии. Результаты укоренения в среднюю точку совпали с результатами прошлого практикума, а именно укоронение произошло в тривиальную
ветвь {CLOTE} vs. {FINM2, ENTFA, GEOKA, BASCU, STAAR, STAES}. Вряд ли данную гипотезу можно принять как верную, потому как максимальную длину в построении до переукоренения имеет ветвь с листом FINM2 (14.801), указанная же ветвь,
с листом CLOTE, имеет максимальную длину в построении после переукоренения (16.724). А в эталонном дереве укоренение вообще уходит в нетривиальную ветвь {CLOTE, FINM2}
vs. {ENTFA, GEOKA, BASCU, STAAR, STAES}. Что касается топологии, то она совпадает с таковой у дерева, полученного методом NJ.
Задание 2. Использование внешней группы.
В методе максимальной экономии (Maximum Parsimony, MP) не учитываются длины ветвей, а значит, невозможно найти самый длинный путь,
чтобы переукоренить дерево в его среднюю точку. Для этого используют внешнюю (аут-) группу - кластер неблизкородственных организмов. Укоренение дерева белков ENO
выбранных бактерий производилось с использованием в качестве аутгруппы енолазы Escherichia coli. По аналогии с предыдущим практикумом, было построено
выравнивание указанных ранее последовательностей с добавленной последовательностью E. coli при тех же параметрах.
Дерево, построенное с использованием аутгруппы
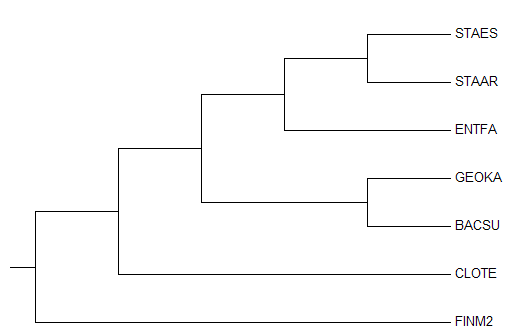
По выравниванию в MEGA было реконструировано филогенетическое дерево методом MP. Корневой была выбрана ветвь, ведущая к ECOLI.
С помощью функции "Show Subtree Separately" было получено нужное дерево без E. coli.
При сравнении этого дерева с эталонным не обнаруживаются следующие нетривиальные ветви: {CLOTE, FINM2} vs. {ENTFA, STAAR, STAES, GEOKA, BASCU}; {CLOTE, FINM2, ENTFA} vs. {STAAR, STAES, GEOKA, BASCU}, а бактерии ENTFA и (STAAR, STAES) объединены в общую кладу, которой нет ни в основном дереве, ни в деревьях, полученных методами NJ или переукоренением в среднюю точку, в двух последних, однако, встречается клада (ENTFA, (BACSU, GEOKA)). Вероятно, недостаточно судить о взаимосвях между выбранными организмами, основываясь только на анализе данного белка, потому как клада близкородственных енолах ((STAAR, STAES), (BACSU, GEOKA)) в различных алгоритмах по-своему разбивается в разрешенных деревьях. В таком случае приоритетно использование небинарных деревьев.
При сравнении этого дерева с эталонным не обнаруживаются следующие нетривиальные ветви: {CLOTE, FINM2} vs. {ENTFA, STAAR, STAES, GEOKA, BASCU}; {CLOTE, FINM2, ENTFA} vs. {STAAR, STAES, GEOKA, BASCU}, а бактерии ENTFA и (STAAR, STAES) объединены в общую кладу, которой нет ни в основном дереве, ни в деревьях, полученных методами NJ или переукоренением в среднюю точку, в двух последних, однако, встречается клада (ENTFA, (BACSU, GEOKA)). Вероятно, недостаточно судить о взаимосвях между выбранными организмами, основываясь только на анализе данного белка, потому как клада близкородственных енолах ((STAAR, STAES), (BACSU, GEOKA)) в различных алгоритмах по-своему разбивается в разрешенных деревьях. В таком случае приоритетно использование небинарных деревьев.
Задание 3. Bootstrap.
Bootstrap (дословно - петля на заднике ботинка) - практический компьютерный метод исследования распределения статистик, основанный на многократной
генерации выборок методом Монте-Карло на базе имеющейся выборки. [1]
При построении дерева из исходного выравнивания создается множество «бутстрэп-реплик». В каждой реплике случайно выбранная половина столбцов заменяется на копии так же случайно выбранных других столбцов, для всех реплик строится дерево, а затем на базе "индивидуальных" деревьев строится общее методом расширенного большинства ("Extended majority-rule tree"). К дереву, построенному из ветвей, встретившихся в большинстве "индивидуальных" добавляются ветви, не противоречащие уже имеющимся, начиная с наиболее "поддержанных". Числа на ветвях обозначают количество «бутстрэп-реплик», в которых имеется та или иная ветвь.
Бутстрэп-анализ филогении белков был проведен в MEGA с использованием метода NJ. Число реплик равно 100.
При построении дерева из исходного выравнивания создается множество «бутстрэп-реплик». В каждой реплике случайно выбранная половина столбцов заменяется на копии так же случайно выбранных других столбцов, для всех реплик строится дерево, а затем на базе "индивидуальных" деревьев строится общее методом расширенного большинства ("Extended majority-rule tree"). К дереву, построенному из ветвей, встретившихся в большинстве "индивидуальных" добавляются ветви, не противоречащие уже имеющимся, начиная с наиболее "поддержанных". Числа на ветвях обозначают количество «бутстрэп-реплик», в которых имеется та или иная ветвь.
Бутстрэп-анализ филогении белков был проведен в MEGA с использованием метода NJ. Число реплик равно 100.
Original tree |
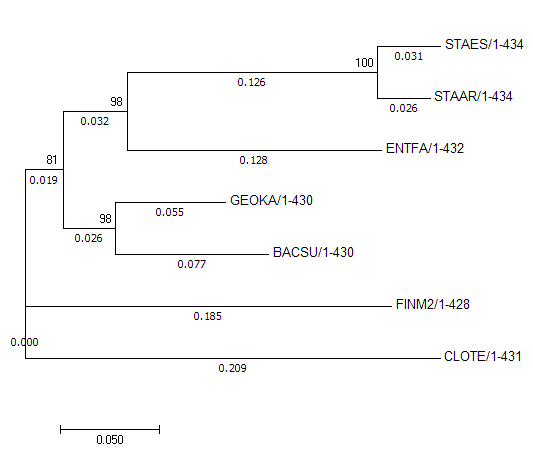 |
Consenus tree |
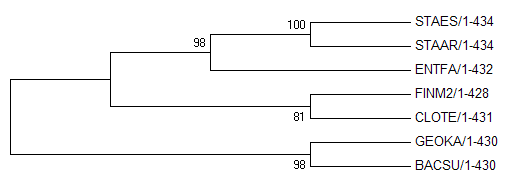 |
Топология оригинального дерева:
1) {CLOTE, FINM2} vs. {ENTFA, STAAR, STAES, GEOKA, BASCU}
2) {CLOTE, FINM2, GEOKA, BACSU} vs. {ENTFA, STAAR, STAES}
3) {CLOTE, FINM2, GEOKA, BACSU, ENTFA} vs. {STAAR, STAES}
Топология консенсусного дерева:
1) {GEOKA, BACSU} vs. {FINM2, CLOTE, ENTFA, STAAR, STAES}
2) {GEOKA, BACSU, FINM2, CLOTE} vs. {ENTFA, STAAR, STAES}
3) {CLOTE, FINM2, GEOKA, BACSU, ENTFA} vs. {STAAR, STAES}
Таким образом, топология консесусного и оригинального деревьев различается (одна общая нетривиальная ветвь под номером 3). Несмотря на то, что укоренение в консенсусном дереве прошло в ранее нигде не встречавшуюся ветвь {GEOKA, BACSU} vs. {FINM2, CLOTE, ENTFA, STAAR, STAES} с поддержкой большей, чем у ветви, в которую укоренено original tree и само эталонное (98 реплик против 81 одной реплики), я считаю данную ветвь и укоренение ложными. При этом, ни одно из деревьев не совпадает с эталонным.
*И вообще, все-все деревья хоть одной ветвью, да отличаются, ПРИЧИНА? (Подумать)
1) {CLOTE, FINM2} vs. {ENTFA, STAAR, STAES, GEOKA, BASCU}
2) {CLOTE, FINM2, GEOKA, BACSU} vs. {ENTFA, STAAR, STAES}
3) {CLOTE, FINM2, GEOKA, BACSU, ENTFA} vs. {STAAR, STAES}
Топология консенсусного дерева:
1) {GEOKA, BACSU} vs. {FINM2, CLOTE, ENTFA, STAAR, STAES}
2) {GEOKA, BACSU, FINM2, CLOTE} vs. {ENTFA, STAAR, STAES}
3) {CLOTE, FINM2, GEOKA, BACSU, ENTFA} vs. {STAAR, STAES}
Таким образом, топология консесусного и оригинального деревьев различается (одна общая нетривиальная ветвь под номером 3). Несмотря на то, что укоренение в консенсусном дереве прошло в ранее нигде не встречавшуюся ветвь {GEOKA, BACSU} vs. {FINM2, CLOTE, ENTFA, STAAR, STAES} с поддержкой большей, чем у ветви, в которую укоренено original tree и само эталонное (98 реплик против 81 одной реплики), я считаю данную ветвь и укоренение ложными. При этом, ни одно из деревьев не совпадает с эталонным.
*И вообще, все-все деревья хоть одной ветвью, да отличаются, ПРИЧИНА? (Подумать)