Геномное окружение. База данных GO.
Задание 1. Получение информации о COG`е, к которому относится белок.
Для выполнения практикума был использован белок фенилацетат-коэнзим A лигаза из организма Bacteroides thetaiotaomicron VPI-5482
(с идентификатором NP_809341.1), выданный для работы в 1 семестре. Его длина составляет 435 аминокислотных
остатков. С помощью сервиса CDD (Conserved Domain Database) для моего белка был получен список
хитов, откуда были выбраны хиты, относящие его к тому или иному КОГу.
Список хитов белка NP_809341.1
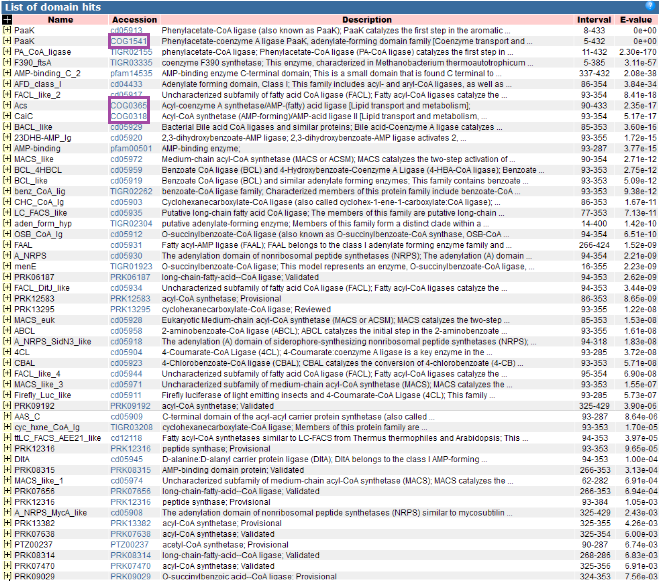
Для данного белка нашлось 3 кластера ортологических групп, в таблице ниже представлена информация по каждому КОГу в соотвествии с
последним релизом базы данных.
| ID | Координаты | E-value | Название (annotation) | Функциональная категория (cat) |
| COG1541 | 5-432 | 0e+00 | Phenylacetate-coenzyme A ligase PaaK, adenylate-forming domain family (фенилацетат-коэнзим А лигаза Paak (название гена), семейство аденилат-формирующих доменов | H: сoenzyme transport and metabolism (транспорт и метаболизм коэнзимов) |
| COG0365 | 90-433 | 2.35e-17 | Acyl-coenzyme A synthetase/AMP-(fatty) acid ligase (Ацил-коэнзим A синтетаза/АМФ-лигаза жирных кислот) | I: lipid transport and metabolism (транспорт и метаболизм липидов) |
| COG0318 | 93-354 | 5.17e-17 | Acyl-CoA synthetase (AMP-forming)/AMP-acid ligase II (Ацил-КоА синтетаза (АМФ-образующая)/АМФ-кислота лигаза II) | IQ: lipid transport and metabolism (транспорт и метаболизм липидов) и secondary metabolites biosynthesis, transport and catabolism (биосинтез, транспорт и катаболизм вторичных метаболитов) |
Задание 2. Визуализация геномного окружения.
Изображение геномного окружения КОГа было получено c помощью сервисов
STRING или COGNAT. Был выбран COG с лучшим E-value - COG1541. Поиск проводился по последовательности нашего белка (идентификатор STRING - BT_0428) по организму
Bacteroides thetaiotaomicron. Изображение STRING было получено при следющих параметрах:
| Max number of interactors to show/максимальное число взаимодействующих белков | 1st shell/первого уровня сродства | 2nd shell/второго уровня сродства |
| No more than 5 interactors/не более 5 | No more than 5 interactors/не более 5 |
| Minimum required interaction score/минимально требуемый балл взаимодействия | High confidence (0.700)/Высокий уровень достоверности |
Геномное окружение COG1541 (STRING)
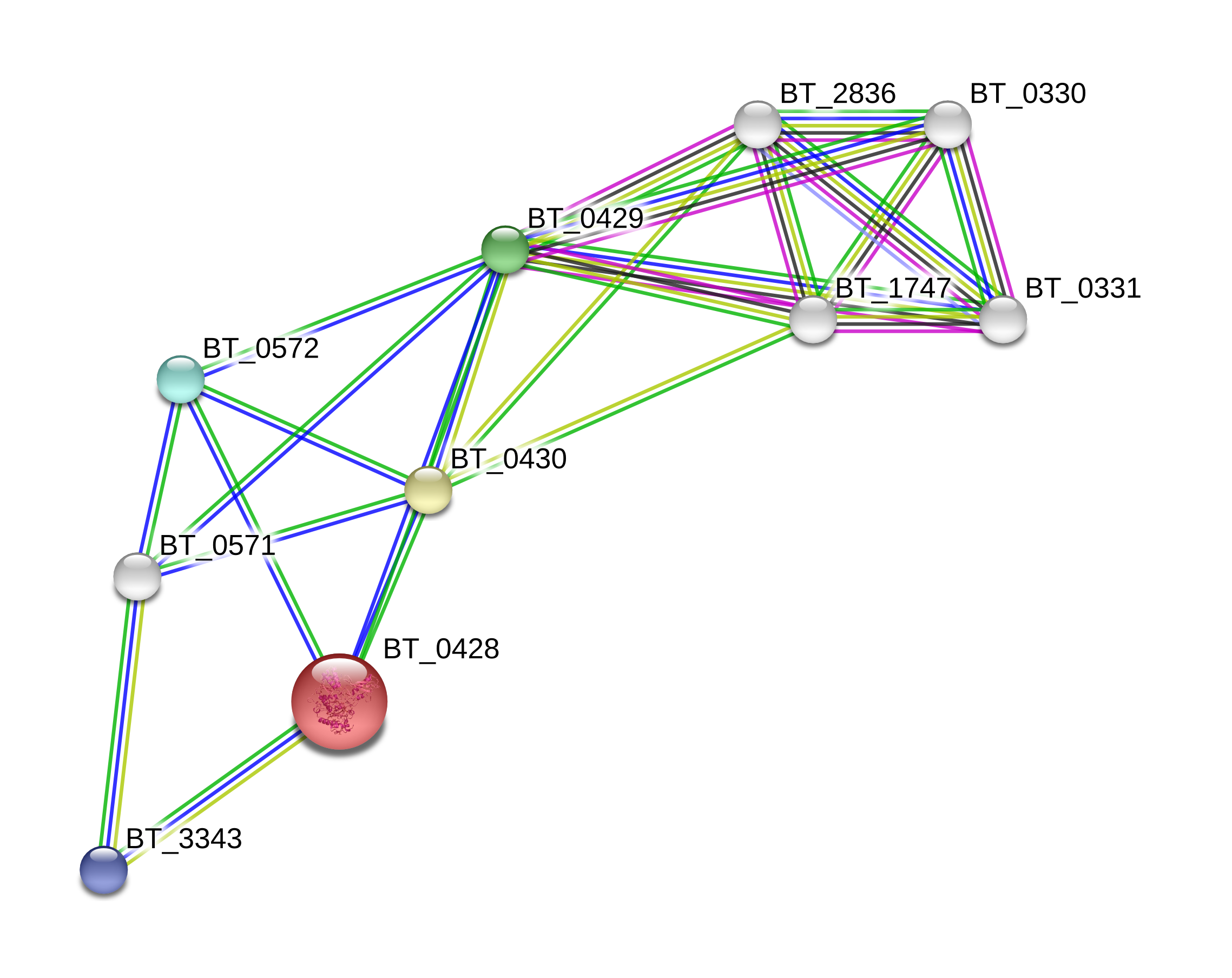
На рисунке в каждом узле графа расположен белок (совокупность изоформ, транскрибирующихся с одного локуса гена). Ребра графа символизируют
белок-белковые взаимосвязи (не обязательно физические связи, могут также наблюдаться функциональные связи). Белки с известной 3D-структурой отображаются вместе с
изображением структуры в больших узлах, цветные узлы - query protein и 1st shell interactors, белые узлы - 2nd shell interactors.
Цвет ребер отражает тип взаимосвязей.
На нашем графе наибольшее число ребер окрашены в синий, темно- или светло-зеленый цвета, отражающие не самые достоверные взаимосвязи. Доказанных экспериментально взаимосвязей не наблюдается.
Цвет ребер отражает тип взаимосвязей.
| Цвет ребра | Тип связи |
| Голубой | Информация о взаимосвязях из курируемых баз данных (from curated databases) |
| Фиолетовый | Экспериментально доказанные взаимосвязи (experimentally determined) |
| Темно-зеленый | Cоседство в геноме (gene neighborhood) |
| Красный | Cлияние генов (gene fusions) |
| Синий | Совместная встречаемость в геноме (gene co-occurrence) |
| Светло-зеленый | Cовместное упоминание белков в Pub-Med Abstract (textmining) |
| Черный | Коэкспрессия (co-expression) |
| Светло-синий | Гомология (protein homology) |
На нашем графе наибольшее число ребер окрашены в синий, темно- или светло-зеленый цвета, отражающие не самые достоверные взаимосвязи. Доказанных экспериментально взаимосвязей не наблюдается.
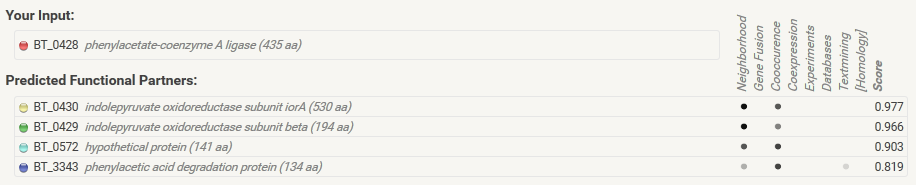
Таблица взаимосвязей для BT_0428 (STRING)
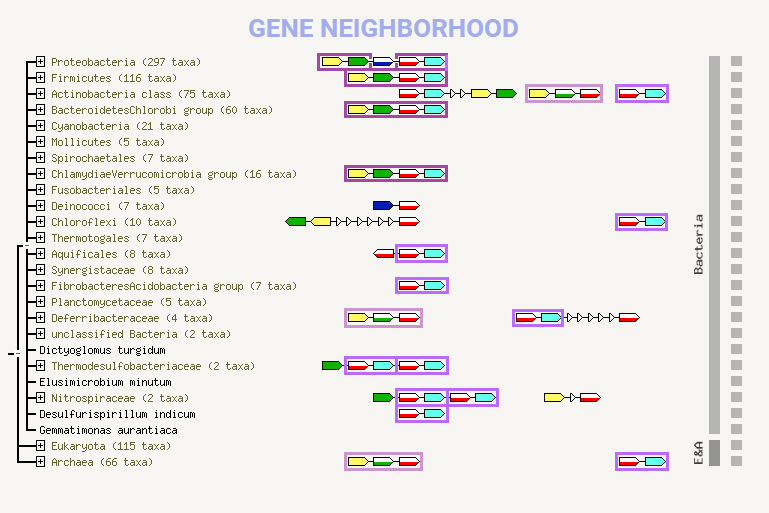
В полученных белках наблюдается консервативное окружение, что подтверждается наличием различных паттернов, указанных на рисунке (в графе,
соответственно, каждый белок связан с другим темно-зеленым ребром). Самый длинный паттерн из 4 белков образован следующими КОГами:
>| ID | Цвет на схеме | Название | Функциональная категория |
| COG4231 | Желтый | Индольпируват ферредоксин оксидоредуктаза (субъединица α) | C: energy production and conversion (выработка и конверсия энергии) |
| ? (не нашла в базе) | Темно-зеленый | Индольпируват оксидоредуктаза (субъединица β) | Вероятно, функциональная категория совпадает с предыдущим КОГом |
| COG1541 | Красная и белая горизонтальные полосы (отражают сложный характер ортологических отношений, что может быть связано с дупликацией генов или техническими проблемами с определнием ортологии) | Фенилацетил-коэнзим А лигаза | H: сoenzyme transport and metabolism (транспорт и метаболизм коэнзимов) |
| COG3830 | Бирюзовый | ACT domain protein, связывает аминокислоты и другие маленькие лиганды | T: signal transduction mechanisms (механизмы каскада трансдукции сигнала) |
Встречается паттерн в таксонах Proteobacteria, Firmicutes, Bacterioudetes Chlorobi group, Chlamydiae Verrucomicrobia group и у протеобактерий.
Очевидно, что 2 субъединицы индольпируват ферредоксин оксидоредуктазы должны быть связаны функционально. Кроме того, упомянутый ACT домен [1],
[2] является С-концевым регуляторным доменом 3-фосфоглицератдегидрогеназы, которая по топологии упаковки напоминает ферредоксин.
Геномное окружение достаточно консервативно, однако можно наблюдать дупликации и индели у некоторых групп организмов.
Геномное окружение достаточно консервативно, однако можно наблюдать дупликации и индели у некоторых групп организмов.
Gene neighbourhood для BT_0428 (STRING)
Задание 3. Отнесение белка NP_809341.1 к терминам GO.
Использовался поиск по последовательности моего белка инструментом AmiGO. Самым
близким белком по результатам работы программы оказался белок Q74CD7 (Uniprot) - фенилацетат-коэнзим А лигаза из
организма Geobacter sulfurreducens PCA. Несмотря на то, что бактерии относятся к достаточно далеко отстающим друг от друга кладам - типам Bacteroides и
Proteobacteria, думаю, что белки схожи, так как в выравнивании из 235 позиций наблюдаются 15 блоков, E-value составляет 2.0e-102, число совпадений удовлетворительно
.
Лучшее выравнивание по результатам BLAST AmiGO

На странице белка по ссылке
N term associations размещена информация о соответствующих данному белку терминах GO. Для этого белка нашелся только 1 термин GO, поэтому я объединила
описанные в задании таблицы 1 и 2 в одну.
| Аспект | Идентификатор GO | Название термина | Перевод названия термина | Код типа достоверности |
| Молекулярная функция (Molecular function) | GO:0016874 | Ligase activity | Лигазная активность | ISS |
| Код | Расшифровка | Объяснение | ISS | Inferred from sequence similarity/в основе - сходство последовательностей или структурное | ISS код или одна из его разновидностей используется, когда в основе аннотации лежал анализ последовательностей, при этом он должен был быть проверен вручную. Если такой проверки не производилось, корректно использовать код IEA. Общий код ISS должен быть указан, если применяется комбинация анализирующих сиквенсы инструментов и методов. В случае использования лишь одного метода следует указывать одну из подкатегорий ISS: ISA (Inferred from Sequence Alignment) - при анализе парного или множественного выравнивания, ISO (Inferred from Sequence Orthology) - при оценке ортологичности продуктов генов из разных организмов, ISM (Inferred from Sequence Model) - при использовании какого-либо метода моделирования (например, скрытой марковской модели). ISS также можно использовать при наличии структурного сходства с экспериментально описанными продуктами генов, установленного с помощью кристаллографии, ЯМР или вычислительных предсказаний. На практике код ISS практически никогда не применяют для аннотации, основанной только на информации о структуре. Если информация о ней и включается, то обычно она представляет собой данные моделирования вторичной структуры или предсказания структуры, основанного на последовательности. Информация о вторичной структуре бывает особенно полезна в качестве одного из компонентов предсказаний РНК-генов и в некоторых моделях доменов. |