Поиск сигналов. Chip-seq.
Определение сайтов связывания ТФ в заданном участке хромосомы человека.
1. Проверка качества чтений в FastQC
Работа велась с выданным мне файлом chipseq_chunk40.fastq (с ридами Illumina, полученными в результате ChIP-seq эксперимента). Был проведен контроль качества прочтений с помощью программы FastQC, работа которой описана в практикуме 10 прошлого семестра:
fastqc chipseq_chunk40.fastq
Качество по нуклеотидам из выдачи FastQC
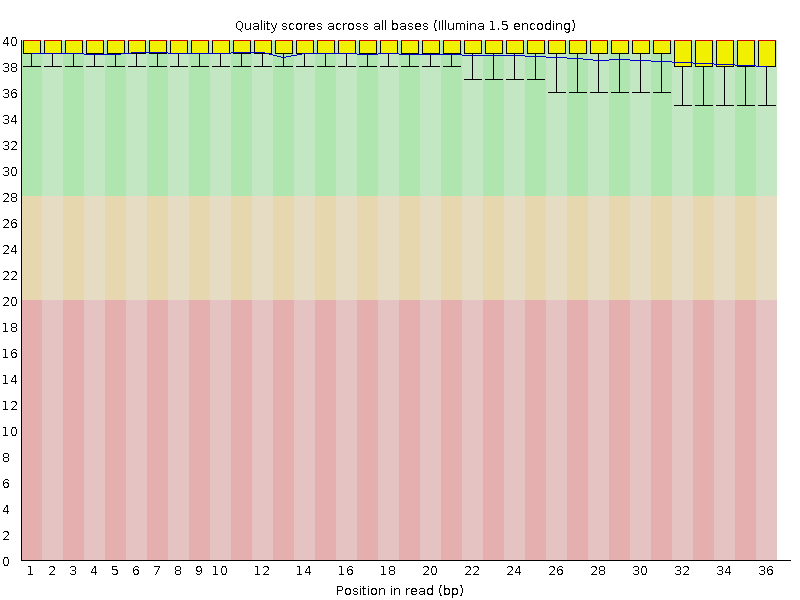
Как можно видеть, качество ридов очень хорошее: даже «усы» гистограммы попадают в зеленую область, соответствующую отличному качеству (>28).
2-4. Картирование прочтений на геном человека
Было произведено картирование чтений на геном человека (референсная последовательность hg19) посредством алгоритма BWA-MEM. На выходе — выравнивание в формате sam:
bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk40.fastq >Перевод sam-выравнивания в бинарный bam-формат:
chipseq_chunk40.sam
samtools view chipseq_chunk40.sam -b -o chipseq_chunk40.bamCортировка bam-выравнивания по координате начала прочтения в референсе. Параметр -T нужен для указания пути, по которому сохраняются временные файлы. В итоге имеем файл sorted_chunk40.bam:
samtools sort chipseq_chunk40.bam -T temp -o sorted_chunk40.bamИндекcирование отсортированного файла:
samtools index sorted_chunk40.bamПолучение информации о количестве картированных прочтений и запись ее в файл idxstats_chunk40:
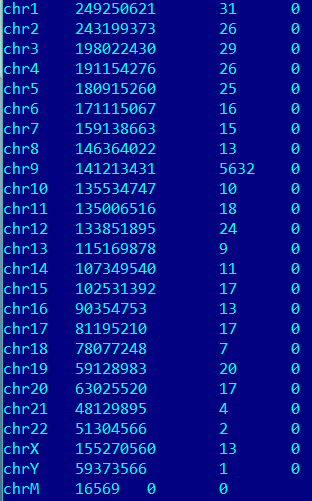
samtools idxstats sorted_chunk40.bam > idxstats_chunk40Откартировались все чтения: 6005. Больше всего (5632) ридов картировалось на девятую хромосому, следовательно, были выданы риды 9 хромосомы.
5. Поиск пиков
Для поиска пиков (peak calling) воспользовались программой MACS, которая установлена на kodomo. Параметр outdir задает директорию, имя эксперимента указывается в -n (--name) (для меня это "chunk40" в названии). Дополнительный параметр --nomodel использован для того, чтобы не включать построение модели shifting model, что в данном случае невозможно, так как наблюдается мало пиков.macs2 callpeak -t sorted_chunk40.bam --nomodel --outdir callpeak -n chunk40Так были получены следующие файлы: .narrowPeak, .xls, .bed. В таблице ниже приведена информация из chunk40_peaks.xls по лучшим пикам.
Информация о пиках из выдачи MACS2
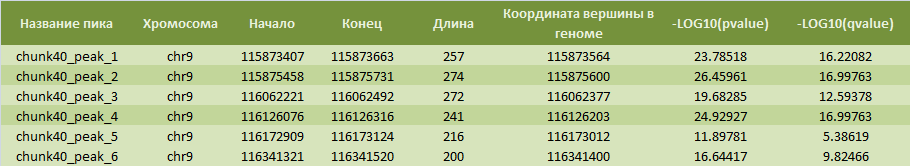
Всего нашлось 6 пиков, принадлежащих 9 хромосоме. Если посмотреть на ширину пиков, то варьируется она в пределах сотни: от 200 (peak_6) до 274 (peak_2). Так как мы берем отрицательные десятичные логарифмы p-value и q-value, то сами значения тем лучше (меньше), чем больше их логарифмы, чем и определяется достоверность пиков. Самой надежной находкой является пик (peak_2) имеет показатели: -log10(p-value): 26,46 и -log10(q-value): ≈17. Худшие же показатели у peak_5: 11,9 и 5,39 соотвественно.
6. Визуализация пиков
Визуализированы пики при помощи UCSC Genome Browser. Для работы программы нужно было подкорректировать файл .narrowPeak, добавив следующие строки:track type=narrowPeak visibility=3 db=hg19 name="chunk40_peaks"
description="Peaks from chunk 40" browser position chr9:115873000-116342000
Скриншот выдачи UCSC Genome Browser
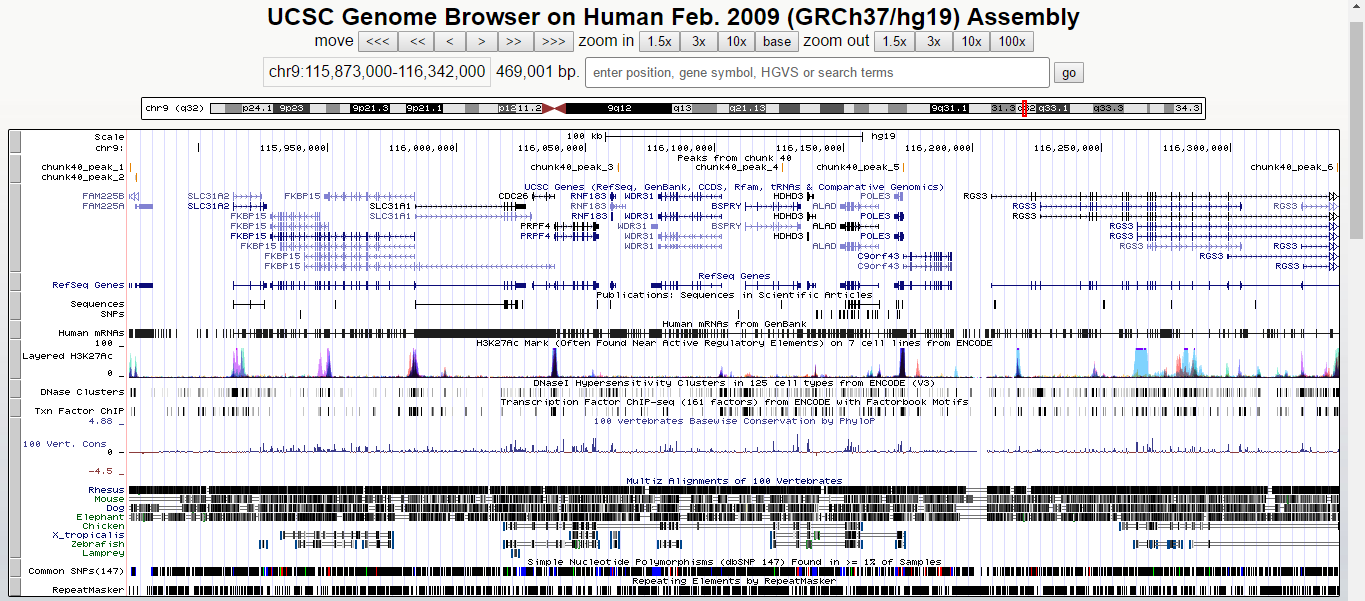
7. Описание некоторых пиков
Рассмотрим более подробно второй и четвертый пики, поскольку они самые широкие и наилучшие по качеству.Крупномасштабное изображение peak_2 в геномном браузере UCSC
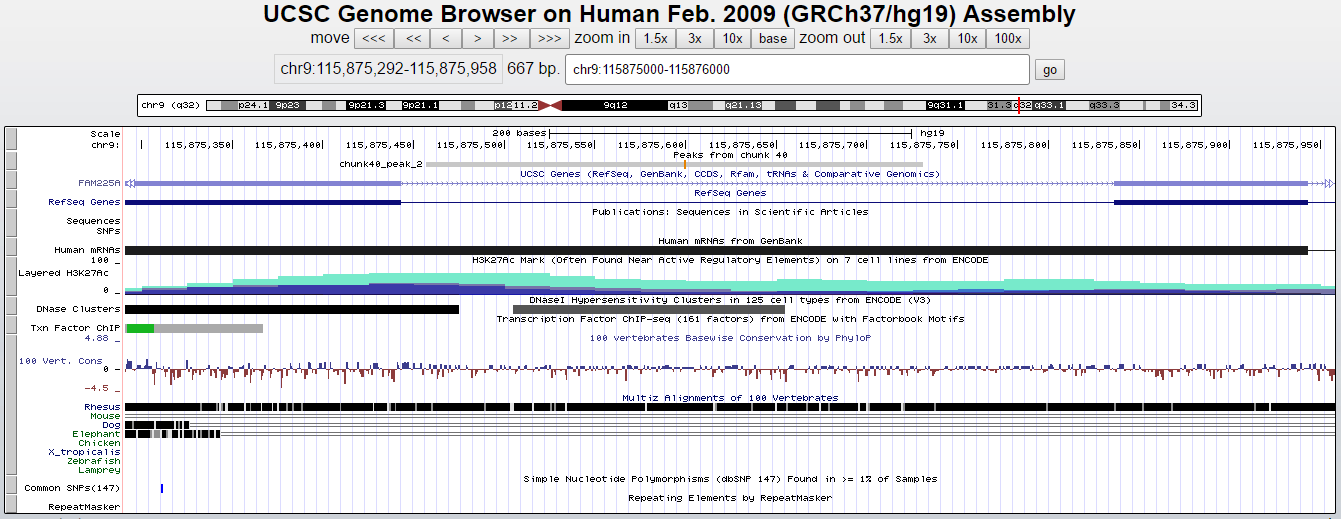
Параметры:
- координаты в геноме: 115875458-115875731
- ширина пика: 274
- расстояние от начала пика до вершины: 142
- расположение вершины в пике: 142:132
- достоверность: -log10(p-value): 26,46 и -log10(q-value): ≈17
Крупномасштабное изображение peak_4 в геномном браузере UCSC
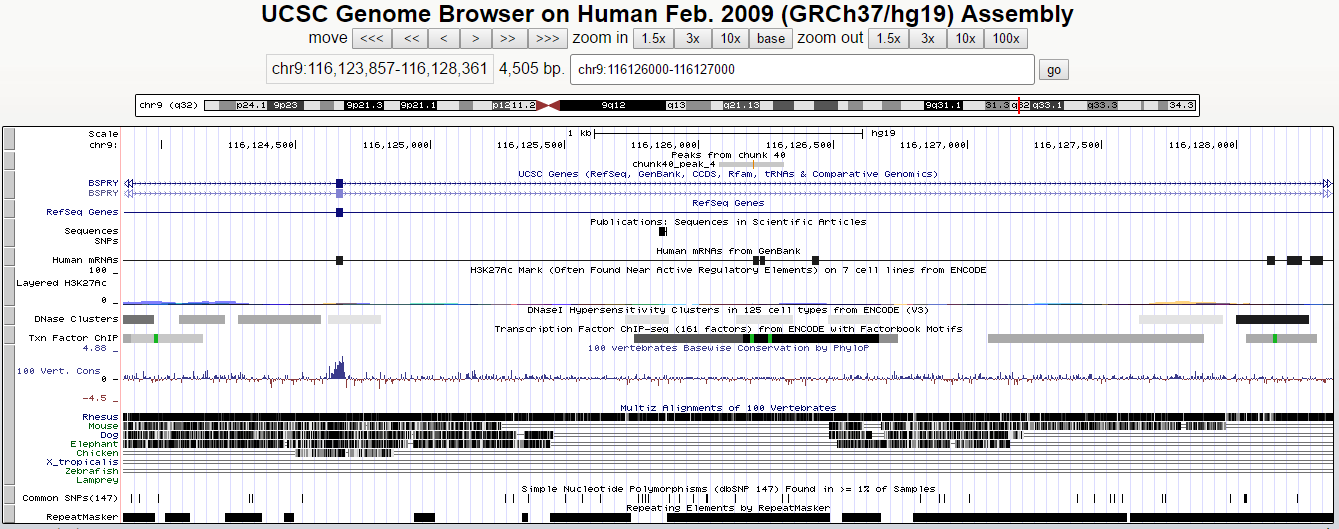
Параметры:
- координаты в геноме: 116126076-116126316
- ширина пика: 241
- расстояние от начала пика до вершины: 127
- расположение вершины в пике: 127:114
- достоверность: -log10(p-value): 24,92 и -log10(q-value): ≈17
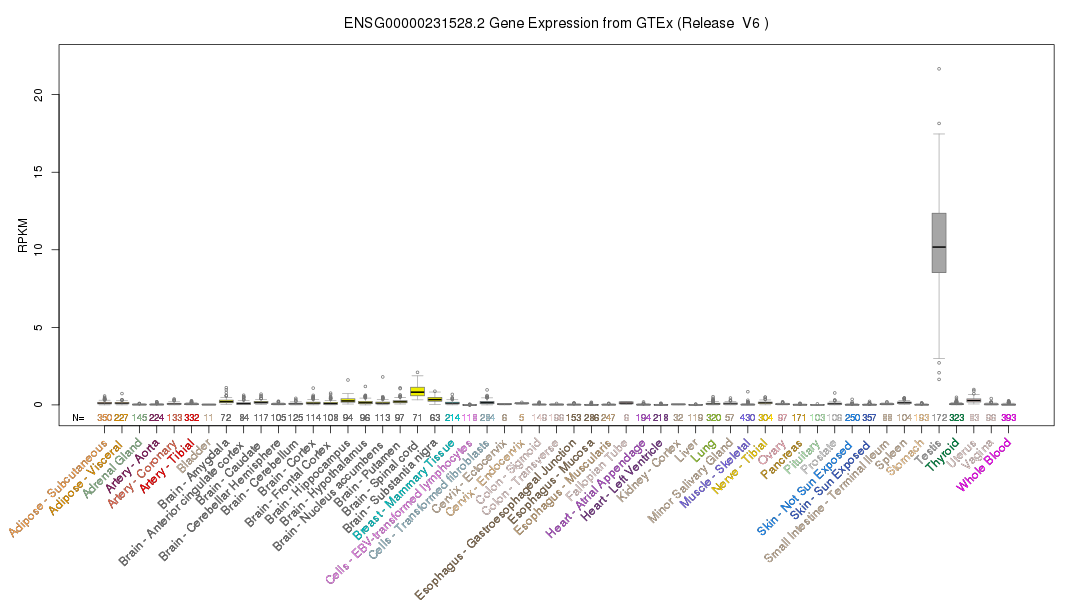
Что касается пересечений данных пиков с функциональными элементами генома, то в моем случае пик 2 пересекается с интроном гена FAM225A.
Экспрессия гена FAM225A
Пик 4 перескается c интроном гена BSPRY. Вообще на NCBI пишут: «Description: Homo sapiens B-box and SPRY domain containing (BSPRY)». SPRY-домен назван в честь SPla-RYanodine рецептора.