Практикум 8
Варианты BLAST для выбранного контига
Я выбрал участок континга 2ой хромосомы NC_062545.1, с координатами: 70547-71436. Он содержит CDS, кодирующий белок XP_047636145.1 .
Координаты всего континга:1..280040501
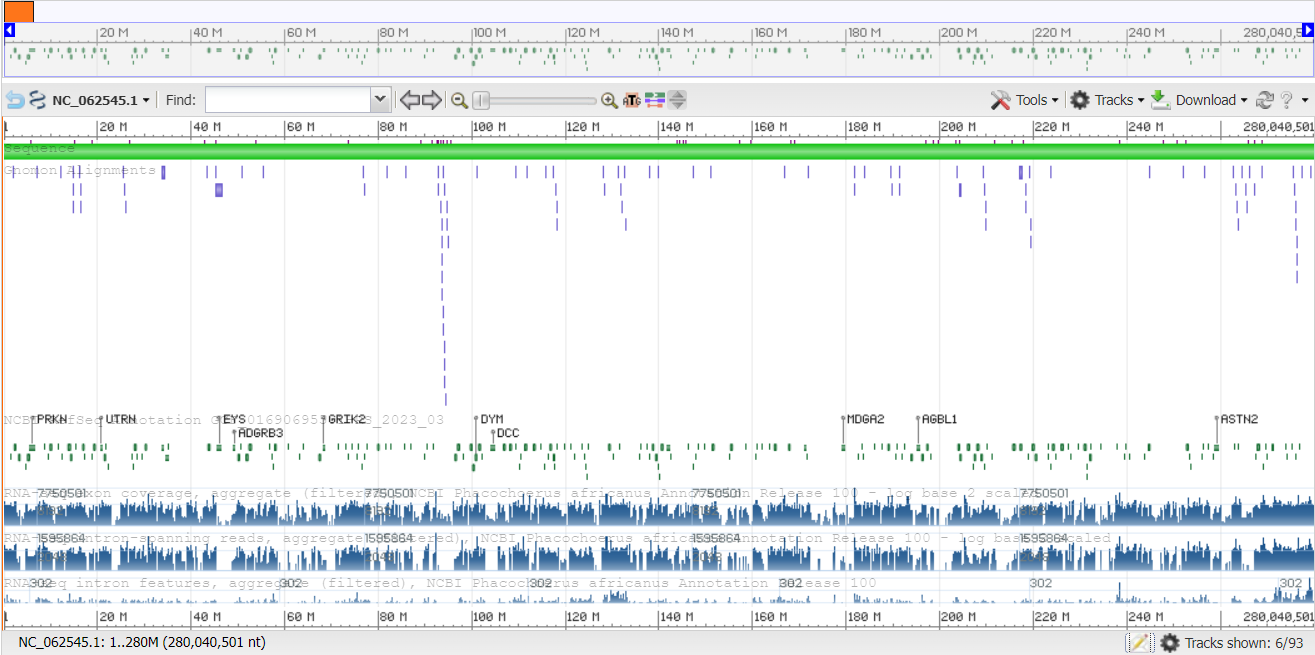
1 строка - расположение генов, 2 - аннотации, 3 - покрытие экзонами, 4 - считывание RNAseq с охватом интронов, 5 - признаки интрона (полученные из выравниваний по RNAseq.)
Скачать последовательность можно по ссылке,(transeq).
MegaBlast
В мегабласте я искал среди рыб,птиц - ничего не находилось,и только при поиске среди homo sapiens - программа выдала 2 находки, обе - синтетические клоны. Длина слова по умолчанию.
Решает задачи типа: поиск с высокой степенью идентичности, картирование на известный геном, поиск мутаций.
BlastX
В бласт икс более 100 находок, но почти все - синтетические конструкции белков программируемой клеточной смерти.
BLASTx ‐ мощный инструмент для поиска или прогнозирования генов. Рекомендуется для идентификации белок‐кодирующих генов в геномной ДНК / кДНК. Он также используется для определения того, является ли новая нуклеотидная последовательность геном, кодирующим белок, или нет, и его можно использовать для идентификации белков, кодируемых транскриптами или вариантами транскриптов.
TBlastX
TBlastX более чувствителен к расходящимся последовательностям. Он может лучше определять сходство между отдаленно родственными последовательностями, чем BlastN.
BlastN
Находок - 9.
BlastN быстрее, потому что он производит гораздо меньше сравнений, и каждое сравнение более простое, чем tblastx.
Поиск генов основных рибосомальных РНК по далекому гомологу
В этом задании мы должны проиндексировать последовательности генома нашего эукариота для работы локального BLAST и провести локальный поиск BLAST отдельно для каждой рРНК
wget ftp://ftp.ncbi.nih.gov/genomes/refseq/vertebrate_mammalian/Phacochoerus_africanus/all_assembly_versions/GCF_016906955.1_ROS_Pafr_v1/GCF_016906955.1_ROS_Pafr_v1_genomic.fna.gz
gzip -d GCF_016906955.1_ROS_Pafr_v1_genomic.fna.gz
makeblastdb -in GCF_016906955.1_ROS_Pafr_v1_genomic.fna -dbtype nucl
blastn -task "blastn" -query "16S_ecoli.fasta" -db "GCF_016906955.1_ROS_Pafr_v1_genomic.fna" -out "16S.tsv" -outfmt 7
Для 16S рнк было получено 8 находох, среди которых 3 имеют высокий уровень схожести, но e-value слишком большие.
Для 23S рнк было получено 13 находох, из которых первые 5 на одну хромосому. Почти все имеют высокий уровень схожести,но 2 не подходят по evalue, и только 2 из оставшихся имеют приемлимую длину выравнивания.