1. Значение кода заданного фермента
На странице БД UniProt с описанием белка UXAC_ECOLI был найден его EC код: EC=5.3.1.12. С помощью этого кода и номенклатуры IUBMB (INTERNATIONAL UNION OF BIOCHEMISTRY AND MOLECULAR BIOLOGY) можно определить функцию данного фермента.Первая цифра кода (5) означает, что фермент принадлежит к классу изомераз (Isomerases), вторая цифра (3) указывает на принадлежность к подклассу внутримолекулярных оксидоредуктаз (Intramolecular Oxidoreductases). Третья цифра (1) - данный фермент участвует в реакции превращения альдоз в кетозы (Interconverting Aldoses and Ketoses), четвертая (12) - окончательно конкретизирует его функцию, а именно - фермент катализирует реакцию изомеризации глюкуронатов (glucuronate isomerase).
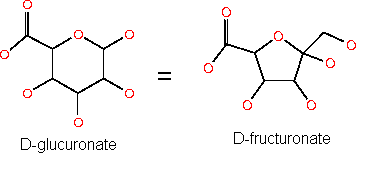
Уравнение катализируемой реакции: D-glucuronate = D-fructuronate, также может катализировать реакцию D-galacturonate=D-tagaturonate.
Схема реакции:
2. Метаболические пути, в которых участвует изучаемый фермент
Имя локуса гена белка UXAC_ECOLI - b3092. Проведем поиск в БД KEGG по названию локуса гена и идентификатору штамма Escherichia coli K12 - eco.С данным геном ассоциированы 2 метаболических пути:
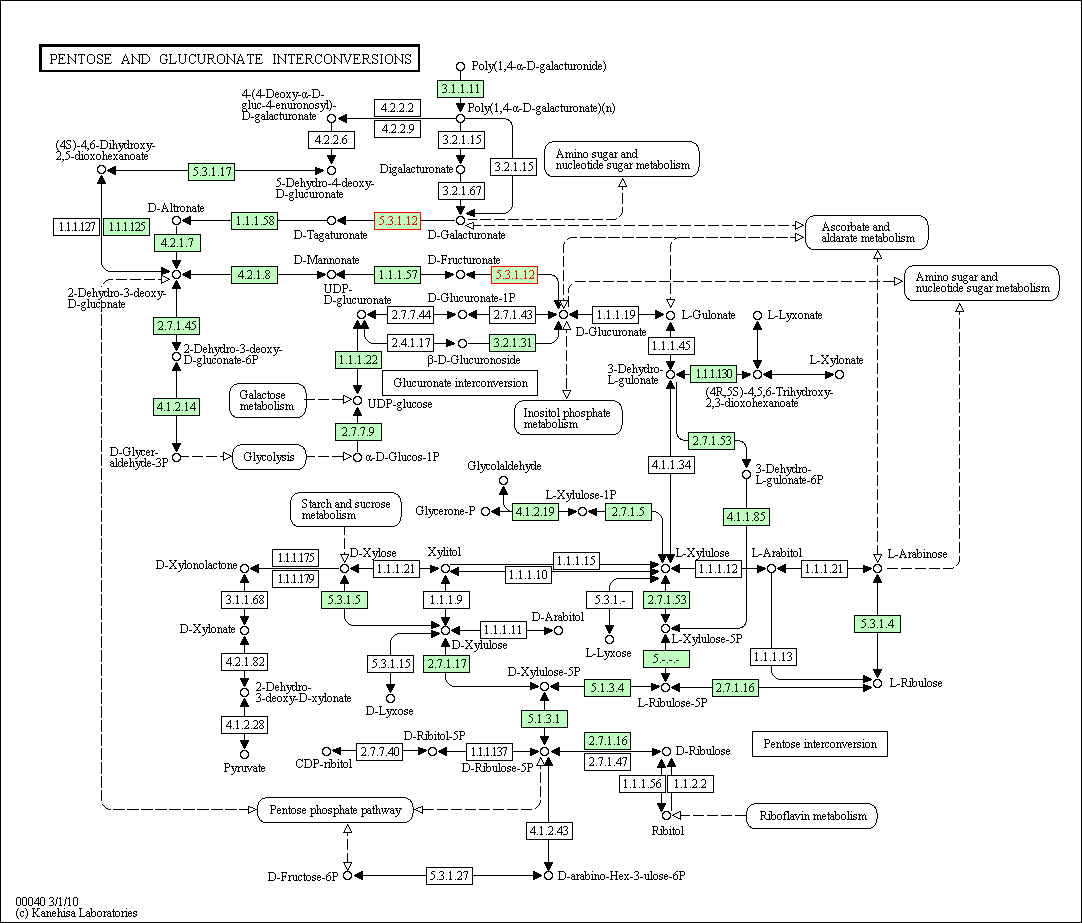
1. eco00040 Pentose and glucuronate interconversions (Превращения пентозы и глюкуронатов)
2. eco01100 Metabolic pathways (Метаболические пути)
Карта первого пути:
3. Поиск в KEGG структурных формул заданных соединений
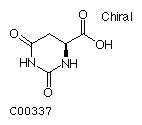
Воспользуемся БД KEGG Ligand:1. Дигидрооротат (dihydroorotate): C00337
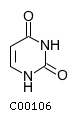
2. Урацил (uracil): C00106
4. Поиск метаболического пути от одного заданного вещества к другому
Воспользуемся БД KEGG Pathway:Выбранная цепочка ферментативных реакций:
путь: метаболизм пиримидинов,
цепочка: дигидрооротат > урацил.
Файл png (красным цветом обозначено начальное соединение, зеленым - конечное, желтым - промежуточные).
5. Сравнение метаболических путей у разных организмов
| Организм | Возможна ли цепочка реакций (да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 | да | цепочка превращений полностью совпадает с приведенной выше (см. Файл png), в ней присутствуют все ферменты, необходимые для осуществления данных реакций |
| Archaeoglobus fulgidus | да | -||- |
| Arabidopsis thaliana | да | -||- |
| Homo sapiens | да | -||- (см. карту) |
Оказалось, что рассмотренная мною часть цикла метаболизма пиримидинов полностью совпадает в очень далеких друг от друга организмах.
Скорее всего, данная цепочка ферментативных реакций является наиболее эффективным способом получить урацил из дигидрооротата, а может быть и единственно возможным. Поэтому эта цепочка реакций осталась неизменной в ходе эволюции.
6. Сравнение ферментов из далеких организмов
1. Найдем ферменты с EC:2.5.1.61 y человека и археи (Archaeoglobus fulgidus).Запрос к SRS: ([uniprot-ECNumber:2.5.1.61] & ([uniprot-ID:*_HUMAN] | [uniprot-ID:*_ARCFU])). В запросе также отключим опцию использования маски, чтобы не находились ферменты с EC:2.5.1.61* (например 2.5.1.613).
По запросу было найдено 2 белка - 1 у человека и 1 у археи: HEM3_HUMAN и HEM3_ARCFU.
2. Воспользуемся режимом SW_InterProMatches для сравнения доменной организации найденных белков.
HEM3_HUMAN

Домены: Porphobil_deam (20-236 а.о.) и Porphobil_deamC (244-324 а.о.).
HEM3_ARCFU

Домены: Porphobil_deam (2-212 а.о.) и Porphobil_deamC (217-289 а.о.).
Как мы видим, доменная организация этих белков очень похожа.
3. Для определения % совпадения последовательностей гомологичных доменов из археи и человека проведем выравнивание (needle) fasta-файлов с последовательностями доменов (Porphobil_deam) белков.
Результаты выравнивания:
Identity: 85/218 (39.0%) Similarity: 123/218 (56.4%) Gaps: 8/218 ( 3.7%)Процент совпадения = 39,0.
4. Найдем лучшего ортолога HEM3_HUMAN (имя гена - HMBS) из архей:
mac:MA0582, имя гена - hemC, организм - Methanosarcina acetivorans, процент совпадения - 38,8, длина выравнивания - 317 а.о.
Лучший ортолог белка HEM3_ARCFU (имя гена - hemC) из эукариот:
hmg:100211781, имя гена - отсутствует, организм - Hydra magnipapillata, процент совпадения - 45, длина выравнивания - 450.
Назад