Краткий обзор генома и протеома бактерии Brachybacterium halotolerans subsp. Kimchi
Голуб Е.Р.
Факультет биоинженерии и биоинформатики, МГУ имени М.В. Ломоносова,
bioeng.msu@gmail.com
Аннотация:
В данном мини-обзоре были исследованы геном и пересекающиеся гены белков Brachybacterium halotolerans subsp. Kimchi CBA3105T
Контакты: goliro22@mail.ru
1 Введение
Brachybacterium halotolerans subsp. Kimchi – подвид грамположительных, оксидазоотрицательных, каталазополо-жительных и кокковидных бактерий, обозначается как CBA3105T. Таксономическое название было опубликовано фактически и не в соответствии с правилами Международного кодекса номенклатуры прокариот (Бактериологический кодекс)
[1]. Данный штамм относится к виду Brachybacterium halotolerans , роду Brachybacterium, семейству Dermabacteraceae, подклассу Micrococcales, классу Actinomycetia (high G+C Gram-positive bacteria, типу Actinomycetota и надтипу Terrabacteria group)
[1]. О данной бактерии мало что известно, так как заявка на регистрацию была подана Всемирным институтом Кимчи 16 мая 2022 года
[2], а сама научная статья об открытии двух новых штаммов бактерий рода Brachybacterium была опубликована Журналом Микробиологии при сотрудничестве с Микробиологическим обществом Кореи 4 июля 2022 года[3]. Тем не менее, филогенетический анализ на основе последовательностей генов 16S рРНК показал, что штамм CBA3104T образует отдельную филогенетическую линию внутри рода Brachybacterium, тогда как штамм CBA3105T тесно связан с Brachybacterium halotolerans MASK1Z-5T. Среди результатов биохимического анализа штамм CBA3105T был положительным по щелочной фосфатазе и α-фукозидазе среди двух новых штаммов, близкородственных штаммов и типовых видов рода Brachybacterium. По сравнению со штаммом CBA3105T и Brachybacterium halotolerans JCM 34339T, штамм CBA3105T был дифференцированно положительным по кислотной продукции D-арабинозы, D-адонитола и 5-кетоглюконата калия и активности фермента β-глюкуронидазы. Результаты биохимического/физиологического, хемотаксономического и геномного анализов показали, что штаммы CBA3104T и CBA3105T представляют собой новый вид рода Brachybacterium и новый вид B. halotolerans, соответственно, для которых предложены названия Brachybacterium kimchii sp. nov. и B. halotolerans subsp. kimchii subsp. nov.
2 Материалы и методы
Информация о геноме и протеоме бактерии была взята из базы данных NCBI Genome(https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/969/905/GCF_000969905.1_ASM96990v1/). Обработка данных проводилась с помощью электронных таблиц Google sheets. Были использованы строки таблицы особенностей генома, а именно кодирующие последовательности (CDS) в колонке “features” (значения были отобраны благодаря фильтру). В пункте 3.1 с помощью функций в Google sheets на основе таблицы с данными о геноме было рассчитано распределение длин белков и была построена гистограмма числа аминокислотных остатков в белках бактерии.
В пункте 3.2 с помощью функций Google sheets на основе таблицы с данными о геноме были выбраны кодирующие последовательности на плюс-цепи самой большой хромосомы, пересекающиеся со следующей CDS на той же цепочке (строки были отсортированы по начальной координате гена белка (CDS) благодаря фильтру). Гены, не пересекающиеся с другими, были удалены также с использованием фильтра. Была построена гистограмма длин пересечений.
3 Результаты
3.1 Распределение длин белков
Гистограмма протеома(Рис.1) отражает количество белков и их длину в протеоме бактерии. Наибольшее количество белков составляет 200-300 аминокислот в длину и достигает 1738 штук, это соответствует среднему значению аминокислот- 283. Следом идут 300-400 аминокислот, которые составляют меньшее число белков- 1530. На гистограмме можно наблюдать всего один пик. Есть резкий перепад между диапазонами 300-400 и 400-500 аминокислот.

Рисунок 1. Гистограмма длин белка в протеоме Brachybacterium halotolerans subsp. Kimchi
3.2 Исследование межгенных промежутков
Из гистограммы видно, что наибольшее количество расстояний между CDS имеют длину до 100 пар нуклеотидов, их 4772. Длину 100-200 пар нуклеотидов имеют уже значительно меньше расстояний, их 580. В диапазон от 200 до 300- 76 межгенных расстояний.

Рисунок 2. Гистограмма расстояний между последовательными кодирующими последовательностями (CDS) на плюс-цепи самой большой хромосомы в геноме Brachybacterium halotolerans subsp. Kimchi
3.3 Распределение генов по репликонам
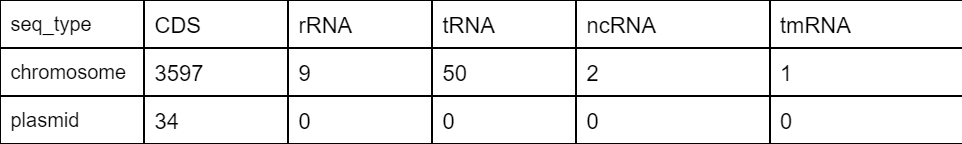
Таблица 1.
Прежде чем анализировать полученные данные, нужно дать некоторые определения:
CDS- кодирующая последовательность- последовательность нуклеотидов в ДНК или РНК. Всего у Brachybacterium halotolerans subsp. Kimchi 3597 кодирующих последовательностей.
ncRNA- молекулы РНК, которые не транслируются в белки.
tmRNA - это небольшая РНК длиной от 260 до 430 нуклеотидов, которая участвует в высвобождении рибосом, «застрявших» во время трансляции проблемных участков мРНК, а также разрушении получившихся в ходе неполной трансляции дефектных пептидов.
tRNA- транспортная РНК, обеспечивающая взаимодействие аминокислоты, рибосомы, матричной РНК в ходе трансляции.
rRNA— несколько молекул РНК, составляющих основу рибосомы.
Можно сделать вывод, что в клетках бактерии присутствует каждый тип РНК в количествах, представленных в таблице 1.
4 Сопроводительные материалы
(C1) Таблица с расчетами по распределению длин белков и пересечениям кодирующих последовательностей:Golub_genome
5 Благодарности
Приносятся благодарности преподавательскому составу курса практической биоинформатики 2023 года факультета биоинженерии и биоинформатике за обучение в течение семестра и предоставление возможности принимать участие во всемирных исследованиях, а также материалов для написания краткого обзора генома бактерии.
6 Заключение
В данном мини-обзоре были изучены вариации в длине белков, распределение генов по репликонам и исследованы межгенные промежутки кодирующие последовательности в геноме бактерии Brachybacterium halotolerans subsp. Kimchi. Эти данные могут быть полезными в последующих исследованиях как данной бактерии, так и организмов.
Meier-Kolthoff, J.P., Göker, M. TYGS is an automated high-throughput platform for state-of-the-art genome-based taxonomy. Nat Commun 10, 2182 (2019).
https://tygs.dsmz.de
Пакет данных генома, включая геном, транскрипт и последовательность белка, аннотацию и отчет о данных о Brachybacterium halotolerans subsp. Kimchii:
https://www.ncbi.nlm.nih.gov/datasets/taxonomy/2887346/"
Источники
3.Kim, Y., Kim, Y.B., Kim, J. et al. Brachybacterium kimchii sp. nov. and Brachybacterium halotolerans subsp. kimchii subsp. nov., isolated from the Korean fermented vegetables, kimchi, and description of Brachybacterium halotolerans subsp. halotolerans subsp. nov.. J Microbiol. 60, 678–688 (2022):
https://link.springer.com/article/10.1007/s12275-022-1581-6