1. Выбор и описание семейства доменов из Pfam
Для проведения анализа из таблицы доменов Pfam было выбрано семейство доменов PF07361 Cytochrome b562.
| AC | ID | SEED | Full | Domain Architectures | 3D structures | Taxonomy |
|---|---|---|---|---|---|---|
| PF07361 | Cytochrome b562 | 32 | 2k | 6 | 445 | 2k |
Таблица 1. Таблица характеристик.
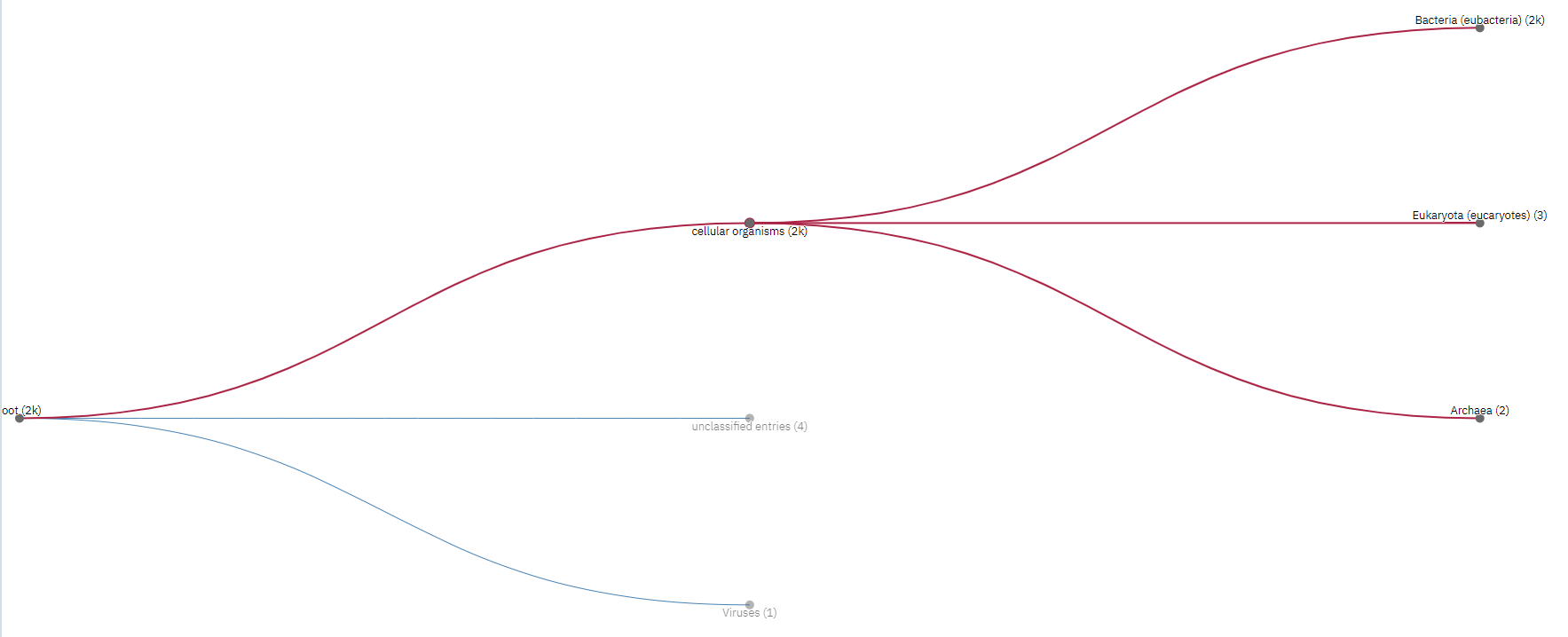
Рис. 1. Таксономия .
Всего в графе Taxonomy представлено 2000 таксонов. Среди них абсолютное большинство бактерий, всего 3 эукариота и 2 археи.
Цитохром b562 - периплазматический белок кишечной палочки. Он образует четырехспиральный пучок, который нековалентно связывает одну простетическую группу гема
3. Описание выравнивания seed с точки зрения гомологичности всех последовательностей или их подмножества
1.Максимальный блок достоверного выравнивания - включающий фрагменты из всех последовательностей
С помощью окрашивания через параметр Above identity threshold (100%) я получила единичные аминокислоты, консервативные во всех последовательностях: 6 и 112.
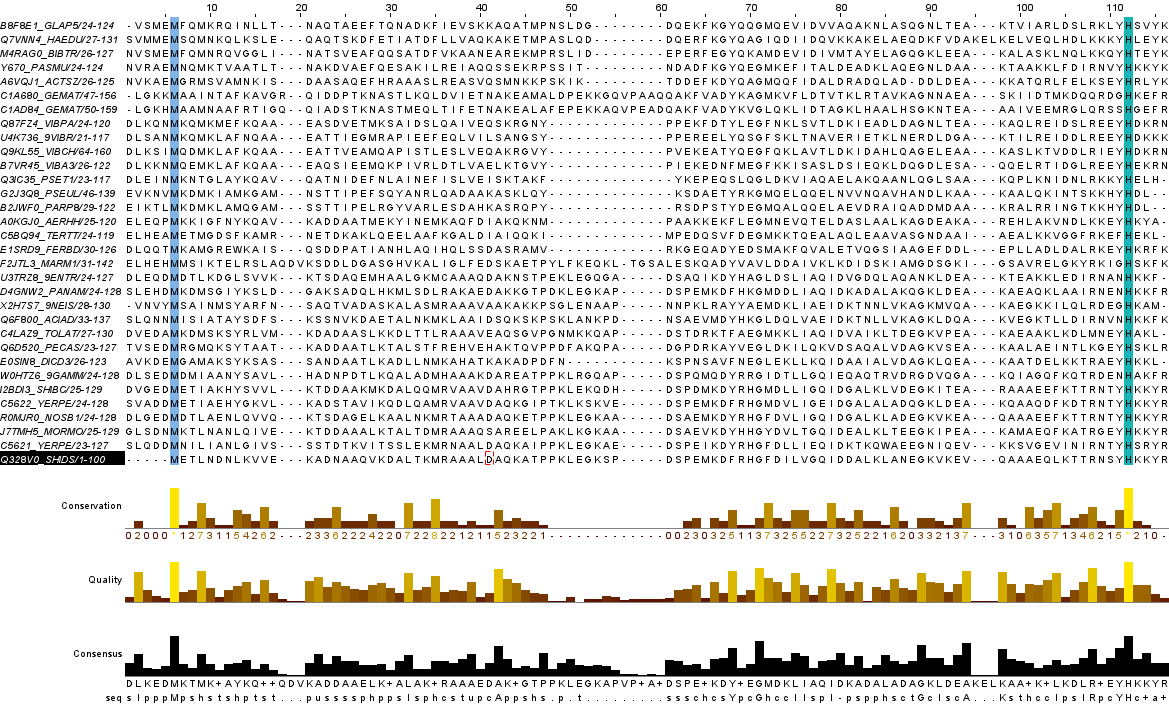
Рис. 1. Выравнивание seed.
2. Максимальный блок достоверного выравнивания - включающий фрагменты не из всех последовательностей
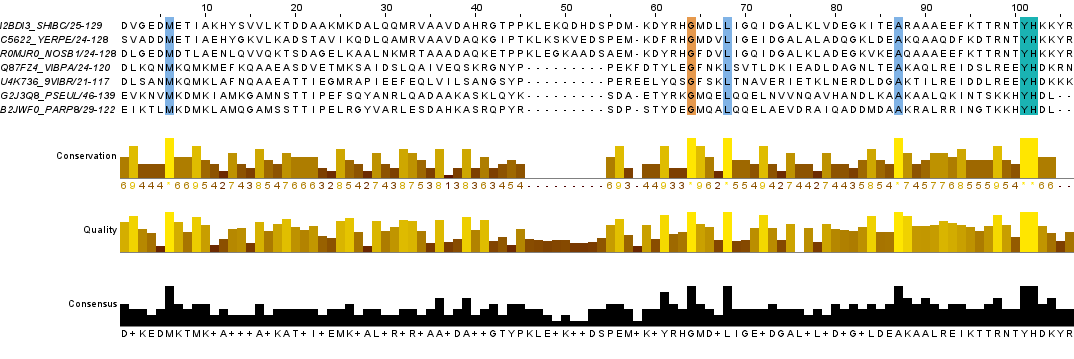
Рис. 2. Выравнивание seed для группы последовательностей.
Мною были выбраны наиболее схожие последовательности. Перевыравнивая тем же способом в новом окне, я получила максимальный блок - 101-102. А также единичные аминокислоты, консервативные во всех последовательностях: 6, 64, 68, 87.
3. Участок выравнивания, на котором нет основания считать, что выравнивание отражает ход эволюции
Участки 18-20, 47-61, 95-97 не отражают ход эволюции, в них нельзя выделить достоверные подблоки из-за гэпов в большинстве последовательностей.
5. Построение карты локального сходства (dotplot) двух последовательностей с одним и тем же доменом, но с разной доменной архитектурой
Для этого задания были выбраны две архитектуры: PF07361 (AC:O32554) и PF07361 - PF07361 (AC: A0A5M7PZ51).
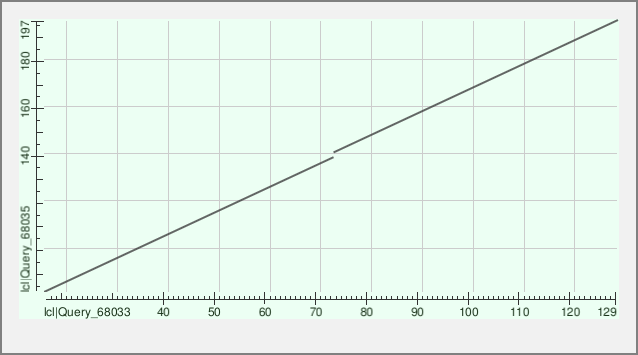
Рис. 3. Карта локального сходства двух белков.
На карте локального сходства присутствует 1 разрыв, что соответствует инделям в выравнивании.
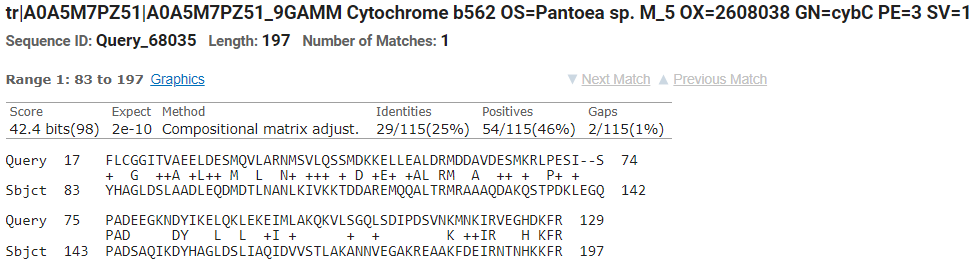
Рис. 3. Выравнивание BLAST.