Практикум 10
Поиск подходящих последовательностей
Я решил сравнить две близкородственные бактерии рода Thermus, для поиска геномов я использовал продвинутый поиск на сайте ENA со следующим запросом.
Далее я выбрал референсные последовательности. Thermus antranikianii(CP076325.1), Thermus aquaticus(CP010822)
Анализ карты локального сходства
Для того чтобы результаты выравнивания c помощью megablast и blastn с параметрами по умолчанию как либо отличались пришлось перебрать огромное количество сочетаний, в целом разница заметна только для достаточно коротких последовательностей(до тысячи килобаз).
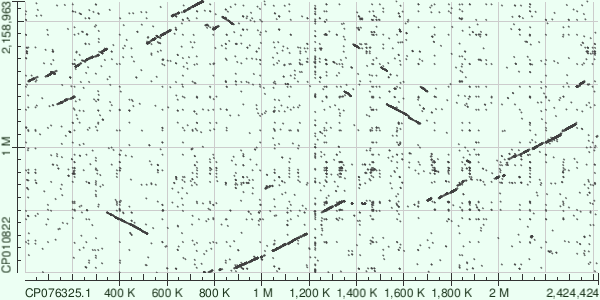
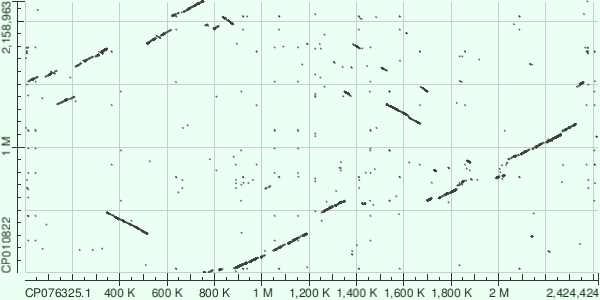
Тем не менее я решил рассмотреть эти организмы так как ну данной карте локального сходства можно увидеть большое количество мутаций. Далее я буду рассматривать карту сходства построенную с помощью blastn так как полученное выравнивание имеет сильно меньшее e-value.
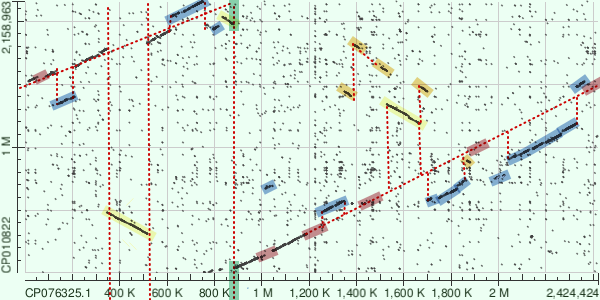
- Зеленым обощначен разрыв, который показывает что на кольцевой хромосоме были разные точки начала записи последовательности.
- Желтым обозначены участки с транспозицией и инверсией. Можно увидеть два крупных инвертированных гомологичных участка и 6 коротких.
- Красным обозначены неконсервативные участки которые оказались не выравнены.
- Синим обозначены участки с инсерциями и делециями.
Как можно заметить, несмотря на то что были рассмотрены два вида из одного рода, они имеют достаочно крупные геномные перестройки, что соотносется с тем что в дереве построенному по 16s rRNA Thermus antranikianii и Thermus aquaticus наъодятся достаточно далеко.