Практикум 14
Подготовка чтений
Для данного практикума использовалось чтение с Illumina для бактерии Buchnera aphidicola под кодом SRR4240356. Все дальнейшие действия проходили в дериктории /mnt/scratch/NGS/gorshkovleonid/
для загрузки чтений я использовал команду.
Сперва нужно убрать оставшиеся адаптеры из чтений с помощью программы Trimmomatic для чего был создан единый файл adapters.fa
После чего адаптеры удаляются из чтений.
Можно заметить что целых 2% ридов оказались адаптерами.
Затем с помощью этой же программы отсекаются чтения с качеством ниже 20 и длиной меньше 32 нуклеотидов.
Остеклось еще 4% чтений.
Изначальный файл с чтениями весил 174mb, после удаление чтений с адаптарами он стал весить 171mb, после выполнения последней команды 162 mb
Сборка генома с помощью velvetg
Далее из полученного файла trim_20.fq.gz с помощью программы velveth были получены все возможные k-меры длины 31.
На основе полученных k-меров с помощью программы velvetg была запущена сборка генома
Для данной сборки параметр N50 = 65554. С помощью следующей команды были получены 3 самых длинных контига.
| ID контига | Длина | Покрытие |
|---|---|---|
| 8 | 111962 | 38.66 |
| 6 | 107488 | 34.17 |
| 10 | 80939 | 37.52 |
С помощью следующей команды я нашел контиги с аномально высоким покрытием.
Не включая вырожденные контиги длины 1, аномально высокое покрытие имеют контиги под номерами 21(458.42), 47(453.26), 17(447.49), в скобках указан процент покрытия.
Следующей командой я разбил файл с контигами на отдельные файлы для каждого, предварительно создав директорию ./contigs
Анализ сборки
Далее 3 самых длинных контига были выравненны совместно с хромосомой Buchnera aphidicola (AC — CP009253) с помощью программы megablast. Пустые участки соответсвуют гэпам и инделям.
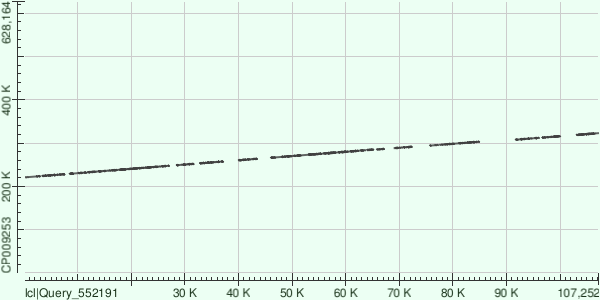
Файл с характеристиками полученного выравнивания. Можно видеть 18 комплементарных участков.
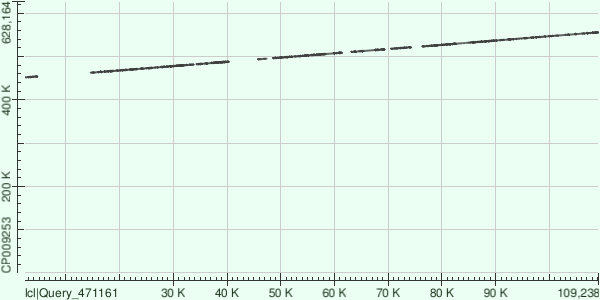
Файл с характеристиками полученного выравнивания. Можно видеть 15 комплементарных участков.
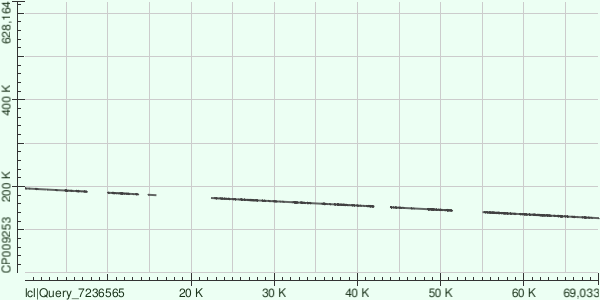
Файл с характеристиками полученного выравнивания. Можно видеть 10 комплементарных участков.