Практикум 3
Филогенетическая реконструкция и сравнение деревьев по нуклеотидным последовательностям
Построение дерева по 12S митохондриальной рРНК
1. Для организмов POEOC, DANRE, ANGIN, TETNG были найдены размеченные гены 12S рРНК длиной около 900 п.н. Для остальных организмов были найдены странные частичные последовательности длиной около 300 п.н., а для некоторых - всего 170 п.н., что показалось странным. Однако для всех этих организмов были доступны полные митохондриальные геномы длиной около 16 тыс. п.н., но средства автоматической аннотации почему-то не нашли в них нормально ген 12S рРНК. Поэтому я решил, помимо 4 генов именно 12S, добавить 6 полных митохондриальных геномов оставшихся организмов. Гэпы при построении дерева просто удалятся, так что я думаю, можно поступить таким образом.
2. Результаты выравнивания меня порадовали - несмотря на то, что оно проводилось по всем 16 килобазам, ген 12S рРНК однозначно хорошо выравнялся для всех организмов. Далее я на всякий случай с помощью скрипта обрезал все гэпы, чтобы никакие алгоритмы построения деревьев не смущались. Затем я построил дерево с помощью программы IQ-TREE.
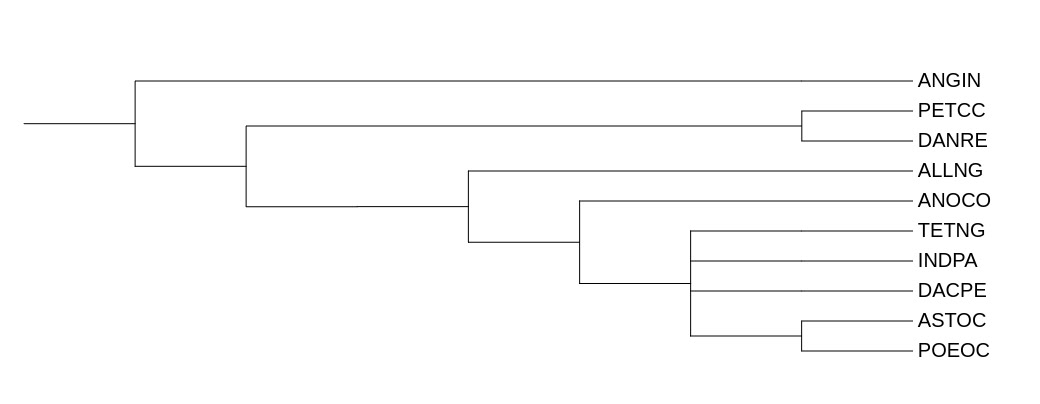

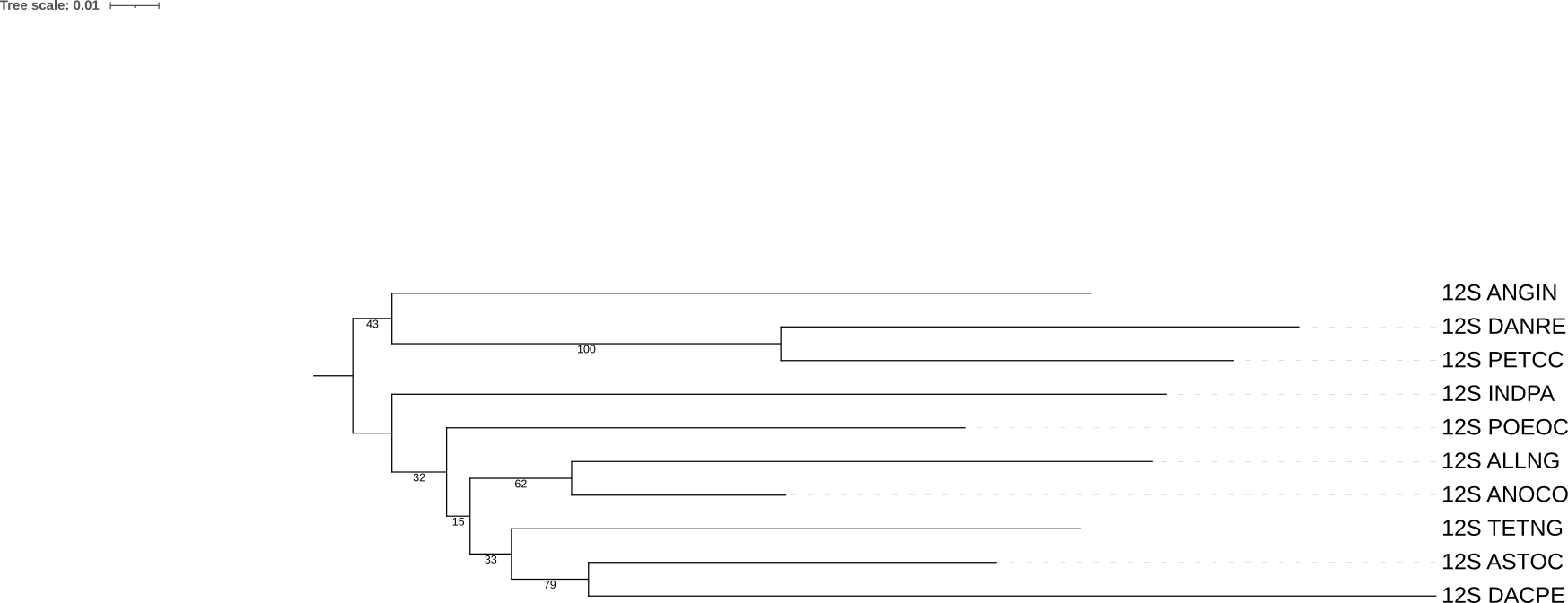
Укоренено оно в ANGIN, затем отходит ветвь сожержащая PETCC и DANRE, а далее идут остальные организмы чьл теперь полностью соответствует систематике. Ветвь содержащая INDPA ANOCO ALLGN POEOC, полностью неверна.
Ветвь содержащая TETNG ASTOC DACPE, хотя бы содержит организмы из одной неразрешенной ветви, хотя в исходном дереве DACPE и ASTOC не в одном узле, a иные алгоритмы в предыдущем практикуме по цитохрому b выделяли DACPE ASTOC POEOC, чего тут нет.Добавление аутгруппы
3. В качестве аутгруппы к Teleostei (Костистые рыбы) я выбрал Holostei (Костные ганоиды). Живых представителей осталось не так много, я решил взять панцирную щуку Lepisosteus oculatus (LEPOC). Дерево абсолютно верно укоренилось, сохранив топологию расположения ANGIN, (PETCC DANRE) и далее ветвь с остальными организмами. Опять же ветвь INDPA ANOCO ALLGN POEOC не верна и вся остальная топология предыдущего дерева без аутгруппы сохранилась.

Бутстреп
4. Здесь я решил добавить бутстреп-поддержку к дереву, построенному по 12S рРНК. Как можно видеть, с поддержкой 100 отделилась только ветвь с DANRE и PETCC, Что является единственной правильной ветвью. Остальная топология в сравнении с деревом из IQ-TREE хоть и поменялась верных ветвей больше не стало, но оно так же как и IQ-TREE выделело ветвь DACPE, ASTOCC и TETNG и показала относительно большую бутстреп поддержку.