Практикум 4
Ортологи и паралоги
Я выбрал следующие 7 протеомов бактерий: ECOLI, BRUSU, POLAQ, RHIME, SHEDO, BURMA, ROSDO.
Далее объединил их в один файл, создал базу данных для локального BLAST и запустил поиск по гену CLPX_ECOLI.

Далее я объединил все последовательности находок в один файл. Осуществил множественное выравнивание с помощью MUSCLE, построил дерево с помощью FastME (модель MtRev) с бутстреп-поддержкой ветвей.

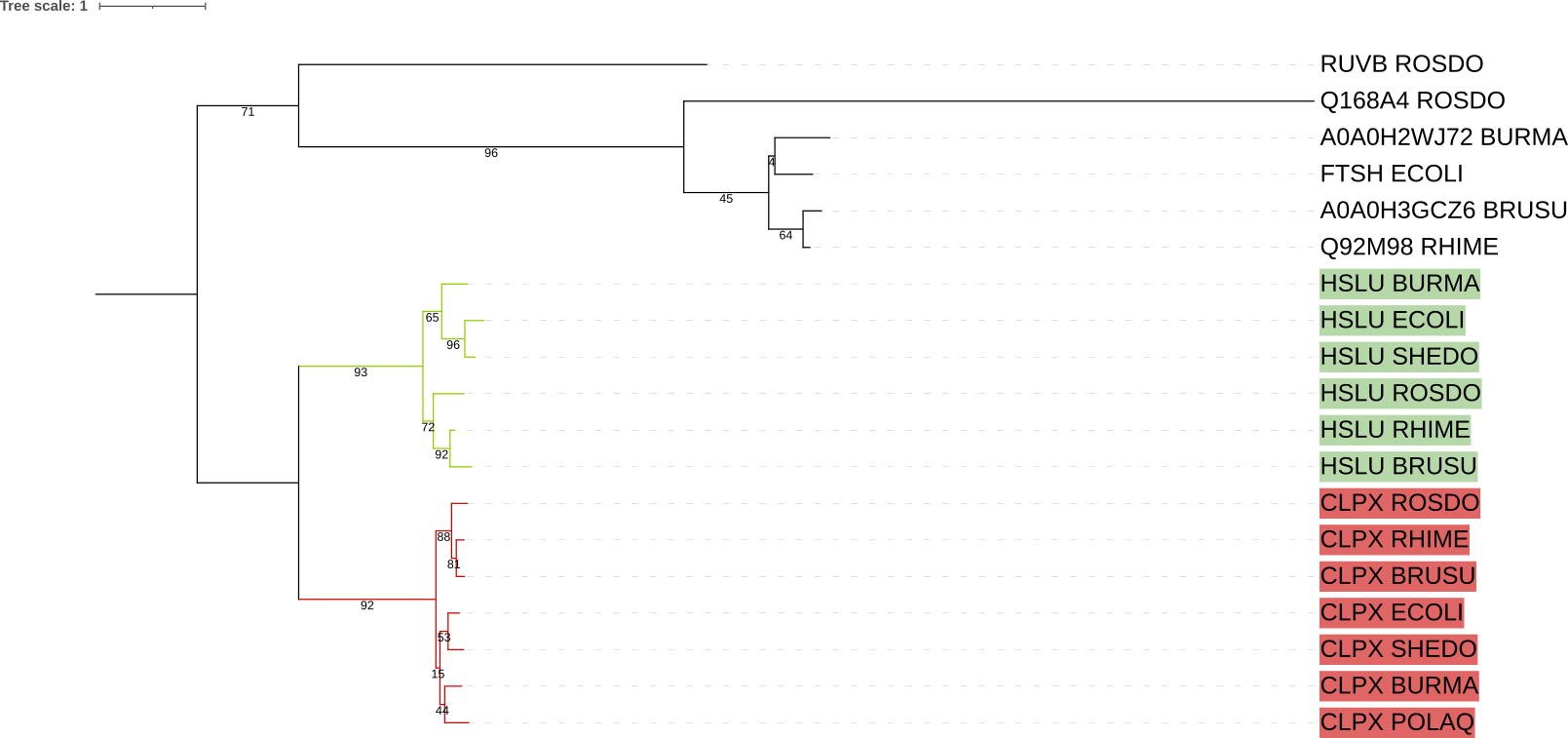
Как можно видеть на дереве, выделились 2 группы ортологов, соответственно CLPX - АТФ-связывающая субъединица АТФ-зависимой протеазы и HSLU - АТФазная субъединица АТФ-зависимой протеазы.
- Примерами пар ортологов соответственно являются: CLPX_POLAQ и CLPX_BURMA, HSLU_ECOLI и HSLU_SHEDO, A0A0H3GCZ6_BRUSU и Q92M98_RHIME.
- Примерами пар паралогов соответственно являются: HSLU_SHEDO и CLPX_SHEDO, HSLU_BRUSU и A0A0H3GCZ6_BRUSU, HSLU_BURMA и CLPX_BURMA.

На этом изображении ортологические группы были схлопнуты. В группе ортологов CLPX филогения полностью соответствует рекомендуемой. В группе HSLU топология расположения ROSDO, RHIME, BRUSU соответствует данному нам дереву, также соответствует расположение ECOLI, SHEDO, BURMA. Единственное, последовательность HSLU_POLAQ не попала в список находок BLASTp с пороговым E-value, поэтому она не оказалась в дереве, и группа является неполной.
Итого можно видеть, что в данном наборе белков и с использованными алгоритмами выравнивания и построения дерева, филогения не сильно отличается от исходной.