Практикум 8
Сигнал oriC
Для практического задания был выбран участок oriC — сайт инициации репликации в кольцевых хромосомах большинства бактерий. Его длина варьируется от ~250 до 2000 нуклеотидов, при этом первичная структура сильно различается между видами. Несмотря на вариабельность, сохраняются общие принципы организации: последовательность содержит множественные мотивы, распознаваемые белками, контролирующими начало репликации. В качестве модельного объекта использован штамм Escherichia coli K-12 MG1655, для которого функционально важные элементы oriC детально описаны и локализованы в интервале 3 923 767–3 923 998 н. о. [1]
Функционально участок можно разделить на три компонента:
- 1. Сайты связывания DnaA: пять высокоаффинных R-боксов (консенсус TTATCCACA) и три I-бокса с пониженной аффинностью. R-сайты связывают как ATP-, так и ADP-форму DnaA, тогда как I-сайты предпочтительно взаимодействуют только с ATP-DnaA. Олигомеризация ATP-DnaA на этих сайтах приводит к образованию ядра, компактизирующего ДНК и создающего положительное суперскручивание.
- 2. DNA-unwinding element (DUE): три тандемных 13-мерных повтора с консенсусом GATCTNTTNTTTT, богатые AT-парами. Термодинамическая нестабильность этого участка обеспечивает локальную денатурацию при механическом напряжении, генерируемом комплексом DnaA, и служит точкой входа для дальнейших компонентов репликационной вилки.
- 3. Сайты регуляторных белков: в E. coli белок IHF (integration host factor), связываясь с последовательностью ihF, индуцирует изгиб ДНК и способствует последовательному заполнению I-боксов DnaA. Белок FIS (factor for inversion stimulation), взаимодействуя со своим сайтом, подавляет преждевременную инициацию. Дополнительные факторы (SeqA, Cnu, Rob и др.) модулируют доступность oriC через метилирование и конформационные изменения.
Таким образом, oriC представляет собой многофакторный регуляторный модуль, точное распознавание которого белками-компонентами инициационного аппарата необходимо для своевременного и единичного запуска репликации. Нарушение структуры или экспрессии любого из адресатов сигнала снижает скорость роста и увеличивает частоту летальных событий, что подчеркивает высокую селективную значимость данного участка.

Я решил рассмотреть геном [3] Salmonella enterica, которая принадлежит к тому же семейству Enterobacteriaceae и является популярным модельным объектом. Координаты ориджина репликации не указаны, но я нашел ген mioC (4084179-4084622) и gidA (4081911-4083800), а судя по исследованиям это практически достоверный знак того, что координаты oriC будут в этом промежутке.

Поиск сигнала
Для нахождения ориджина репликации я использовал сервис Ori-Finder [4], который позволяет достаточно достоверно находить точку начала репликации прокариот, а также предоставляет различные средства визуализации результата.

В результате работы алгоритма можно видеть, он нашел два предполагаемых ориджина репликации, однако, исходя из предыдущих рассуждений, практически наверняка искомым является второй (4083801-4084178), длина ориджина — 378 нуклеотидов. Интересно, что сервис определил координаты mioC с точностью до нуклеотида, вероятно, потому что я взял популярный референсный геном.

Сервис строит Z-кривую исходя из GC, AT, MK, RY составов. Как можно видеть, два предполагаемых ориджина находятся в минимуме GC-состава, что согласуется с теорией.

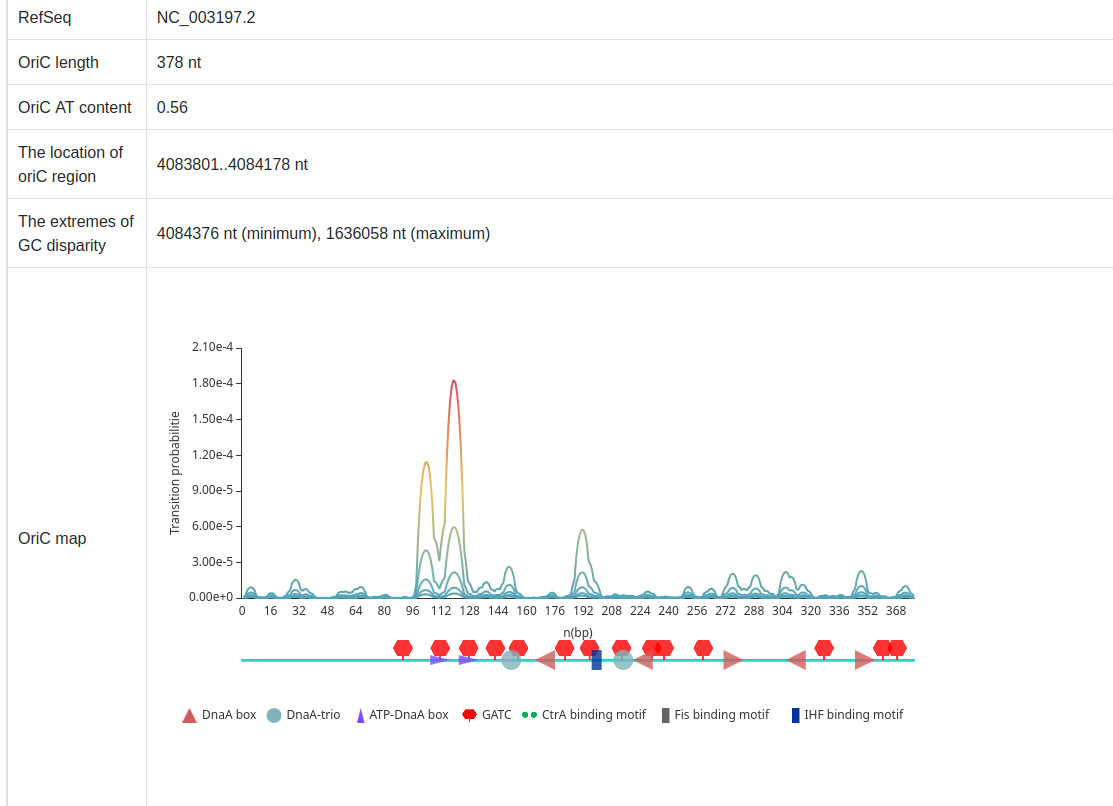
Выводы:
- Сервис Ori-Finder достаточно точно определил координаты oriC, что само по себе является нетривиальной биоинформатической задачей.
- Подтверждена гипотеза о локализации ориджина между генами gidA и mioC у Salmonella enterica.
- Наблюдаемое снижение GC-содержания в районе oriC коррелирует с AT-богатым DUE, описанным у E. coli, и служит дополнительным критерием достоверности предсказания.
Список литературы
- Jon M. K. Replication initiation at the Escherichia coli chromosomal origin. DOI: 10.1016/j.cbpa.2011.07.016.
- Ryo S. Kazutoshi K. Kenya M. A novel mode of DnaA–DnaA interaction promotes ADP dissociation for reactivation of replication initiation activity. DOI: 10.1093/nar/gkz795
- Complete genome sequence of Salmonella enterica serovar Typhimurium LT2.
- Mei-Jing D. Hao L. Feng G. Ori-Finder 2022: A Comprehensive Web Server for Prediction and Analysis of Bacterial Replication Origins. DOI: 10.1016/j.gpb.2022.10.002