Практикум 9
Поиск de novo сигналов в ДНК Acinetobacter calcoaceticus
В рамках данного практикума была предпринята попытка идентификации сигнальной последовательности Шайна–Дальгарно (SD), которая играет ключевую роль в инициации трансляции у прокариот, обеспечивая связывание рибосомы с мРНК. Объектом исследования стал геном бактерии Acinetobacter calcoaceticus, по которому ранее был подготовлен мини-обзор.
Для проведения анализа были получены нуклеотидная последовательность хромосомы в формате FASTA и соответствующая аннотация в формате GFF3 из базы данных NCBI. На основе этих данных с помощью «честно позаимствованного у Георгия Муравьёва» скрипта (который извлекает заданные участки генома на основании аннотации и формирует наборы последовательностей) были сформированы три группы:
- Положительный контроль (promotors.fasta) — последовательности по 100 нуклеотидов, предшествующих старт-кодону каждого CDS с учётом направления транскрипции (согласно литературным данным, SD-последовательность обычно располагается примерно за 10 нуклеотидов до старт-кодона).
- Отрицательный контроль (negative.fasta) — последовательности по 100 нуклеотидов, следующих непосредственно за старт-кодоном каждого CDS, с учётом ориентации.
- Группа для обучения (housekeeping.fasta) — подмножество последовательностей из положительного контроля, включающее гены высококонсервативных белков (таких как рибосомные белки, факторы транскрипции и трансляции), в которых наиболее вероятно присутствие SD-последовательности или её вариантов. Этот набор использовался для выявления потенциального паттерна и консенсуса SD в исследуемом организме.
Все группы представлены в виде FASTA-файлов с соответствующими последовательностями.
MEME
После подготовки данных производился анализ с помощью программы MEME, запущенной на сервере kodomo.
meme housekeeping.fasta -dna -nmotifs 3 -minw 6 -maxw 50
Ожидаемая длина мотива SD — 6–8 нуклеотидов; однако при таких параметрах у находок был ужасно высокий E-value. С верхней границей в 50 сильно лучше не стало, и никакие находки особенно не напоминают последовательность Шайна–Дальгарно.
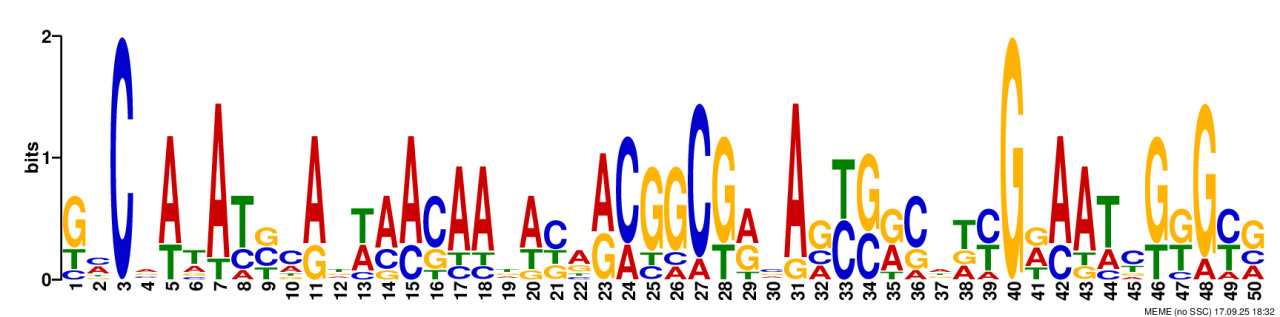
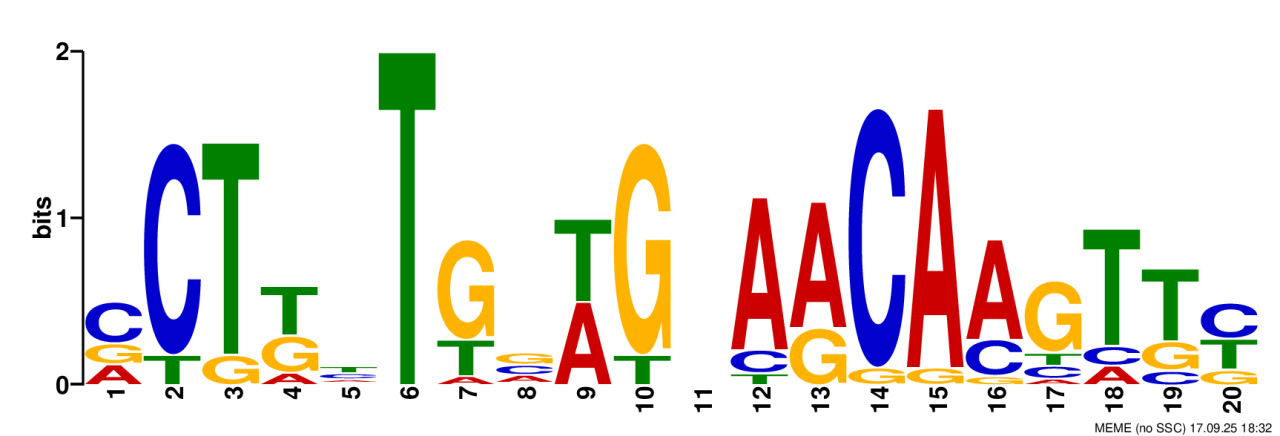
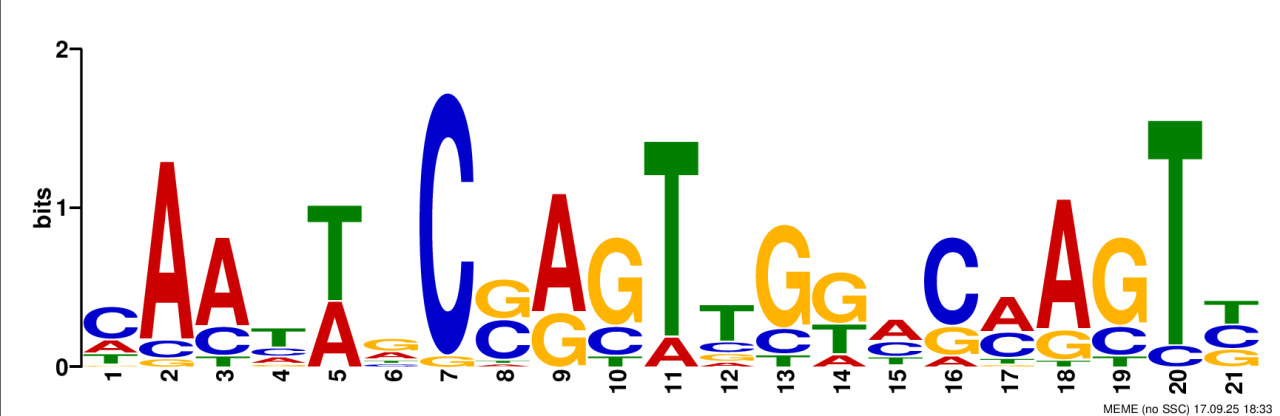
Для дальнейшего анализа я решил рассматривать 2-й мотив, так как он короче первого и чуть более «AG-богат», чем третий.
FIMO
Далее я провёл анализ встречаемости мотива с помощью программы FIMO на тестовой выборке и отрицательном контроле соответственно.
fimo --norc -motif VCTKHTGVWGNAACAAGTTY -thresh 0.001 meme.txt promotors.fasta
Отчёт программы в формате HTML
fimo --norc -motif VCTKHTGVWGNAACAAGTTY -thresh 0.001 meme.txt negative.fasta
Отчёт программы в формате HTML
Мотив является значимой находкой (p-value < 0.001) в 554 последовательностях и в 50 последовательностях отрицательного контроля, что ожидаемо. Это может говорить о том, что находка не совсем случайна: возможно, её часть «AACAAG» всё же является последовательностью Шайна–Дальгарно для данной бактерии.