Практикум 12. Blast
Задание 1
В первом задании осуществлялся поиск гомологов белка сульфидхинонредуктазы из бактерии Acidithiobacillus ferrooxidans ATCC 23270 в базе данных Swissprot. Ограничением по числу находок было выставлено на 20000, в связи с чем результатом поиска стали 18630 находок. Поиск осуществлялся по идентификатору белка (ACK80359.1) в не избыточной базе данных (non-redundant protein sequences, nr; в базе данных обьединены записи с идентичными последовательностями).
Таблица с результатами поиска.Для первого задания был выбран стандартный алгоритм blastp, осуществляющий сравнение белка из запроса с базой данных.
Ожидаемое число находок в модели со случайным подбором было выставлено на 10 (то есть, в случайно составленной базе данных моделей белка может быть найдено 10 белков с таким же или большим весом выравнивания при заданной длине слова; чем меньше значение этого параметра, тем больше будут ограничены результаты поиска). Длина слова для локальногго - 6.
Количество совпадений между исходной и искомыми последовательностями ограничено не было.
Матрицей для рассчета веса замен была выбрана BLOSUM62.
Штраф за гэп: 11; за продолжение инделя: 1.
Матрица корректирует работу в зависимости от аминокислотного состава.
Фильтр для маскировки участков низкой сложности, маски подключены не были.
Для дальнейшего рассмотрения было выбрано 10 предположительно гомологичных белков, информация о которых изложена в таблице 1.
| Белок | Идентификатор | Организм | E-value | Покрытие (%) |
|---|---|---|---|---|
| FAD-зависимая оксидоредуктаза | WP_062298346.1 | Nostoc piscinale | 4.17e-100 | 99 |
| Пиридин-нуклеотид-дисульфид-оксидоредуктаза | OGB41243.1 | Burkholderiales bacterium RIFOXYC2 | 0.0 | 98 |
| Пиридин-нуклеотид-дисульфид-оксидоредуктаза | HAP41495.1 | Nitrospira sp. | 4.94e-157 | 97 |
| Гипотетический белок AUH99 04515 | OLB52696.1 | Candidatus Rokubacteria bacterium | 5.44e-04 | 88 |
| Пиридин-нуклеотид-дисульфид-оксидоредуктаза | WP_002775238.1 | Leptonema illini | 1.53e-15 | 85 |
| NAD (P) / FAD-зависимая оксидоредуктаза | WP_016078151.1 | Bacillus cereus | 5.15e-10 | 83 |
| Гипотетический белок FOIG 15850 | EXL90950.1 | Fusarium oxysporum f. sp. cubense tropical race 4 54006 | 6.5 | 81 |
| Многофункциональная NAD(P)/FAD-зависимая оксидоредуктаза | WP_078292506.1 | Mycobacterium | 9.3 | 81 |
| NAD(P)/FAD-зависимая оксидоредуктаза | WP_011777241.1 | Paenarthrobacter aurescens | 1.23e-11 | 80 |
| NADH дегидрогеназа | KJF16986.1 | Acidithrix ferrooxidans | 5.69e-11 | 80 |
На основе выравнивания этих белков (ProbconsWS; скачать изображение с раскраской Cluster) было построено древо, представленное на рисунке 1.
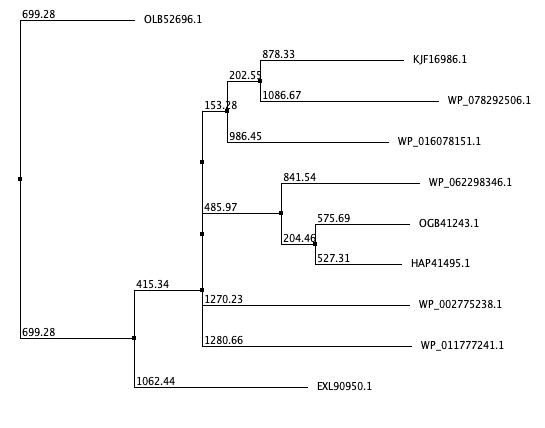
На этом дереве видно, что гипотетические белки (OLB52696.1 и EXL90950.1) сильнее всего выбиваются из подборки белков. В окончательном проекте они были удалены из выравнивания. Также исключены были белки WP_011777241.1 и WP_002775238.1.
Также из данного дерева становится ясно, что рассматриваемые белки можно разделить на две группы с высокой степенью гомологии.
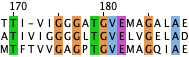
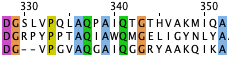
В первую группу вошли FAD-зависимая оксидоредуктаза (WP_062298346.1) и две Пиридин-нуклеотид-дисульфид-оксидоредуктазы (OGB41243.1 и HAP41495.1). В этой группе плотнее всего представлены консервативные позиции. На рисунках 2, 3, 4, 5 представлены фрагменты выравнивания с участками, доказывающими гомологию белков: начинаются и заканчиваются с абсолютно консервативной позиции, длиной более 6 колонок, без колонок с гэпами и высокой плотностью консервативных позиций.






Во вторую группу вошли NADH дегидрогеназа (KJF16986.1), FAD-зависимая оксидоредуктаза (WP_016078151.1) и многофункциональная NAD(P)/FAD-зависимая оксидоредуктаза (WP_078292506.1), что интересно, потому как в первой группе также состоит FAD-зависимая оксидоредуктаза. В данном выравнивании не так хорошо представлены участки, доказывающие гомологию между этими белками, но их все еще достаточно.


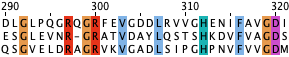

Во втором задании
Задание 2
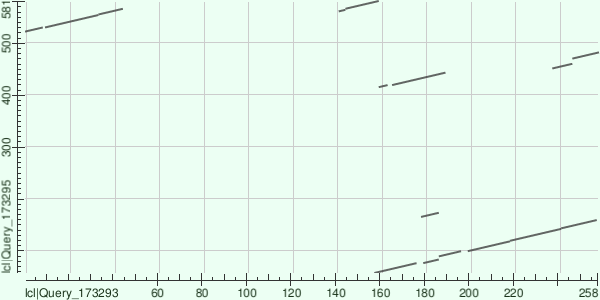
В рамках этого задания с помощью уже упомянутого выше blastp была построена карта локального сходства белков (не охарактеризованные белки из Ceriporiopsis subvermispora (strain B) (M2R9A4_CERS8) и Fomitopsis pinicola (strain FP-58527) (S8FKT8_FOMPI)), показанная на рисунке 8. По оси X отложен M2R9A4_CERS8 ("Х"), по оси Y - S8FKT8_FOMPI ("Y"). Стоит сказать, что в карте имеются наложения значительные наложения. Отчетливо видно, например, что в белке Х произошла транслокация участка 520…571 из Y в район 1…52. Участок белка Х 160-190 дублируется в белке Y, также есть крупный, немного разрывный участок совпадения в районе 140…258 остатков в белке Y и 1…200 - в X.
Задание 3
На первом этапе "игры" в поиск blastp, с параметрами, аналогичными первому заданию, вместо последовательности был внесен запрос "I AM NoT CoDiNg sequence". Результатом поиска стали 20000 последовательностей (из 20000 возможных), хотя программа заменила буквы, не соответсвующие ни одной из аминокислот, обозначением X - неизвестная аминокислота.

Без использования "Automatically adjust parameters for short input sequence" единственным результатом поиска было сообщение: "No significant similarity found". Программа так же заменила некоторые буквы на "Х".
В процессе "игры" мне в первую очередь захотелось проверить колличество находок в разных базах данных. Напомню, что в стандартном "Non-reduntant protein sequences" было 18630 находок с самыми разными значениями идентичности и E-value. При замене базы данных на "Reference protein" (референсные белки, записи о которых прошли проверку экспертов) количество находок сократилось до 13018, в том числе были исключены записи о некоторых из выбранных для второго задания белках. Последняя из показавшихся мне интересной баз данных - Model organisms. Она включает в себя протеомы 27 модельных организмов из самых различных таксономических групп. Эта база данных содержит очень разнообразный и не избыточный набор белков, поиск в ней может сыграть вспомогательную роль, например, в анализе распространенности белка в различных группах. В базе данных содержится 13 протеомов бактерий (в том числе E.coli), 2 протеома архей и 12 протеомов эукариотных организмов, в том числе человека, домовой мыши, некоторых грибов, растений и протист. В этой базе данных было найдено 24 записи. Покрытие варьируется от 96 до 9%, разброс значений E-value также достаточно велик: от 1e-83 до 6.0. В результатах поиска по-прежнему можно обнаружить различные дегидрогеназы и оксиредуктазы, неохарактеризованные белки. Интересным мне показался белок-фактор апоптоза из Glycine max (соя культурная), хотя покрытие с данным белком составляет всего 26%, значение E-value - 0.045.
Поскольку число результатов в избыточной базе данных составляет почти 19 тысяч результатов, что сильно затрудняет работу, в дальнейшем будет использоваться база референсных записей.
Следующий параметр, который я хотела бы рассмотреть - Expect threshold, определяющий порог статистической значимости. Значение этого параметра говорит о том, сколько белков с тем же или большим весом выравнивания будет найдено в случайно составленной базе данных. В таблице 2 вы можете видеть, как этот параметр влияет на число результатов. Ожидаемо, зависимость прямая: чем меньше значение, тем меньше результатов.
| Значение expect threshold | Число результатов |
|---|---|
| 1 | 10538 |
| 5 | 12219 |
| 10 | 13018 |
| 15 | 13492 |
| 20 | 13866 |
Эксперимент с изменением длины слова оказался малопоказателен, поиск с длиной 2 и 3 дал максимальный результат в 20000 результатов. Уменьшение длины слова увеличивает вероятность возможных совпадений, в данном случае до критических значений.
Пользуясь BLAST можно получить много полезной информации: найти гомологичные белковые последовательности, выделить консервативные участки, понять эволюционную взаимосвязь белков. Также можно сделать вывод о функциях и распространенности подобных рассматриваемому белков.