Практикум 10. Геномные браузеры
Задание 1
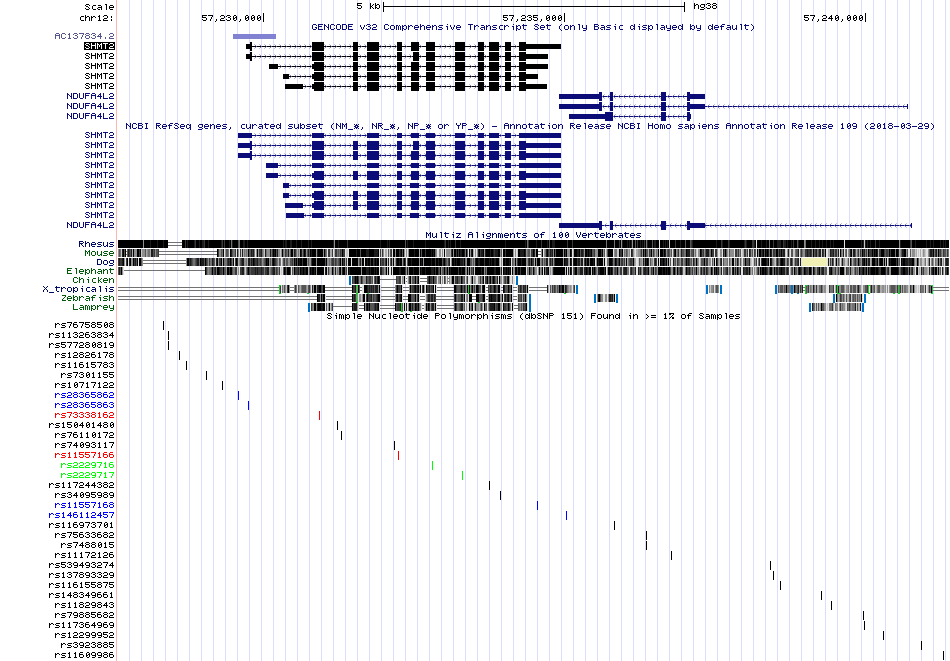
Для выполнения данного практикума я решила взять человеческую метилтрансферазу (угадайте, чем навеяно) SHMT2.
| Имя гена | SHMT2 |
| Идентификатор Gencode | ENSG00000182199 |
| Цепь | Прямая |
| Хромосома, координаты транскрипта | chr12:57,229,711-57,234,935 |
| Плечо и полоса | q13.3 |
| Число альтернативных транскриптов | 5* |
*Помимо 5 вариантов транскриптов самого SHMT2 на обратной цепи геномный браузер показал ген маркера чувствительности к лечению рака (AC137834.2) и три варианта митохонриального комплекса (NDUFA4L2).
Рассмотрим 3 произвольных транскрипта.
| Транскрипт | ID Gencode | Координаты | Число экзонов | Длина последовательности белка |
|---|---|---|---|---|
| variant 1 | ENST00000328923.8 | chr12:57,229,711-57,234,935 | 12 | 504aa |
| variant 2 | ENST00000557487.5 | chr12:57,229,711-57,234,726 | 12 | 494aa |
| variant 3 | ENST00000414700.7 | chr12:57,230,086-57,234,726 | 11 | 483aa |
![]()
С помощью геномного браузера Ensembl было получено выравнивание SHMT2 из H. sapiens и шимпанзе. Далее с помощью infoalign была получена информация, указанная в таблице 3 (сразу в форме html таблицы), в том числе - 'change'. Данная опция демонстрирует нам процент замен в последовательности, что и требовалось получить в задании. Можно сказать, что в среднем замена происходит через каждые 140 нуклеотидов.
| Организм | Длина последовательности | %identity | %Change |
|---|---|---|---|
| homo_sapiens_1-5364 | 5363 | 5363 | 0.018643 |
| pan_troglodytes_1-5364 | 5362 | 5326 | 0.708427 |
antropogenez.ru пишет, что геномы шимпанзе и человека отличаются всего на 1%, хотя у шимпанзе на одну пару хромосом больше. rationalwiki.org оценивает различия в 1,23%, хотя там представлено несколько вариантов сравнения: по однонуклеотидным заменам (результат упомянут ранее), замены и "процессы вставки и удаления", по числу идентичных белков, по "степени различия несинонимических пар оснований в белок-кодирующих районах". Все эти цифры сводятся к различию в 1-2%. Беглый осмотр остальных результатов гуглинга показал, что все оценки держатся в этом районе.
Задание 3
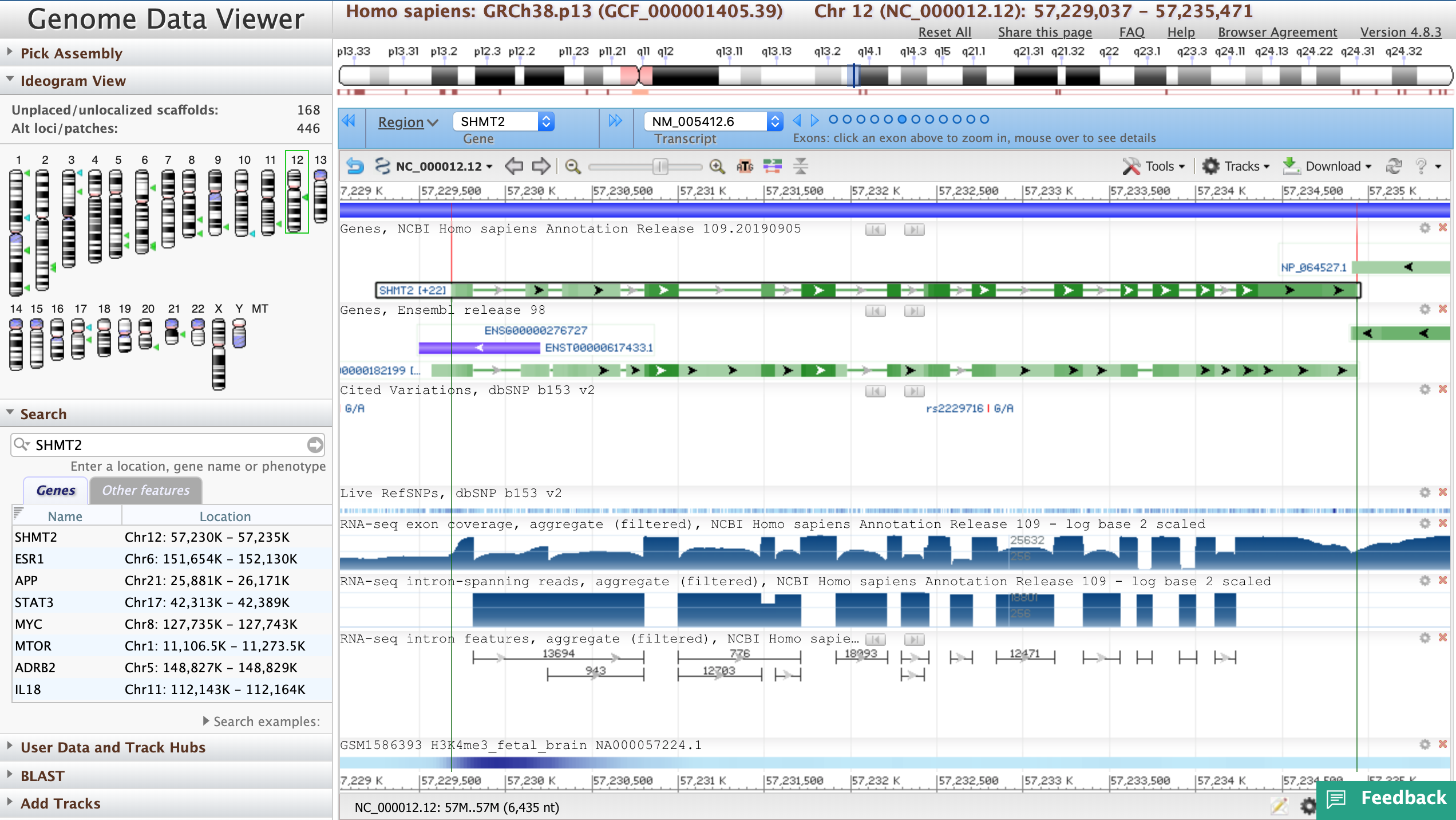
Первое, что бросается в глаза при открыти геномного браузера NCBI - тот факт, что для получения дополнительной информациии о транскриптах, о самом геноме не нужно открывать дополнительные вкладки. Здесь более ясная и удобная навигация по хромосомам - слева есть небольшая карта всех существующих, выше - рассматриваемой (но это есть и в других браузерах). Есть возможность выделить и рассмотреть отдельный экзом.
При поиске конкретного гена на выдачу в маленьком окошке браузер дает список совпадений (сразу с координатами), по которым можно без дополнительных подгрузок переместиться в нужную часть генома. Также эти совпадения отмечаются на уже упомянутой хромосомной идеограмме.
Наличие треков, как и в других браузерах, регулирутеся с помощью специального меню. По умолчанию демонстрируется покрытие выравниваний мРНК-экзонов, покрытие интронов, полученное из сплайсированных выравниваний мРНК, а также "intron features". Стоит сказать, что подсказки по трекам всплывают при наведении курсора (чего нет в Gencode и Ensembl). В списке треков присутствует "Epigenomics", где, насколько я поняла, в разных формах (heat map, например) можно рассмотреть последовательность из образца конкретной ткани, в том числе эмбриональной. А еще, насколько я поняла, здесь есть возможность создавать свои наборы треков.
В целом хочу сказать, что геномный браузер NCBI показался мне достаточно мобильным и интуитивным, хотя тот Gencode в целом способен с ним конкурировать. NCBI genome data viewer, по моим ощущениям, совмещает в себе многие плюсы Gencode и Ensembl, но я не уверена, что он способен полностью их заменить.