Практикум 11. Ресеквенирование. Поиск полиморфизмов у человека
Задание
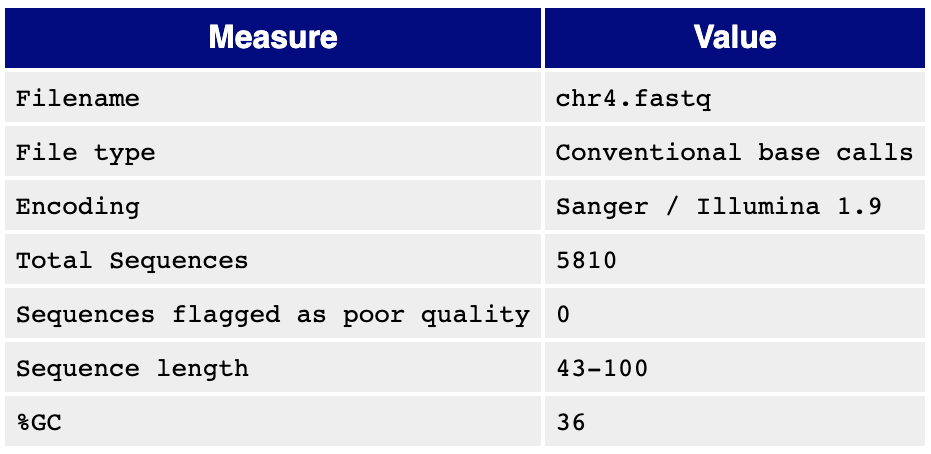
Для выполнения данного задания мне была определена 4 хромосома. Исходное число чтений - 5810
| Команда | Назначение |
hisat2-build chr4.fasta indexed | Индексация RS |
fastqc chr4.fastq | Анализ качества последовательности |
ava -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr4.fastq chr4_trim.fastq TRAILING:20 MINLEN:50 | Очистка: обрезание ридов с качеством ниже 20 и длиной ниже 50 |
fastqc chr4_trim.fastq | Анализ качества после очистки |
hisat2 -x indexed -U chr4_trim.fastq -S chr4_align.sam --no-softclip --no-spliced-alignment | Картирование ридов на RS; параметры требуют выравнивание по всему чтению и отсутсвие гэпов/инделей внутри рида |
samtools view -b chr4_align.sam -o chr4_align.bam | Трансформация в бинарный формат |
samtools sort chr4_align.bam sorted_chr4_align | Сортировка выравниваний по координате RS |
samtools index sorted_chr4_align.bam | Индексирование выравнивания |
samtools flagstat sorted_chr4_align.bam > sorted_chr4_align_quality.txt | Дополнительная информация о выравнивании |
amtools mpileup -uf chr4.fasta -o chr4_polmh.bcf sorted_chr4_align.bam | Вычисление полиморфизмов |
bcftools call -cv -o chr4_polmh.vcf chr4_polmh.bcf | Трансформация в необходимый формат |
vcftools --vcf chr4_polmh.vcf --remove-indels --recode --out delindel_chr4_polmh | Удаление инделей |
convert2annovar.pl -format vcf4 delindel_chr4_polmh.recode.vcf -outfile chr4_polmh.avinput | Перевод в доступный annovar формат |
annotate_variation.pl -out refgene_chr4 -build hg19 -dbtype refGene chr4_polmh.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по refGene |
annotate_variation.pl -filter -out dbsnp_chr4 -build hg19 -dbtype snp138 chr4_polmh.avinput /nfs/srv/databases/annovar/humandb.old/ | Dbsnp |
annotate_variation.pl -filter -dbtype 1000g2014oct_all -buildver hg19 -out 1000g_chr4 chr4_polmh.avinput /nfs/srv/databases/annovar/humandb.old/ | 1000 genomes |
annotate_variation.pl -regionanno -build hg19 -out gwas_chr4 -dbtype gwasCatalog chr4_polmh.avinput /nfs/srv/databases/annovar/humandb.old/ | Gwas |
annotate_variation.pl -filter -dbtype clinvar_20150629 -buildver hg19 -out clinvar_chr4 chr4_polmh.avinput /nfs/srv/databases/annovar/humandb.old/ | Clinvar |
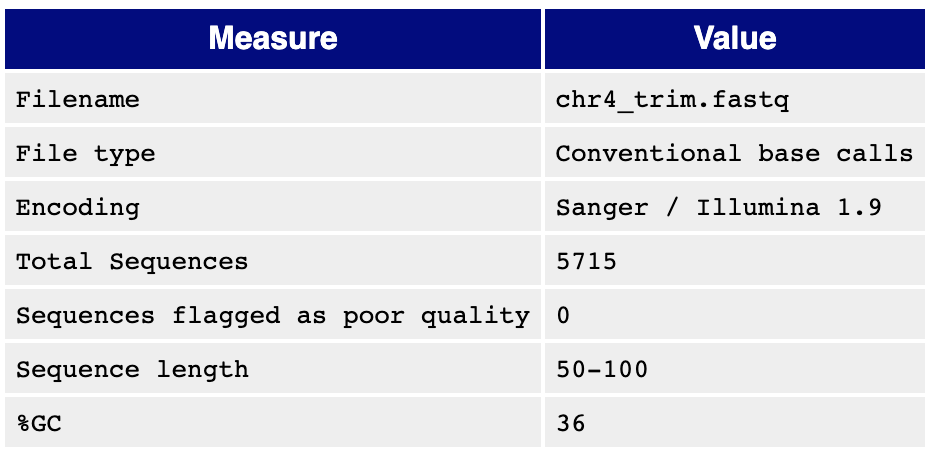
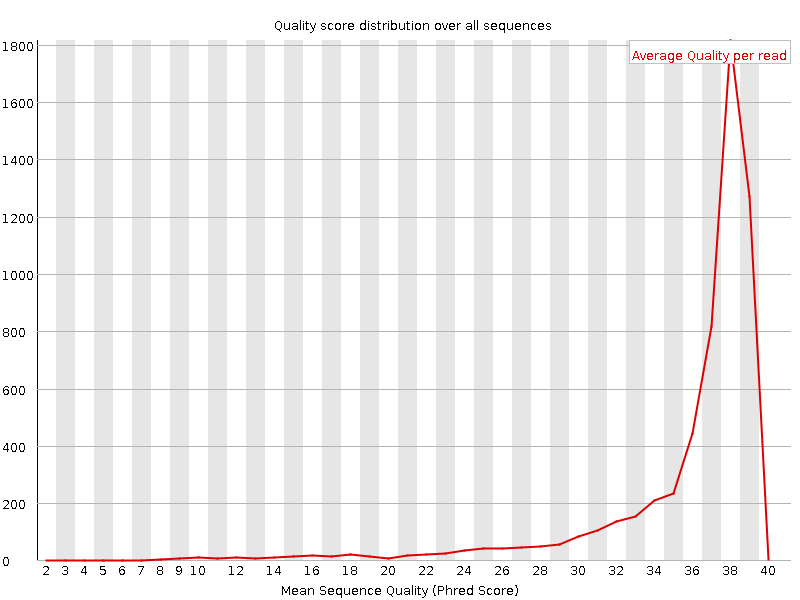
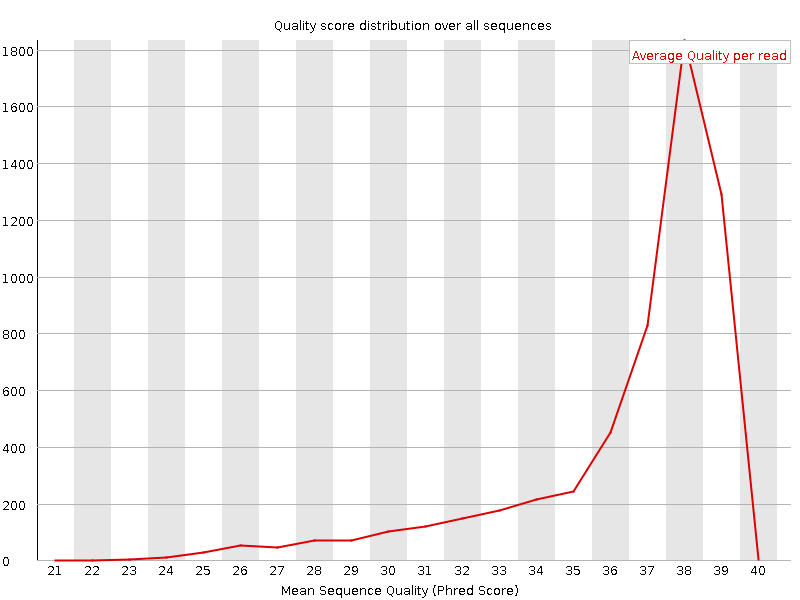
Если взглянуть на скриншоты таблиц (рис. 1, 2), характеризующих последовательности, а также на графики (рис. 3, 4, 5, 6), характеризующие качество "per base" и "per seq", можно сделать вывод, что качество данных последовательностей достаточно хорошее. Параметры, в том числе насыщенность GC-парами и число самих ридов, изменились незначительно (было удалено всего 1,6 процента последовательностей - судя по всему, из-за длины). "Триммирование" в данном случае не было обязательной процедурой.
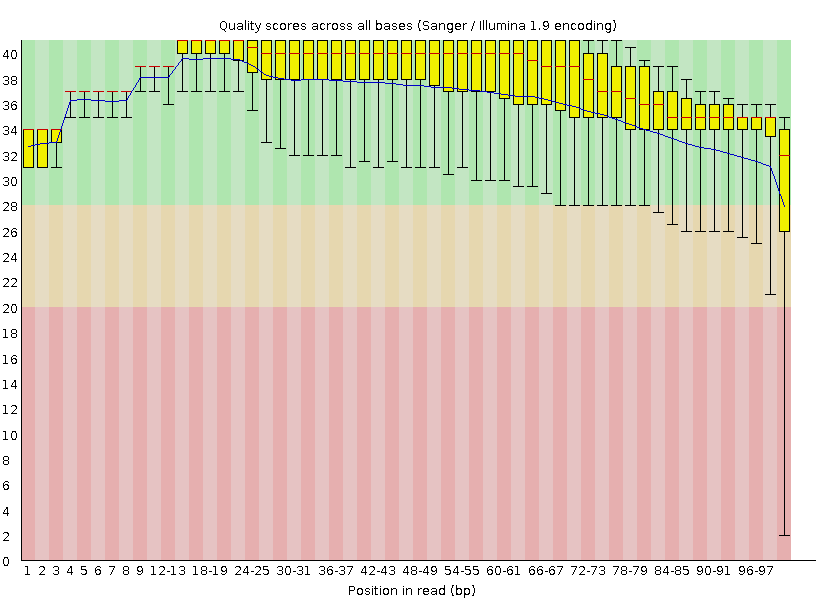
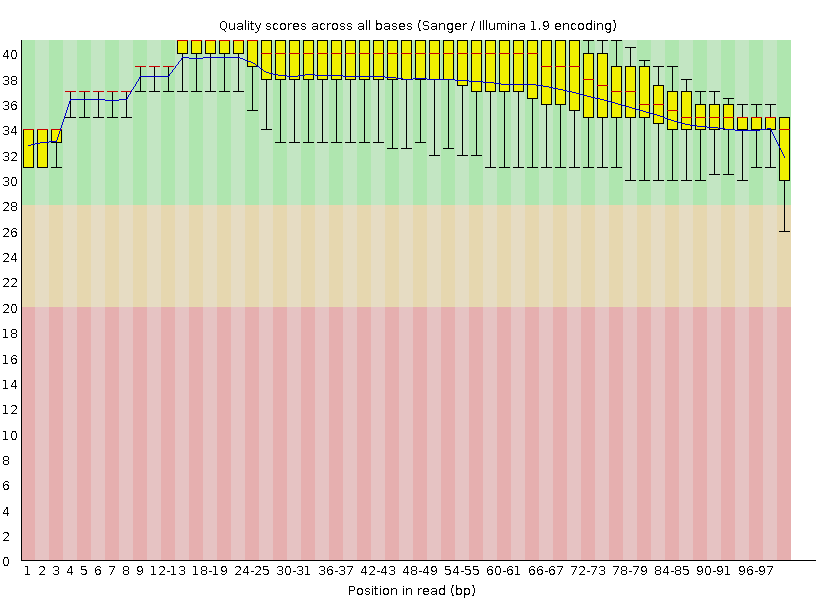
Выдача программы hisat2 говорит мне о том, что всего 19 последовательностей не было уложено на референс, всего для одной нашлось более одного участка схожести. В итоге, более 99,6% ридов однозначно картированы на референс. Выглядит, как очень хорошее качество картирования.
5715 reads; of these:
5715 (100.00%) were unpaired; of these:
19 (0.33%) aligned 0 times
5695 (99.65%) aligned exactly 1 time
1 (0.02%) aligned >1 times
99.67% overall alignment rate
В таблице 2 приведена информация о 3 избранных полиморфизмах. Всего их было найдено 49, 45 из них - SNP. Среднее Quality равно 92,18, медиана - 61,97. Средняя глубина равна 20, медианная - 5.
| Координаты | Референс | Чтение | Глубина | Качество | Тип |
| 187158034 | G | A | 83 | 221.999 | SNP |
| 187171355 | A | G | 49 | 221.999 | SNP |
| 88760642 | AAGAGA | AAGAGAGA | 16 | 81.4666 | INDEL |
С помощью результатов выдачи refgene можно рассмотреть представленные здесь виды SNP: 36 intronic, 2 downstream, 3 intergenic и 3 exonic. Также с помощью refgene мы видим, что всего SNP попали в три экзона KLKB1, и только одна замена оказалась синонимичной.
line30 nonsynonymous SNV KLKB1:NM_000892:exon5:c.G428A:p.S143N, chr4 187158034 187158034 G A hom 221.999 .
line38 nonsynonymous SNV KLKB1:NM_000892:exon10:c.A1072G:p.T358A, chr4 187172943 187172943 A G het 225.009 .
line44 synonymous SNV KLKB1:NM_000892:exon15:c.T1761C:p.N587N, chr4 187179210 187179210 T C het 225.009 .
RS имеют 41 SNP (данные из dbsnp)
Исходя из данных 1000genomes, средняя частота равна 39,7%
Выдача Gwas (как и clinvar, но более ясно) дает нам некоторую клиническую картину:
gwasCatalog Name=Parkinson's disease chr4 68447249 68447249 A G het 176.009 .
gwasCatalog Name=Cardiovascular disease risk factors chr4 88755828 88755828 T C hom 221.999 .
gwasCatalog Name=Metabolite levels chr4 187149540 187149540 G A hom 212.999 .
gwasCatalog Name=Obesity-related traits chr4 187158034 187158034 G A hom 221.999 .
Данная выдача говорит нам о том, что четыре полиморфизма предположительно связаны с болезнью Паркинсона, факторами риска сердечно-сосудистых заболеваний, могут повлиять на уровень метаболизма, а также оказать влияние на склонность к ожирению соответственно. Насколько я понимаю, данные варианты были зафиксированы в статистически значимом количестве у людей с данными отклонениями, но это не является гарантией проявления данных признаков.
Clinvar описал всего один SNP как "pathogenic" и соотнес его с дефицитом прекалликреина - так называемого фактора Флетчера - также имеет отношение к заболеваниям сердечно-сосудистой системы (фактор принимает участие в процессе свертывания крови), хотя нередко данное состояние не имеет значения для клинической картины, то есть, не влияет на здоровье пациента. Интересно, что этот SNP присутсвует также в выдаче Gwas и имеет схожее "предсказание".
CLINSIG=pathogenic;CLNDBN=Prekallikrein_deficiency;CLNREVSTAT=no_assertion_criteria_provided;CLNACC=RCV000012817.24;CLNDSDB=MedGen:OMIM:SNOMED_CT;CLNDSDBID=C0272339:612423:48976006
chr4 187158034 187158034 G A hom 221.999