Практикум 8. Поиск по сходству (BLAST)
Задание 1
Для выполнения этого задания был выбран алгоритм megablast, лучше всего подходящий для поиска близких гомологов. По этой ссылке вы можете скачать hit table для данной сессии. Далее будем рассматривать первые 20 результатов поиска, поскольку их E-value равняется нулю, а % identity >= 90. Для начала были выбраны стандартные параметры поиска (рис. 1), которые оказались оптимальными для данной работы. Все 20 находок принадлежат разным подвидам моллюска Lacuna vincta, сами находки представляют из себя субъединицу 1 цитохромоксидазы (COI) из митохондриального генома (parcial cds).
Цитохромоксидаза является терминальной оксидазой аэробной дыхательной цепи переноса электронов, которая катализирует перенос электронов с цитохрома с на кислород с образованием воды.
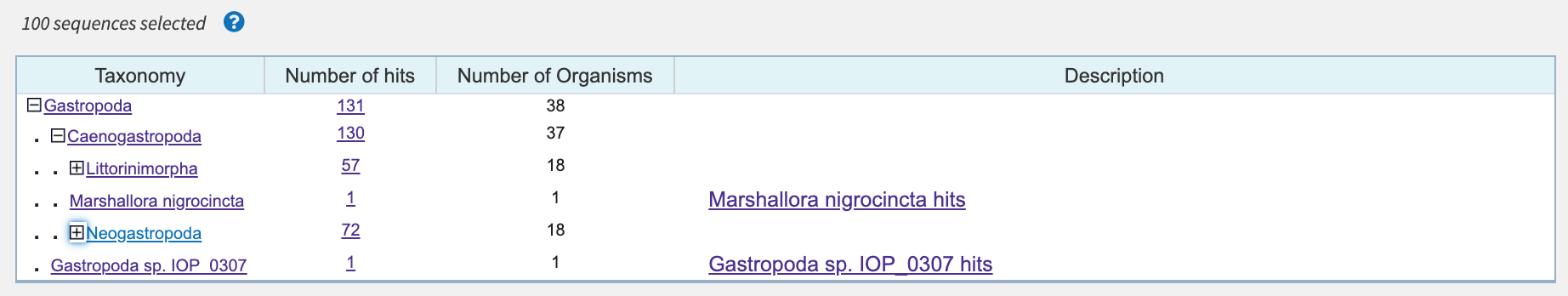
Зайдя во вкладку BLAST'а "Taxonomy", мы увидим, что все находки (131) принадлежат классу Gastropoda, 130 - представители подкласса Caenogastropoda (1 принадлежит некоему Gastropoda sp. IOP_0307). 57 находок принадлежат порядку Littorinimorpha, еще 71 - Neogastropoda. Учитывая, что 20 лучших по всем параметрам находок принадлежат Lacuna vincta, можно с уверенностью говорить о принадлежности белка данного организма порядку Littorinimorpha, а также предположить принадлежность роду Lacuna.
Белки-цитохромоксидазы достаточно сильно консервативны, о чем нам говорит множественное выравнивание 100 лучших находок этой сессии megablast с консенсусной последовательностью. Дерево белков, построенное Jalview, определяет его в одну ветку с уже упомянутым Lacuna vincta. Из этого по прежнему нельзя сделать вывод о том, что белок достоверно принадлежит данному моллюску, но по крайней мере это можно предположить.
Задание 2
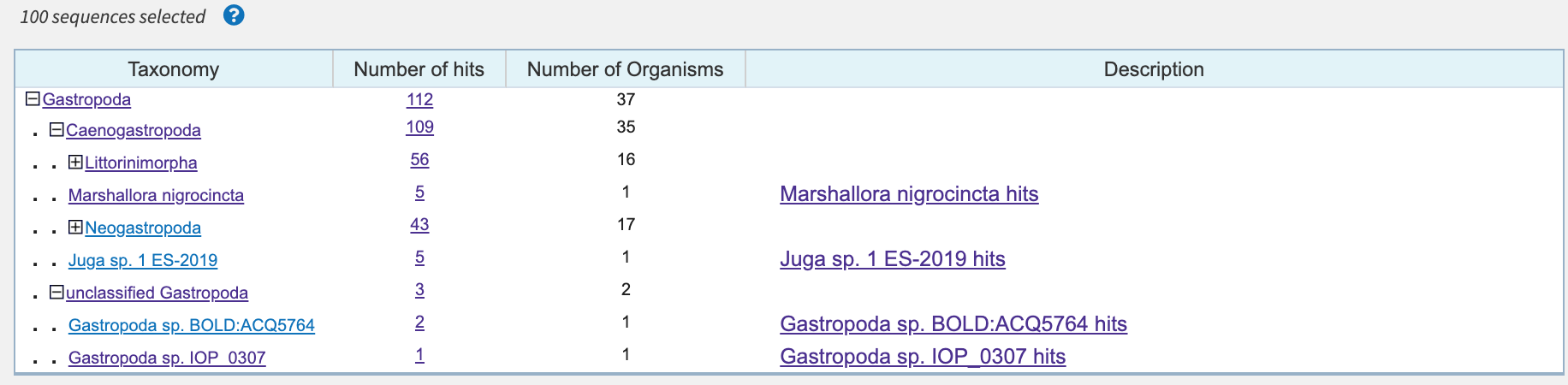
Сравнивая результаты поиска в разных программах, стоит сказать, что blastn с более строгими настройками сработал намного быстрее (менее 30 с против 3 минут работы других программ). Начнем сравнение с цитохромоксидазы. Визуально первые строчки поиска почти не различаются, для сравнения были вычислены средние значения %identity, длины выравниваний и E-value (таблица 1). Как видно из таблицы, с ужесточением параметров растет средняя длина выравнивания, уменьшается средний E-value (эти два пункта не касаются анализа вирусной последовательности), но падает средний процент идентичности. Раздел "Taxonomy" изменений почти не претерпел (рис. 2, 3, 4).
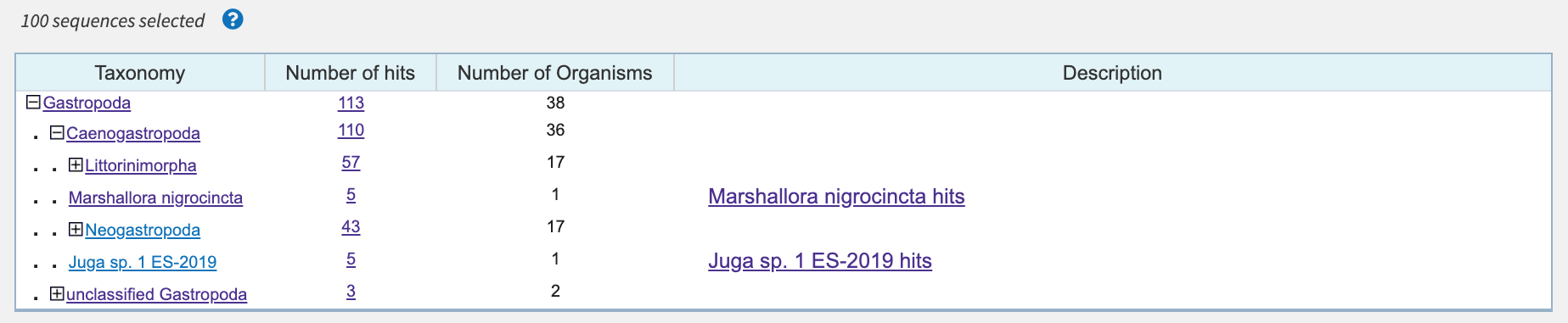
Что касается вируса из предыдущего практикума (Myoviridae sp. isolate ctgg12), результат куда более интересный. Был взят ген integrace, с сайта была скачана его последовательность. Megablast для нее предложил всего одну находку - собственно сама последовательность из генома, тогда как в blastn и strict blastn было 20 и 32 находки соотвественно. Средние значения %identity, длины выравниваний и E-value также указаны в таблице 1, но самое любопытное лежит во вкладке Taxonomy (рис. 5, 6). Помимо находки из самого генома, эти алгоритмы предлагают некоторое количество билатеральных животных, червей, а стандартный blastn даже указал на растение из семейства Мальвовых.
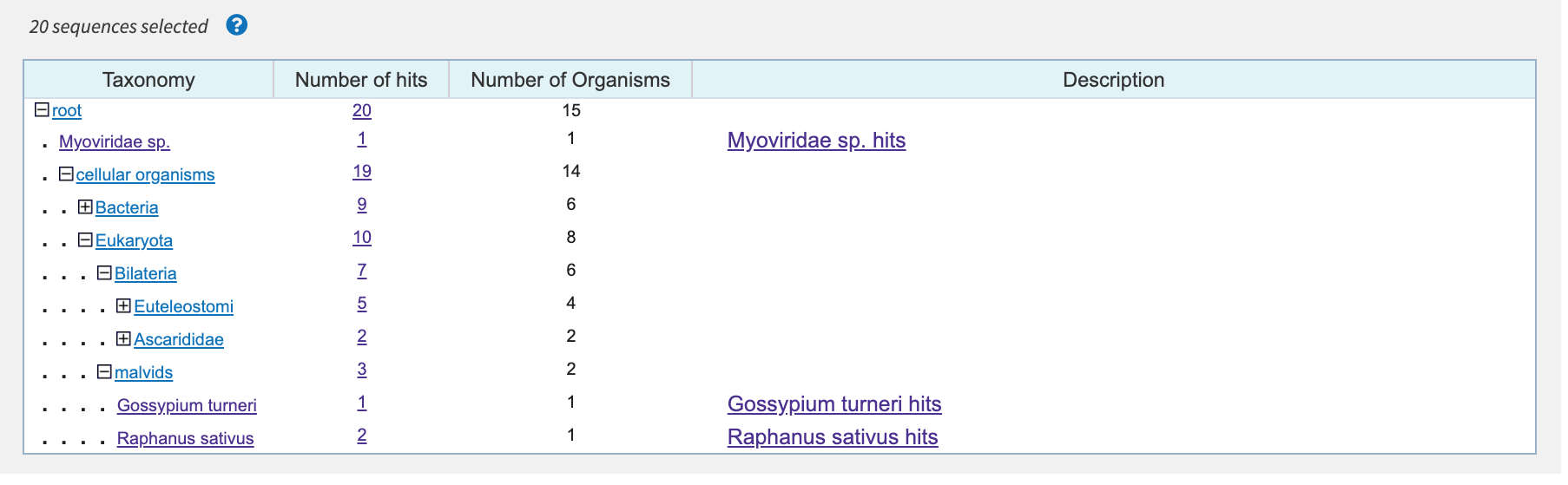
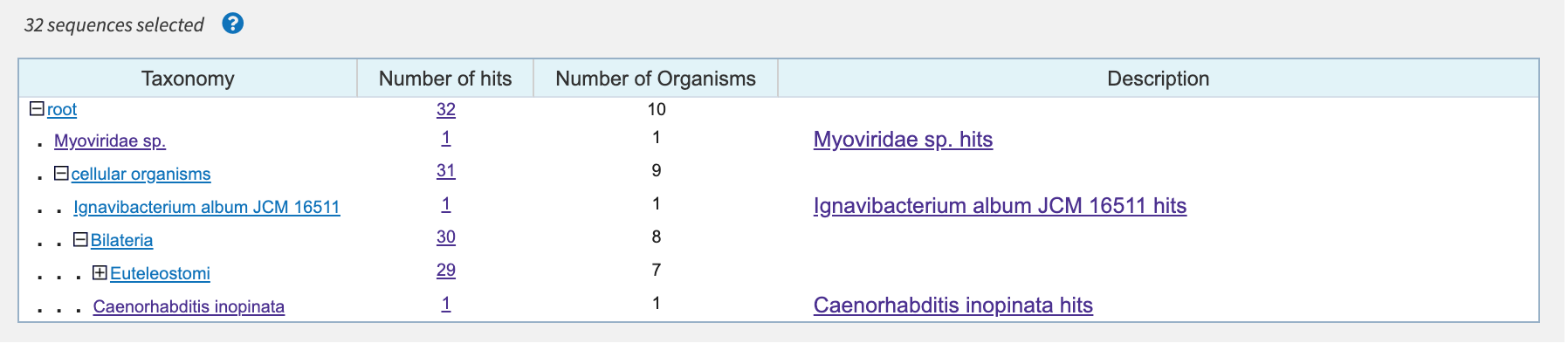
По данным ссылкам вы можете скачать exel-таблицы с hit tables вышеупомянутых сессий поиска для моллюска и вируса.
| Программа | Средний %identity | Средняя длина выравнивания | Средний E-value |
|---|---|---|---|
| Цитохромоксидаза (см. задание 1) | |||
| megablast | 86,44104 | 501,06 | 1,6856E-133 |
| blastn | 84,05986 | 551,39 | 6,462E-153 |
| Strict blastn | 83,66062 | 558,29 | 1,0711E-161 |
| CDS из вируса (integrase) | |||
| megablast | 100 | 1158 | 0 |
| blastn | 91,33165 | 90,85 | 2,693 |
| Strict blastn | 90,8098125 | 67,59375 | 4,6390625 |
Задание 3
Для данного задания я взяла ДНК-зависимую лигазу (Saccharomyces cerevisiae, Q08387), гликоген-расщепляющий фермент (Saccharomyces cerevisiae, Q06625) и АТФ-зависимую хеликазу SGS1 (Saccharomyces cerevisiae, P35187), посчитав, что гомологи этих белков должны быть достаточно сильно распространены среди эукариотических организмов. Данные последовательности были найдены в Uniprot по ключевым словам (например, helicase).
Поскольку перед нами стоит задача поиска белков в геноме Amoeboaphelidium protococcarum, для работы я выбрала программу tblastn, предварительно создав базу данных из генома с помощью следующей команды:
makeblastdb -in *.fasta -dbtype nucl -parse_seqids -out Amoeboaphelidium -title "Amoeboaphelidium protococcarum"
Далее я запустила поиск трех упомянутых белков против этогот генома. Использовалась стандартная таблица генетического кода, хотя всего существует порядка 30 вариантов для различных задач. Например, table 11 является стандартной таблицей для бактерий и архей, а 3 - для митохондриального генома дрожжей. Формат вывода - табулированная таблица.
tblastn -db Amoeboaphelidium -query ligase.fasta -outfmt 7 -out ligase
Результаты поиска были собраны в excel-таблицу, важные строчки были окрашены. По-прежнему считая главными критериями длину выравнивания, % identity, E-value и длину исходных белков, можно сказать, что для хеликазы есть два более-менее достоверных результата со схожими значениями. Судя по координатам находок, их длинам и значениям параметров, можно сказать, что это части одного белка, попавшие в два соседних контига. Для лигазы таковых 4 - их можно разбить на две группы по 2 находки. Эти пары имеют схожие значения, но расположены в контигах с достаточно далекими номерами (100, 243 и 423 и 1071). Судя по q. start и q. end, это не части одного гена, а определенные домены, найденные в разных участках генома. Для гидролазы есть 4 достоверных результата с хорошими значениями. Их опять можно разбить на две группы, но, в отличие от лигазы, q. start и q. end говорят нам о том, что это части одного гена, причем обе его части находятся в одном скаффлоде. Между предполагаемыми частями есть небольшие "пустые" участки. Их присутствие можно обьяснить особенностями найденных гомологов или наличием в геноме интронного участка. Значения s. end и s. start всех четырех находок дают повод считать, что в данном геноме есть два гомолога данного белка.
Задание 4
Поскольку геном тихоходки оказался уже аннотирован, было принято решение использовать один из контигов (000086F) сборки Arabidopsis thaliana (CABPTJ010000062.1).
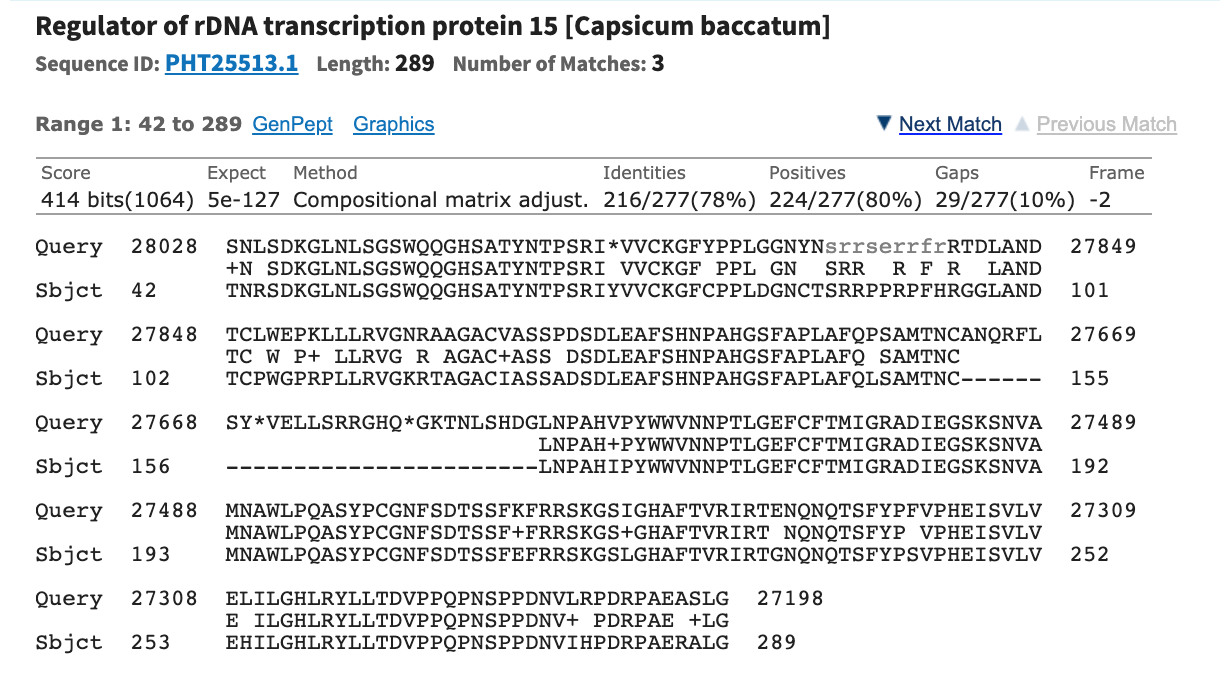
Для поиска белков в контиге был запущен поиск blastx с немного ослабленными параметрами (expect = 15). Среди большого числа неохарактеризованных и гипотетических белков было несколько находок Regulator of rDNA transcription protein 15 [Capsicum baccatum].