Практикум 4. Паралоги, визуализация
Составление списка гомологов
С помощью поиска blasn c ограничением по e-value 0.001 былb обнаружены следующие белковые последовательности: CLPX_CLOTE, CLPX_GEOKA, CLPX_BACSU, CLPX_LISMO, CLPX_STAA8, Q1GAP8_LACDA, CLPY_BACSU, CLPY_BACSU, HSLU_GEOKA, HSLU_GEOKA, HSLU_LACDA, HSLU_LACDA, HSLU_LISMO, HSLU_LISMO, HSLU_STAA8, HSLU_STAA8, Q8Y8B1_LISMO, CLPL_STAA8, CLPE_BACSU, Q1GB74_LACDA, M4YZ72_STREQ, M4YWY5_STREQ, M4YVJ8_STREQ, Q891B9_CLOTE, Q899H3_CLOTE, Q1GBN8_LACDA, Q1G869_LACDA, Q5L436_GEOKA, Q5KUR3_GEOKA, Y1413_STAA8, Q895L6_CLOTE, Q8YAC6_LISMO, Q898D1_CLOTE, Q5L3T1_GEOKA, Q5KZ67_GEOKA, FTSH_BACSU
Результаты выдачи в табличной форме.
Реконструкция и визуализация
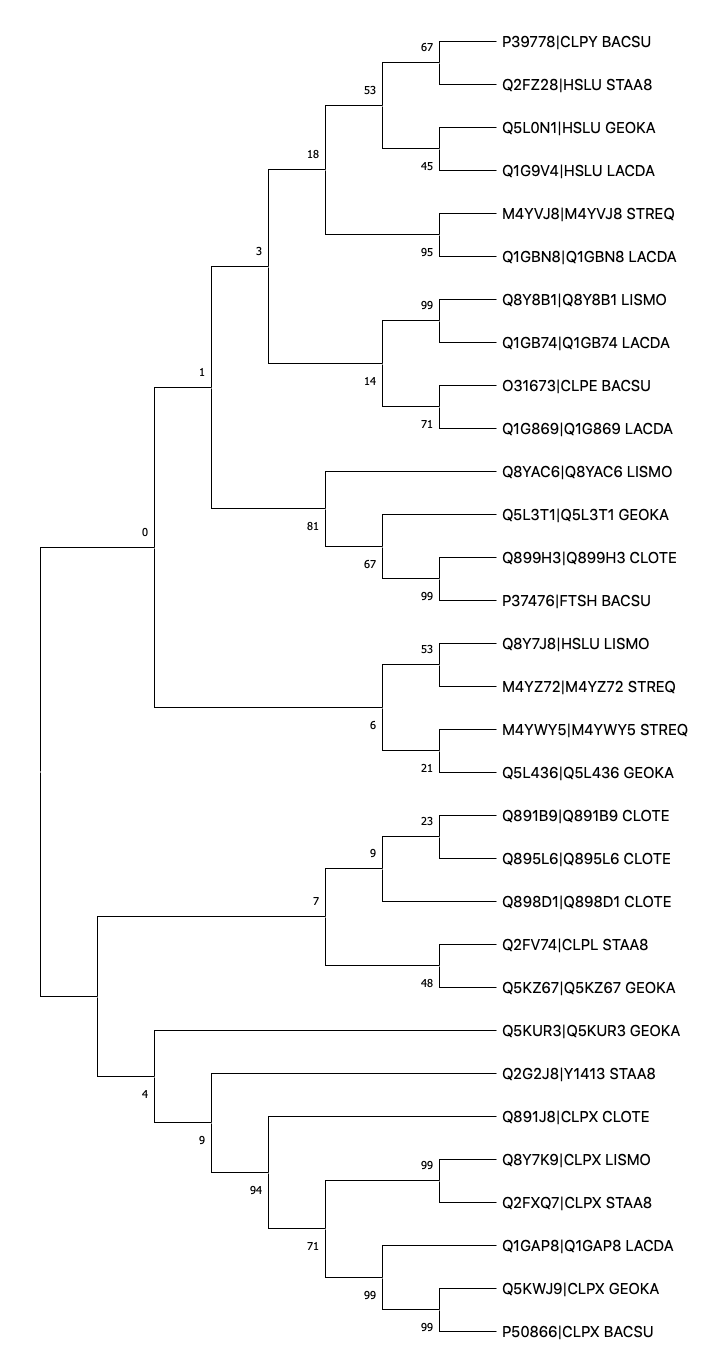
Для построения использовался метод Neighbor-Joining. В результате получилось дерево, представленное на рисунке 1:
((((((P39778|CLPY_BACSU,Q2FZ28|HSLU_STAA8),(Q5L0N1|HSLU_GEOKA,Q1G9V4|HSLU_LACDA)),(M4YVJ8|M4YVJ8_STREQ,Q1GBN8|Q1GBN8_LACDA)),((Q8Y8B1|Q8Y8B1_LISMO,Q1GB74|Q1GB74_LACDA),(O31673|CLPE_BACSU,Q1G869|Q1G869_LACDA))),(Q8YAC6|Q8YAC6_LISMO,(Q5L3T1|Q5L3T1_GEOKA,(Q899H3|Q899H3_CLOTE,P37476|FTSH_BACSU)))),((Q8Y7J8|HSLU_LISMO,M4YZ72|M4YZ72_STREQ),(M4YWY5|M4YWY5_STREQ,Q5L436|Q5L436_GEOKA)),((((Q891B9|Q891B9_CLOTE,Q895L6|Q895L6_CLOTE),Q898D1|Q898D1_CLOTE),(Q2FV74|CLPL_STAA8,Q5KZ67|Q5KZ67_GEOKA)),(Q5KUR3|Q5KUR3_GEOKA,(Q2G2J8|Y1413_STAA8,(Q891J8|CLPX_CLOTE,((Q8Y7K9|CLPX_LISMO,Q2FXQ7|CLPX_STAA8),(Q1GAP8|Q1GAP8_LACDA,(Q5KWJ9|CLPX_GEOKA,P50866|CLPX_BACSU))))))));
Я попыталась выделить ветви, отделяющие конкретные таксономические группы, в данном случае все рассматриваемые объекты принадлежат группе Firmicutes. В целом, узел 53 отделяет только представителей Bacilli, но, учитывая наличие таковых в других частях дерева, никаких особых закономерностей я не обнаружила.
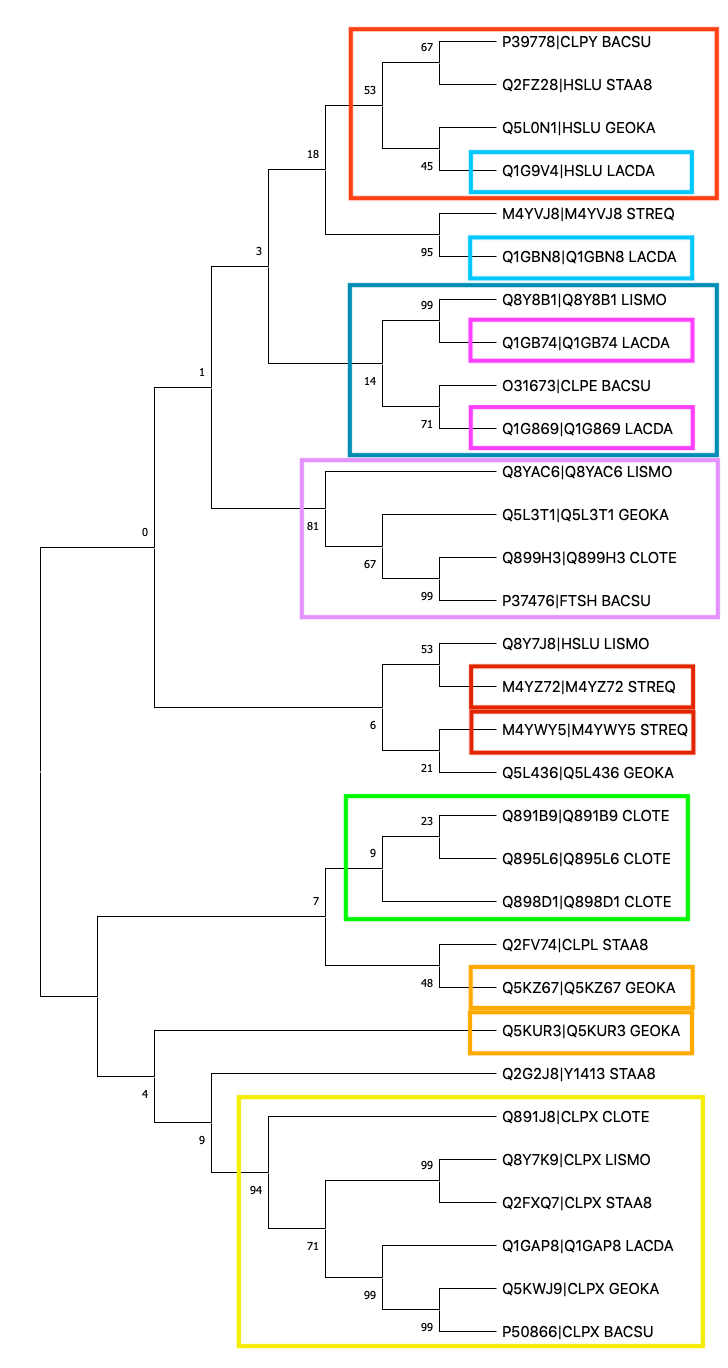
На рисунке 2 я цветом выделила 3 группы ортологов (бирюзовая, лавандовая и желтая рамки) и несколько пар паралогов (голубая, розовая, красная, оранжевая пары рамок). Также ярко-зеленым вылелена группа из трех белков-паралогов.
Три пары ортологов: Q5KWJ9|CLPX GEOKA и Q2FXQ7|CLPX STAA8; Q5L0N1|HSLU GEOKA и Q2FZ28|HSLU STAA8; Q8YAC6|Q8YAC6 LISMO и Q899H3|Q899H3 CLOTE
Три пары паралогов: Q1G9V4|HSLU LACDA и Q1GBN8|Q1GBN8 LACDA; Q1GB74|Q1GB74 LACDA и Q1G869|Q1G869 LACDA; M4YZ72|M4YZ72 STREQ и M4YWY5|M4YWY5 STREQ
Первая и вторая ортологические группы содержат только представителей класса Bacilli. В третью попали представители Bacilli и один организм класса Clostridia, аналогично с четвертой группой. Я не вижу особой связи между таксономическими группами и топологией дерева.
На рисунке 3 показаны три полученные схлопнутые ветви.
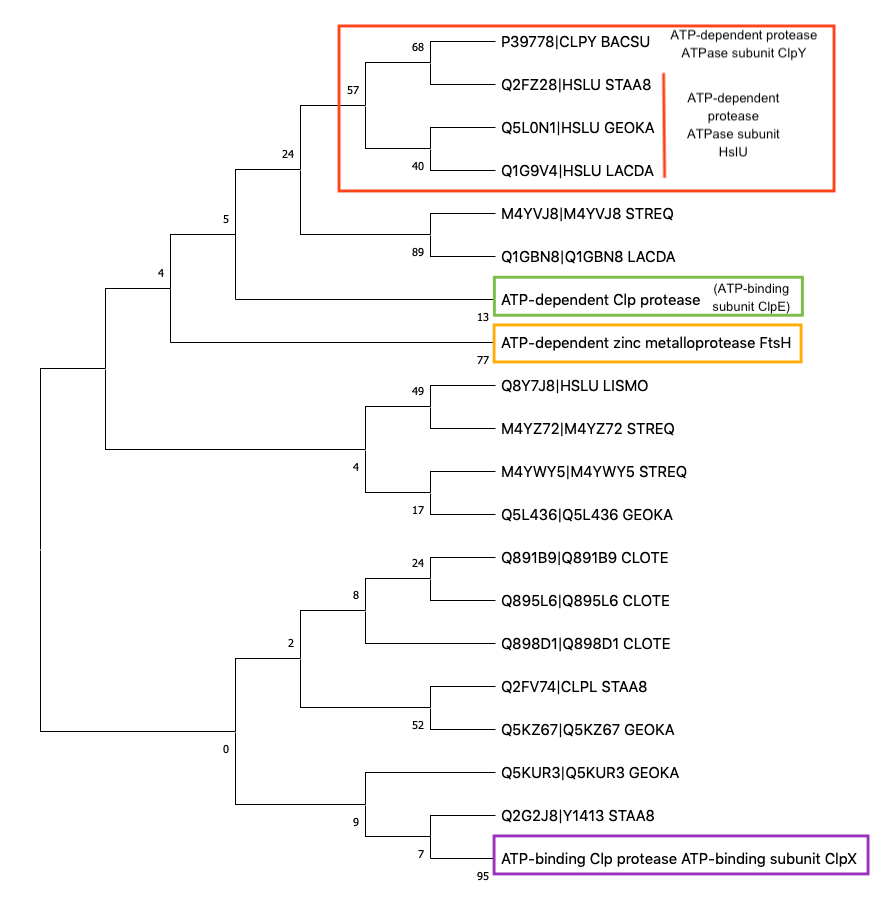
В первой группе оказались три ATP-dependent protease ATPase subunit HsLu и одна subunit ClpY. Обе субъединицы имеют шаперонную активность, являются АТФ-зависимыми. Судя по информации из UniProt, ClpY принадлежит "ClpX chaperone family, HsIU subfamily", так что появление их в одной ортологической группе ничему не противоречит. HsLu присутствует в 3 из 7 представленных видов (класс Bacilli), ClpY - в одном (B. subtilis).
Две из трех оставшихся групп имеют непосредственное отношение к АТФ-зависимой эндопептидазе Clp ('ATP-dependent Clp protease ATP-binding subunit ClpE' и 'ATP-dependent Clp protease ATP-binding subunit ClpX'). ClpE и ClpX обозначены как "шаперонные субъединицы". Конкретно ClpX осуществляет АТФ-зависимое разрешение третичной структуры белков.
ClpE присутствует в 4 из 7 представленных видов (класс Bacilli), ClpX - в 6 (класс Bacilli и представитель класса Clostridia).
'ATP-dependent zinc metalloprotease FtsH' - это также АТФ-зависимая, цинковая металлпептидаза. По информации из UniProt, осуществляет контроль мембранных белков. Присутсвует у всех бактерий (объединяются только принадлежностью к Firmicutes).