Практикум №8. Поиск по сходству (blast)
1. Определение функции нуклеотидной последовательности из практикума 6 и таксона организма, которому она принадлежала.
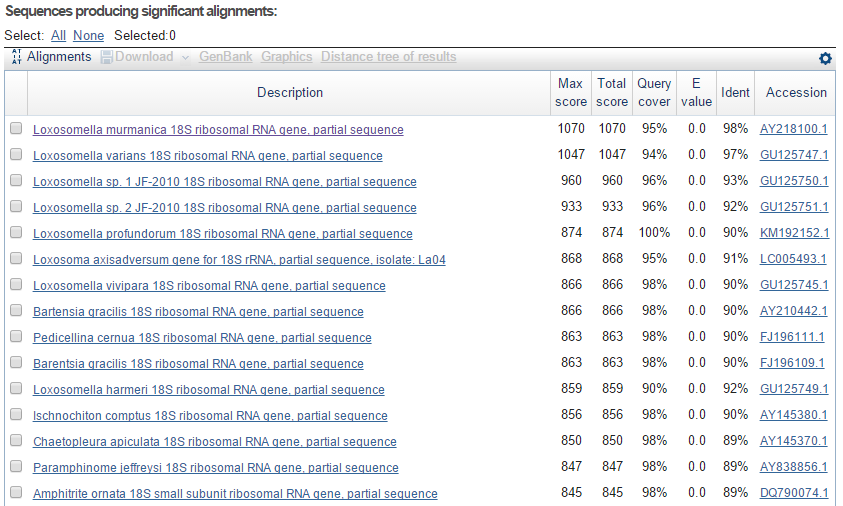
Рис.1 Список находок по результатам работы blastn.
- Так как параметры находок (E-value 0.0, процент идентичных участков 89-98) указывают на высокое сходство с исходной последовательностью, можно сделать вывод, что данная последовательность кодирует 18S рРНК
- Вид организма - Loxosomella murmanica.
- Совпадения для лучшей находки - 621 из 662 (или 2 замены на 100 п.н.)
- Совпадения с представителем другого рода, но того же семейства - 590 из 658 (или 10 замен на 100 п.н.)
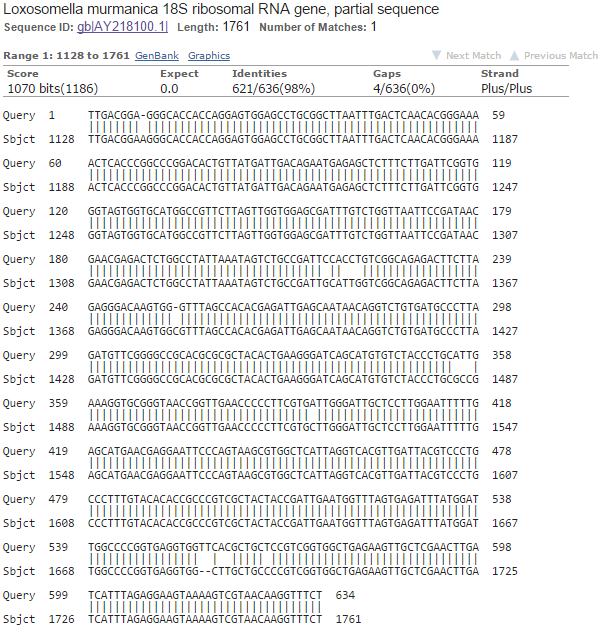
Рис.2 Выравнивание для лучшей находки

Рис.3 Выравнивание с представителем того же семейства, но другого рода.
Сравние списков находок нуклеотидной последовательности 3-я разными алгоритмами blast
Поиск осуществлялся среди организмов, принадлежащих к родам Loxosomella и Glycera одного семейства.
Ссылка на исходную последовательностьBlastn
- Количество находок - 71
- E-value худшей находки - 0.89; Query cover - 1%; Сходство - 100%
- Странные находки, отличающиеся от исходной последовательности и невстречающиеся при поиске иными алгоритмами
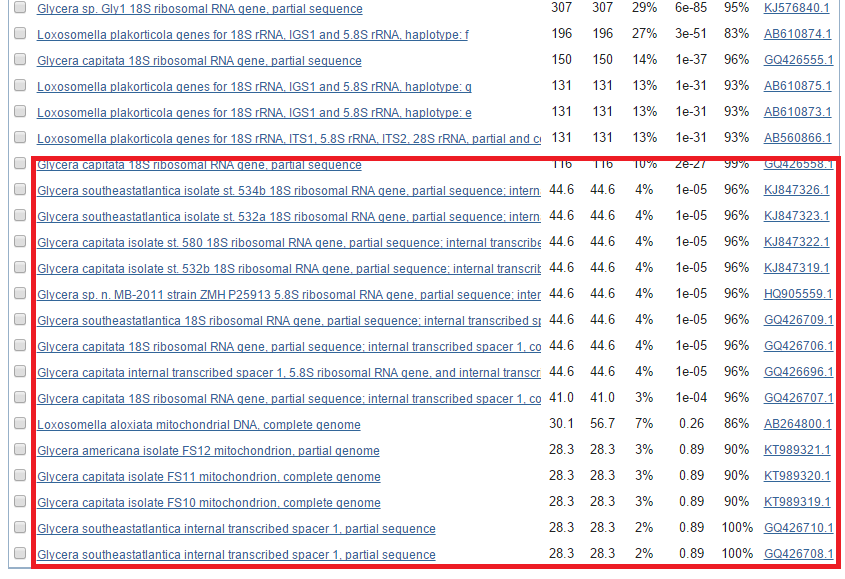
Рис.4 Выравнивание blastn, красным выделены странные находки.
Megablast
- Количество находок - 56
- E-value - 1e-28; Query cover - 10%; Сходство - 99%
- Некоторые последовательности, найденные с помощью blastn и Discontiguous megablast, были упущены
Discontiguous megablast
- Количество находок - 56
- E-value - 2e-27; query cover - 10%; Сходство - 99%
- Большинство находок имеет высокое сходтво с исходной
Выводы
Алгоритмы blastn и megablast устроены таким образом, что основным критерием для построение выравнивания является полное совпадение участка какой-то длины, с каким-то участком исходной последовательности. Поэтому megablast упускает последовательности, не удовлетворяющие критерию, хотя они и имеют высокое сходство с исходной. Не очень высокие штрафы за гэпы в blastn приводят к находкам, имеющим высокое сходство, но только на очень небольшом участке. Дополнительным критерием поиска в discontiguous megablast является указание длины несовпадающих участков, начиная с которой берутся штрафы за увеличение гэпов. В результате discontiguous megablast находит наиболее сходные последовательности.
3 (3.1). Проверка наличия гомологов 5 белков человека в геноме Drosophila simulans.
HSP7C_HUMAN
- Один из белков теплового шока, принимает участие в передаче клеточных сигналов, апоптозе, клеточной дифференциации и свертывание белков.
- Всего 6 находок. Гомологом можно назвать только 1.
- Параметры лучшей находки: E-value - 0.0, query cover - 94%, Ident - 81%
TERT_HUMAN
- Каталитическая субъединица теломеразы, осуществляет обратную транскрипцию шаблонной РНК
- 1 находка, гомологом её нельзя назвать.
- Её параметры: E-value - 0.80, query cover - 8%, Ident - 28%
CISY_HUMAN
- Катализирует реакцию конденсации ацетата и оксалоацетата, в результате чего образуется цитрат
- 3 находки, гомологов - 0
- Лучшая находка: E-value - 1e-58, query cover - 92%, Ident - 41%
RPB1_HUMAN
- Субъединица РНК-полимеразы 2; В сочетании с рядом других субъединиц образует ДНК-связывающий домен полимеразы.
- Всего 2 находки. 1 гомолог.
- Лучшая находка: E-value - 0, query cover - 69%, Ident - 79%
PAB2_HUMAN
- Связывается с поли-А сайтами; необходим для эффективной полимеризации поли-А участков.
- 5 находок, гомологов - 0.
- Лучшая находка: E-value - 3e-44, query cover - 50%, Ident - 64%
4. Классификация геномов родственных вирусов по сходству последовательностей.
Была составлена база данных из полных геномов пяти вирусов:
Bean golden mosaic virus NC_004042.1
Tomato rugose mosaic virus AF291705.1
Macroptilium yellow vein virus JN419021.1
Soybean chlorotic spot virus JX122965.1
Sida golden mosaic Honduras virus Y11097.1
Затем при помощи команд
tblastx -query viri.fasta -db viri.fasta -out blast.out -outfmt 7
python revise_blast_7.py -i blast.out -s 25 -l 50 -e 0.01 -o vir.xls
была получена таблица
ссылка
которая была затем отсортирована по сумме произведений процента идентичности на длину перекрывания.
(ниже таблица отсортирована по этим суммам, считавшимся для каждой пары)
Результат (по убыванию):
JN419021.1 NC_004042.1
AF291705.1 NC_004042.1
AF291705.1 JN419021.1
JX122965.1 NC_004042.1
AF291705.1 Y11097.1
JN419021.1 JX122965.1
NC_004042.1 Y11097.1
NC_004042.1 Y11097.1
JN419021.1 Y11097.1
JX122965.1 Y11097.1
AF291705.1 JX122965.1