Для белка-прототипа в PDB-файле описаны остатки 23-323, пространственное расположение описано для остатков 31-254 (нумерация остатков соответствует нумерации в UniProt). Последовательности двух записей белка-прототипа выравнивались и сравнивались в GeneDoc вручную. Выравнивание:
* 20 * 40 * 60 * 80 *
RatPDB : ----------------------MVAFKGVWTQAFWKAVTAEFLAMLIFVLLSVGSTINWGGSENPLPVDMVLISLCFGLSIATMVQCFGHI : 69
RatAqp : MSDGAAARRWGKCGPPCSRESIMVAFKGVWTQAFWKAVTAEFLAMLIFVLLSVGSTINWGGSENPLPVDMVLISLCFGLSIATMVQCFGHI : 91
100 * 120 * 140 * 160 * 180
RatPDB : SGGHINPAVTVAMVCTRKISIAKSVFYITAQCLGAIIGAGILYLVTPPSVVGGLGVTTVHGNLTAGHGLLVELIITFQLVFTIFASCDSKR : 160
RatAqp : SGGHINPAVTVAMVCTRKISIAKSVFYITAQCLGAIIGAGILYLVTPPSVVGGLGVTTVHGNLTAGHGLLVELIITFQLVFTIFASCDSKR : 182
* 200 * 220 * 240 * 260 *
RatPDB : TDVTGSVALAIGFSVAIGHLFAINYTGASMNPARSFGPAVIMGNWENHWIYWVGPIIGAVLAGALYEYVFCPDVELKRRLKEAFSKAAQQT : 251
RatAqp : TDVTGSVALAIGFSVAIGHLFAINYTGASMNPARSFGPAVIMGNWENHWIYWVGPIIGAVLAGALYEYVFCPDVELKRRLKEAFSKAAQQT : 273
280 * 300 * 320
RatPDB : KGSYMEVEDNRSQVETEDLILKPGVVHVIDIDRGDEKKGKDSSGEVLSSV : 301
RatAqp : KGSYMEVEDNRSQVETEDLILKPGVVHVIDIDRGDEKKGKDSSGEVLSSV : 323
Парное выравнивание последовательности исследуемого белка и последовательности белка-прототипа из PDB построено программами water и needle пакета EMBOSS. Характеристики выравниваний: идентичность 94.7%, сходство 97.3% (water) и 88.2%, 90.7% (needle). Глобальное выравнивание (needle) импортировано в файл marking.msf
По идентификатору PDB белка-прототипа найдено описание ориентации белка в мембране в БД OPM (Orientations of Proteins in Membranes database)
В файле marking.msf третьей строкой вставлена последовательность с названием "OPM" и разметкой ТМ сегментов ("H" - трансмембранные сегменты, "+" - цитоплазматические, "-" - остальные, "?" - не предсказано).TM сегменты: 1(34-56), 2(70-88), 3(98-107), 4(112-136), 5(156-178), 6(189-203), 7(214-223), 8(231-252).
С помощью сервера
TMHMM
была предсказана топология исследуемого белка.
Страничка с результатом предсказания.
К последовательностям в файле marking.msf добавлена искусственная последовательность TMHMM, отражающая результаты данного предсказания.
Готовое выравнивание:
RatPDB ----------------------MVAFKGVWTQAFWKAVTAEFLAMLIFVLLSVGSTINWG
SheepAqp MSDRPAARRWGKCGPLCTRESIMVAFKGVWTQTFWKAVTAEFLAMLIFVLLSLGSTINWG
OPM ??????????????????????+++++++++++HHHHHHHHHHHHHHHHHHHHHHH----
TMHMM ??????????????????????+++++++++++++++HHHHHHHHHHHHHHHHHHHHHHH
RatPDB GSENPLPVDMVLISLCFGLSIATMVQCFGHISGGHINPAVTVAMVCTRKISIAKSVFYIT
SheepAqp GAEKPLPVDMVLISLCFGLSIATMVQCFGHISGGHINPAVTVAMVCTRRISIAKAVFYIA
OPM ---------HHHHHHHHHHHHHHHHHHH+++++++++HHHHHHHHHH++++HHHHHHHHH
TMHMM ---------HHHHHHHHHHHHHHHHHHHHHHH+++++++++++++++++++HHHHHHHHH
RatPDB AQCLGAIIGAGILYLVTPPSVVGGLGVTTVHGNLTAGHGLLVELIITFQLVFTIFASCDS
SheepAqp AQCLGAIIGAGILYLVTPPSVVGGLGVTTVHRNLSAGHGLLVELIITFQLVFTIFASCDS
OPM HHHHHHHHHHHHHHHH-------------------HHHHHHHHHHHHHHHHHHHHHHH++
TMHMM HHHHHHHHHHHHHH-------------------HHHHHHHHHHHHHHHHHHHHHHH++++
RatPDB KRTDVTGSVALAIGFSVAIGHLFAINYTGASMNPARSFGPAVIMGNWENHWIYWVGPIIG
SheepAqp KRTDVTGSIALAIGISVAIGHLFAINYTGASMNPARSFGPAVIMGNWENHWIYWVGPIIG
OPM ++++++++HHHHHHHHHHHHHHH----------HHHHHHHHHH-------HHHHHHHHHH
TMHMM ++++++++HHHHHHHHHHHHHHHHHHHHHHH-------------------HHHHHHHHHH
RatPDB AVLAGALYEYVFCPDVELKRRLKEAFSKAAQQTKGSYMEVEDNRSQVETEDLILKPGVVH
SheepAqp AVLAGGLYEYVFCPDVELKRRFKEAFSKAAQQTKGSYMEVEDNRSQVETDDLILKPGVVH
OPM HHHHHHHHHHHH++++++++++++++++++++++++++++++++++++++++++++++++
TMHMM HHHHHHHHHHHHH+++++++++++++++++++++++++++++++++++++++++++++++
RatPDB VIDIDRGDEKKGKDSSGEVLSSV
SheepAqp VIDIDRGEEKKGKDPSGEVLSSV
OPM +++++++++++++++++++++++
TMHMM +++++++++++++++++++++++
В формате Clustal Результаты предсказания топологии мембранного белка аквапорина из овцы
| Число а.к. остатков | |
| Всего а.к. остатков | рассматривали фрагмент 23-323 |
| Остатки, предсказанные как локализованные в мембране (всего) | 138 |
| Правильно предсказали (true positives, TP) | 119 |
| Предсказали не то, что нужно (а.о. предсказаны как мембранные, а по данным ОРМ таковыми не являются, false positives, FP) | 19 |
| Правильно не предсказали ( не предсказаны, и по данным ОРМ не находятся в мембране, true negatives, TN) | 135 |
| Не предсказали то, что нужно (остатки по данным ОРМ находятся в мембране, false negatives, FN) | 28 |
| Чувствительность (sensivity) = TP / (TP+FN) | 81,0% |
| Специфичность (specificity) = TN / (TN+FP) | 87,7% |
| Точность (precision) = TP / (TP+FP) | 86,2% |
| Сверхпредсказание = FP/ (FP+TP) | 13,8% |
| Недопредсказание = FN / (TN+FN) | 17,2% |
Программа не предсказала два трансмембранных участка, расположение которых отличается от расположения остальных участков: они не пронзают мембрану насквозь, поэтому фрагменты справа и слева от данных трансмембранных участков расположены по одну сторону от мембраны (см. рис.). Именно поэтому непредсказание этих участков не повлекло нарушения ориентации остальных фрагментов. Остальные ТМ участки предсказаны довольно точно.
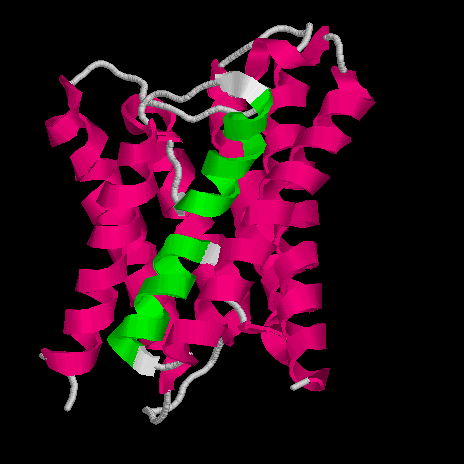
Зеленым обозначены непредсказанные участки.
©Хайруллин Альберт