Нуклеотидный Blast
Продолжаю изучать организм Maccaca mulatta
Выбор CDS
Нуклеотидная запись: NC_041754.1
Координаты участка: 184316725...184321116. В этом участке ген TMCO2, который кодирует белок transmembrane abd coiled-coil domains 2. Он содержит трансмембранные участки и спиральные катушки.
Длина CDS: 4392 нуклеотида
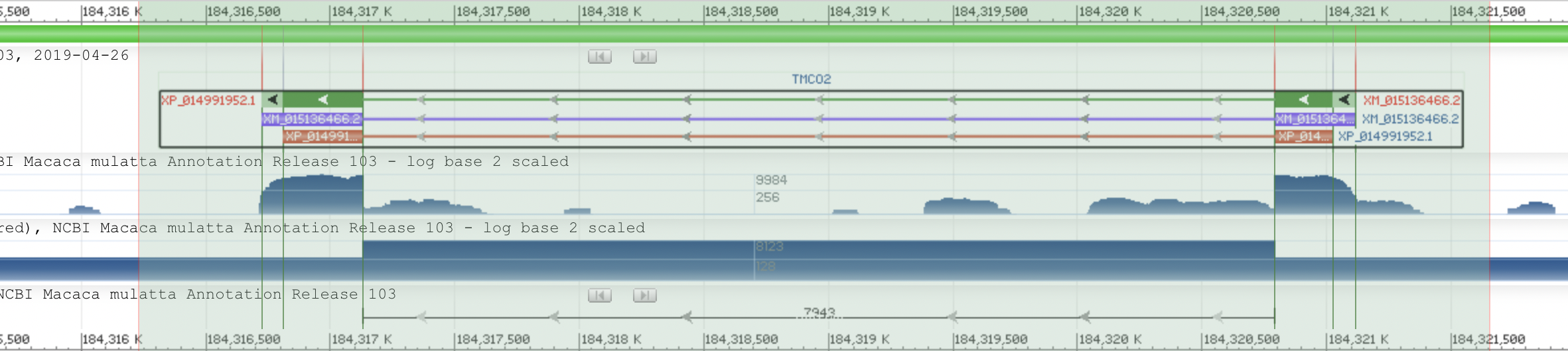
Зеленым на картинке изображен ген, фиолетовым и красным- транскрипты. Ниже (синим)- покрытие экзонов и интронов RNAseq. Самая нижняя полоска- описание интронов.
Сравнивать я буду с классом Aves, так как с изучаемым видом они относятся к одному подтипу Vertebrata.
Поиск Blast
blastn: нашел 86 последовательности (длина слова 15; при длине 11 выдавал больше 250). Blastn позволяет искать гомологичные нуклеотидные последовательности (ДНК, РНК) чтобы, например, найти мутации.
megablast: нашел 71 последовательность (длина слова 16). Megablast используют для поиска сходств между короткими нуклеотидными последовательностями и большими геномными бд. Его можно использовать, например, для поиска последовательностей близкородственных организмов (например, можно понять таксономию).
blastx: нашел 8 последовательностей (длина слова 3). Blastx выравнивает нуклеотидные последовательности с последовательностями аминокислот, позволяя найти кодирующие последовательности или мутации.
tblastx: не нашел ничего ("CPU usage limit was exceeded") при всех вариантах параметров. Tblastx является вариацией blastx, которая позволяет искать скрытых гомологов за счет сопоставления различных рамок перевода. С помощью него можно найти фреймшиты, возникшие вследствие мутаций.
[Далее я буду работать с геномом организма Kogia breviceps, так как у меня не хватает места для распаковки выбранного :)]
Индексирую геном
makeblastdb -in GCF_026419965.1_mKogBre1_haplotype_1_genomic.fna -dbtype nucl
Буду использовать алгоритм blastn для выравнивания, так как используем совсем неродственные последовательности.
С помощью blastn выравниваю мой геном с 16S rRNA (в составе малой субъединицы рибосомы) и 23S rRNA (в составе большой субъедницы рибосомы) E. Coli. Они выполняют структурную функцию, 16S рРНК узнает последовательность Шайн-Дальгарно для начала трансляции, 23S рРНК в составе Р-центра рибосомы, в котором происходит пептидил-трансферазная реакция.
blastn -task blastn -query 16Srna.fa -db GCF_026419965.1_mKogBre1_haplotype_1_genomic.fna -out blastn_16S.out -evalue 0.05 -outfmt 7
blastn -task blastn -query 23Srna.fa -db GCF_026419965.1_mKogBre1_haplotype_1_genomic.fna -out blastn_23S.out -evalue 0.05 -outfmt 7
evalue 0.05 контролирует, чтобы находки уж совсем плохими не были, -outfm 7 вывод в виде таблицы
Результаты:
16S rRNA: 133 находки
23S rRNA: 519 находок
Находок так много из-за того, что, скорее всего, сборка не хромосомного уровня, но при этом участки (это видно по координатам) не повторяются даже для одного скаффолда, из чего мы можем сделать вывод, что для 16S гомологами являются все 133 находки. В случае 23S, находки имеют разные коородинаты в одном скаффолде, так как разделены низкоконсервативными участками, которые не выравниваются. При этом в каждом скаффолде- один гомолог.