Трансмембранные белки
Сравнение предсказаний в бета-листовом белке
Выбрала в ОРМ следующий белок:
type: transmembrane
class: β-barrel transmembrane
superfamily: Sugar porins
family: Malptoporin-like proteins
name: Maltporin
uniprot: LAMB_ECOLI
pdb: 1af6
функция: Участвует в транспорте мальтозы и мальтодекстринов, важен для транслокации мальтодекстринов с тремя и более глюкозильных фрагментов. Гидрофобный путь атоматических остатков служаит для направления транспорта сахаров. Является реуцептором для некоторых бактериофагов.
Скачала полную последовательность из pdb, там оказалось три одиаковых последовательности, поэтому в DeepTMHMM загрузила только одну и получила 17 предсказанных трансмембранных участков:
OPM|DeepTMHMM
12-16|2-13
39-48|39-47
58-68|59-67
75-88|79-88
98-103|99-105
125-132|124-132
138-146|138-145
170-179|171-177
185-194|186-193
213-221|214-221
227-235|228-234
269-278|271-278
284-293|285-292
305-314|307-314
320-329|321-327
343-352|344-352
361-370|357-368
411-420|413-420
Ссылка на выдачу в формате gff3Количество предсказанных разными алгоритами петель одинаково, но при этом все отличаются в основном на 1-2 координаты, но есть и значительные различия (подчеркнуто).
Вероятно, дело в том, что DeepTMHMM все-таки предсказывает по аминокислотной последовательности, а в OPM содержатся уже проверенные данные.
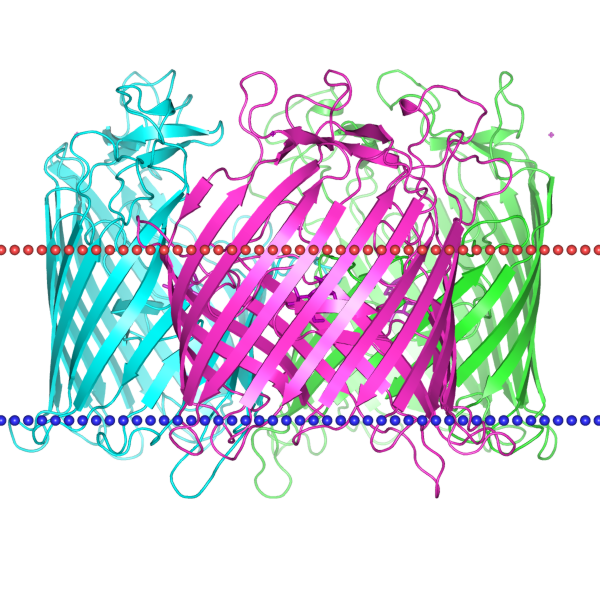
Рис 1. Структура белка lamb_ecoli, pdb entry: 1af6
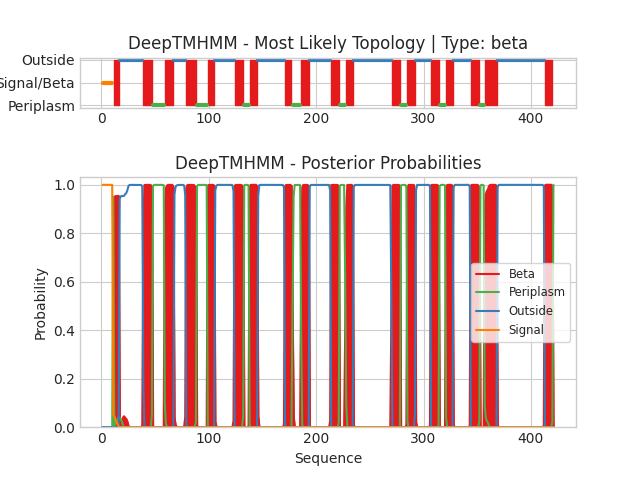
Рис 2. Вероятности и топология для белка с бета-листами
Сравнение предсказаний в альфа-спиральном белке
type: transmembrane
swiss-prot AC: Q48AB7 (CYOE_COLP3)
name: Protoheme IX farnesyltransferase
organism: Colwellia psychererythraea
функция: превращение гема В в гем О путем замены винильной группы на 2 углероде порфиринового кольца гема В на гидроксиэтилфарнезильную группу.
Загружаю последоваттолькость в DeepTMHMM.
Ссылка на выдачуНашел 9 трансмембранных α-спиралей.
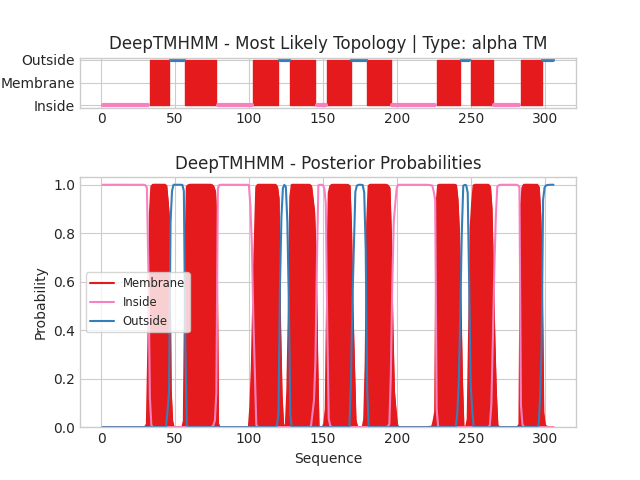
Рис 3. Вероятности и топология для белка с альфа-спиралями
Далее загружаю файл с описанием структуры в PMM 3.0 (дефолтные значения и type of membrane: Gram-negative bacteria inner membrane) для предсказания структуры Alpha Fold. Он тоже предсказал 9 трансмембранных участков:
DeepTHMM|PMM 3.0
33-46|32-52
57-78|58-78
103-120|103-123
128-145|126-146
153-169|153-173
180-196|180-200
227-243|227-247
250-265|249-269
284-298|278-298
Различия в координатах снова есть, в AlphaFold все петли длиной 20 аминокислот, а в DeepTHMM координаты какие-то более "живые", но при этом каких-то критических различий нет, скорее размытие границ.