|
Отобранные бактерии
| Название | Мнемоника |
| Bacillus subtilis | BACSU |
| Clostridium tetani | CLOTE |
| Enterococcus faecalis | ENTFA |
| Lactobacillus delbrueckii | LACDA |
| Staphylococcus epidermidis | STAES |
| Listeria monocytogenes | LISMO |
| Geobacillus kaustophilus | GEOKA |
| Thermoanaerobacter tengcongensis | THETN |
Скобочная формула дерева
((CLOTE,THETN),((LACDA,ENTFA),(STAES,(LISMO,(BACSU,GEOKA)))));
Изображение дерева
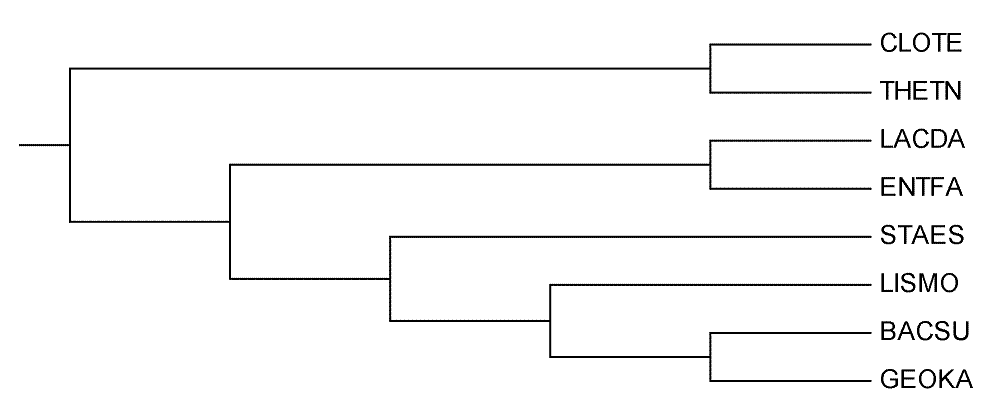
Ветви дерева
Дерево содержит шесть нетривиальных веток:
1) {BACSU, GEOKA} vs {LISMO, STAES, LACDA, ENTFA, THETN, CLOTE}
2) {GEOKA, BACSU, LISMO} vs {STAES, LACDA, ENTFA, THETN, CLOTE}
3) {GEOKA, BACSU, LISMO, STAES} vs {LACDA, ENTFA, THETN, CLOTE}
4) {LACDA, ENTFA} vs {STAES, LISMO, BACSU, GEOKA, THETN, CLOTE}
5) {LACDA, ENTFA, STAES} vs {LISMO, BACSU, GEOKA, THETN, CLOTE}
6) {LACDA, ENTFA, STAES, LISMO} vs {BACSU, GEOKA, THETN, CLOTE}
|
Таксономические ветви
1) Ветка THETN-CLOTE: ветка отличается от остальных классом: Clostridia против Bacilli
2) Ветка LACDA-ENTFA: отличается от оставшихся порядком: они относятся к Lactobacillaes, остальные же - Bacillaes
3) Ветка GEOKA-BACSU: в отличие от оставшихся LISMO и STAES, относятся к одному семейству: Bacillaceae.
Вывод: Образование веток действительно связано с таксонами и действительно обосновано.
Белок
Я выбрал Рибосомальный белок S7 (далее RS7). Выравнивание строится mafftом.
Реконструкция дерева программой fprotpars
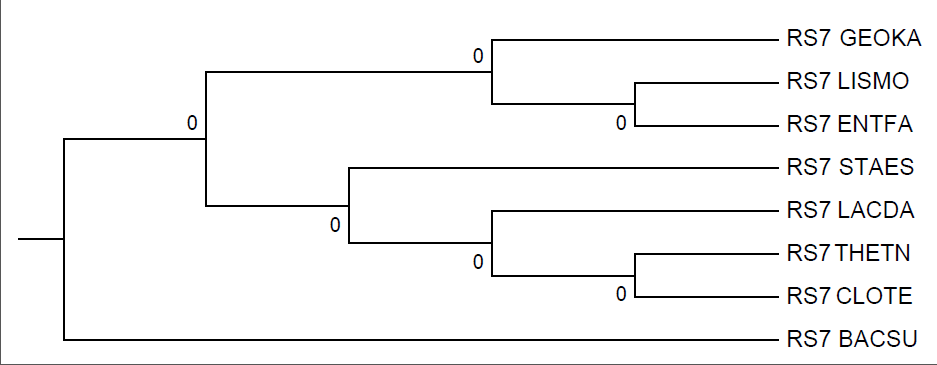
Консервативна только ветвь {CLOTE, THETN}. Прочие ветви сильно изменились. Теперь не существует возможности выделить такую ветвь, чтобы RS7_BACSU было в паре лишь с одним другим белком; в ветви {LACDA,ENTFA} первый таксон был заменен на LISMO. 1 правильная ветвь
Матрица расстояний:
| Название | BACSU | CLOTE | ENTFA | LACDA | STAES | LISMO | GEOKA | THETN |
| BACSU | 0 | 0.384968 | 0.163836 | 0.335291 | 0.180264 | 0.166108 | 0.134584 | 0.357081 |
| CLOTE | 0.384968 | 0 | 0.401406 | 0.400373 | 0.402037 | 0.391701 | 0.377745 | 0.381170 |
| ENTFA | 0.163836 | 0.401406 | 0 | 0.305718 | 0.241060 | 0.182599 | 0.201968 | 0.370512 |
| LACDA | 0.335291 | 0.400373 | 0.305718 | 0 | 0.383471 | 0.322566 | 0.325393 | 0.440637 |
| STAES | 0.180264 | 0.402037 | 0.241060 | 0.322566 | 0 | 0.242088 | 0.238645 | 0.358168 |
| LISMO | 0.166108 | 0.391701 | 0.182599 | 0.383471 | 0.242088 | 0 | 0.183536 | 0.408938 |
| GEOKA | 0.134584 | 0.377745 | 0.201968 | 0.325393 | 0.238645 | 0.183536 | 0 | 0.291746 |
| THETN | 0.357081 | 0.381170 | 0.370512 | 0.440637 | 0.358168 | 0.408938 | 0.291746 | 0 |
Условие аддитивности: {A,B}+{C,D}={A,C}+{B,D}>{A,D}+{B,C}
Проверим для {BACSU,GEOKA} и {CLOTE,THETN}:
{A,B}=0,134584; {C,D}=0,381170. Sum=0,515754
{A,C}=0,384968; {B,D}=0,291746. Sum=0,676714
{A,D}=0,357081; {B,C}=0,377745. Sum=0,734826
Отклонение от аддитивности: 0,058112.
Условие ультраметричности: d(A,B) > d (B,C), то d (A,C) = d (A,B)
Пусть A = LACDA; B = ENTFA; C = LISMO.
{A,B}=0,305718; {B,C} = 0,182599; {A,C}=0,322566;
Отклонение от ультраметричности: 0,016848
FNEIGHBOR
Neighbor-Joining
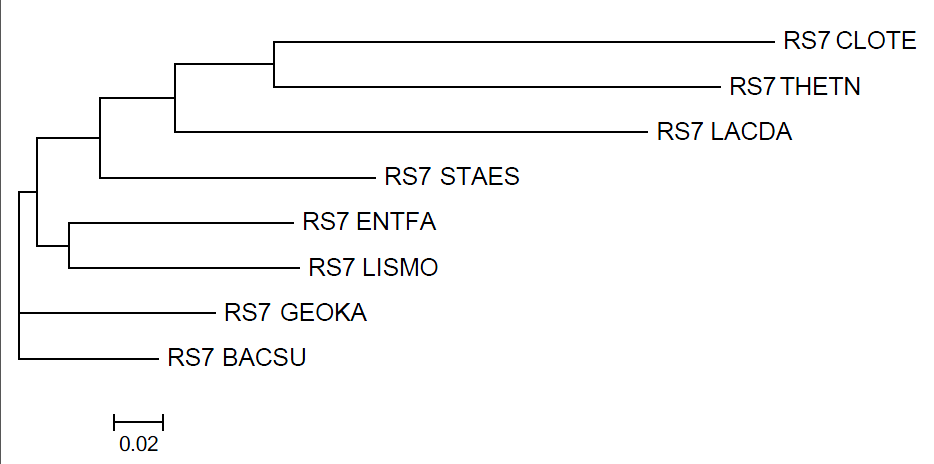
Отличие от правильного дерево в замене ветви {ENTFA,LACDA} на ветвь {ENTFA,LISMO} (как было и в fprotpars), разрушены ветви {{ENTFA,LACDA},{THETN,CLTOTE}}, {LISMO,{GEOKA,BACSU}} . 2 правильные ветви: {BACSU,GEOKA} и {CLOTE,THETN}.
UPGMA
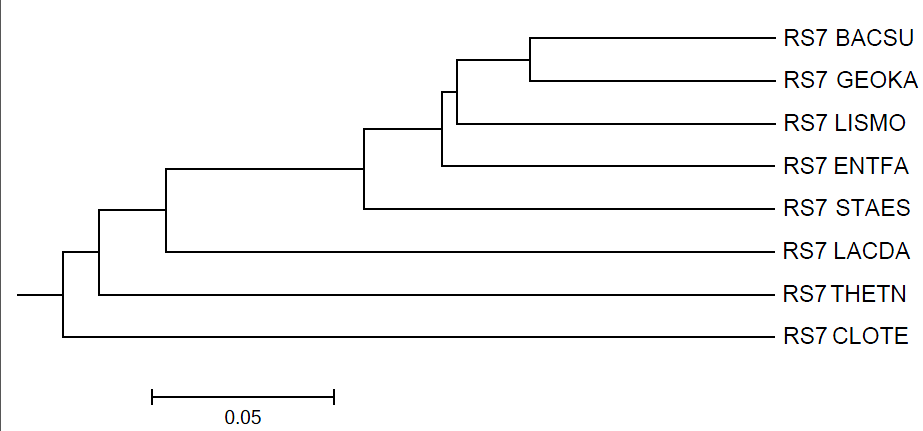
Дерево построенно последовательно: с каждым новым ветвлением отделяется только 1 белок. По структуре как дерево, построенное по методу Neighbor-Joining, исчезла ветвь {ENTFA,LISMO}, но образовалась ветвь {BACSU,GEOKA,LISMO}. 3 верные ветви: {{BACSU,GEOKA},LISMO}, {BACSU,GEOKA}, {CLOTE,THETN}
Алгоритмы реконструкции деревьев
Укоренение в среднюю точку
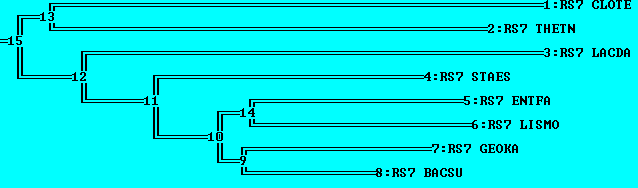
Сравнивая это дерево с тем, что просто было получено без укоренения, видно, что с укорененным деревом удобнее работать, вдобавок стало более хорошо видно отделение {RS7_CLOTE,RS7_THETN} от прочих групп, поскольку укоренение произошло в ветвь {THETN,CLOTE} (что совпадает с оригинальным деревом).
Использование внешней группы
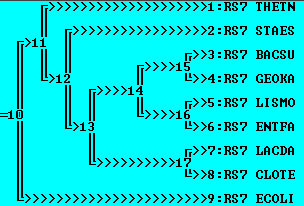
Изменения здесь намного радикальнее. Во-первых, при добавлении внешней группы оказалось, наиболее близкий к ней белок - RS7_THETN. Также от пар {BACSU, GEOKA} и {ENTFA, LISMO} отдалился белок RS7_STAES, образовалась ветвь {RS7_LACDA,RS7_CLOTE}, причем эта ветвь стала ближе к {{BACSU,GEOKA},{LISMO,ENTFA}}. Укоренение прошло в ветвь THETN. 1 правильная ветвь {BACSU,GEOKA}
Бутстрэп
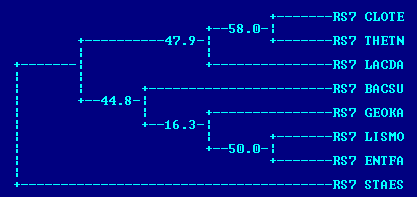
Применяя этот метод получения филогенетического дерева, была получена 1 правильная ветвь - {THETN,CLOTE} (та же самая ветвь была единственной правильной при использовании fprotpars без бутстрэпа;
Построение дерева по нуклеотидным последовательностям
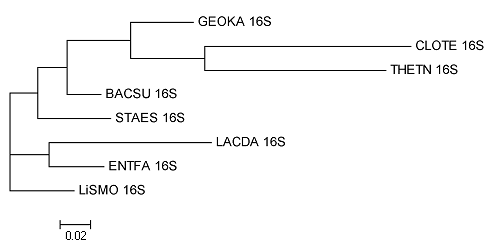
Построено с помощью fdnaml. Вновь была разрушена ветвь {BACSU,GEOKA}, хотя для RS7 {BACSU,GEOKA} довольно близки. Были разрушены оригинальные ветви {{BACSU,GEOKA},LISMO} и {STAES,{LISMO,{GEOKA,BACSU}}} . Консервативна лишь пара {THETN,CLOTE}, также верна по сравнению с оригиналом ветвь {ENTFA,LACDA}. Таким образом только 2 ветви правильны: {CLOTE,THETN} и {LACDA,ENTFA}.
Выравнивание нуклеотидных последователностей
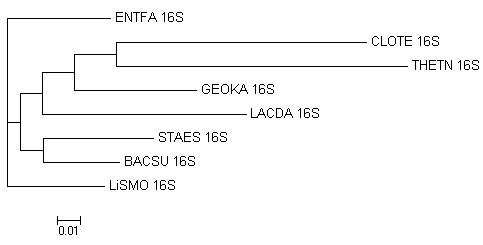
Дерево, построенное с помощью fdnadist и fneighbor. Единственная верная ветвь - {CLOTE,THETN}. Для сравнения, при использовании fprotdist с fneighbor находятся 2 правильные ветви. Таким образом, лучше использовать белковую последовательность, а если её нет - использовать fdnaml.
Построение и анализ дерева с паралогами

Паралоги: CLPY_BACSU-CLPE_BACSU-CLPX_BACSU-CLPC_BACSU; CLPX_STAES-HSLU_STAES-CLPC_STAES; HSLU_THETN-CLPX_THETN; HSLU_GEOKA-CLPX_GEOKA; HSLU_LISMO-CLPX_LISMO; CLPX_ENTFA-HSLU_ENTFA;
Ортологи: CLPC_STAES-CLPC_BACSY; CLPX_CLOTE-CLPX_THETN; CLPX_GEOKA-CLPX_BACSU; HSLU_ENTFA-HSLU_LACDA; HSLU_GEOKA-CLPY_BACSU; HSLU_THETN-HSLU_LISMO; CLPX_STAES-CLPX_LISMO.
Дерево прекрасно иллюстрирует то, что строить деревья гомологов - это все равно, что строить несколько деревьев сразу для определенного белка разных таксонов. Мы имеем дерево, содержащее древо-ветвь CLPX, древо-ветви HSLU и CLPC. Стоит отметить CLPY_BACSU, каким-то образом попавшее в ветвь HSLU. Правда, отсутствие белка HSLU_BACSU намекает на то, что CLPY выполняет его роль. А так деревья схожи (правда, в одном STAES дальше от BACSU, чем в другом. Также отсутствие CLPX_LACDA не дает нормально оценивать CLPX_ENTFA, как и отсутствие HSLU_CLOTE - HSLU_THETN
Выравнивание в fasta-формате
Назад