Доменная структура белка PPAC_BACSU по данным Pfam
Cхема из Pfam:
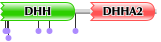
Пояснения к схеме
№
Pfam AC
Pfam ID
Полное название семейства доменов
(по-русски! и желательно с кратким пояснением)
Положение в последовательности белка PPAC_BACSU
Клан
1.
PF01368
DHH
Это предсказанное семейство пирофосфоэстераз (расщепляют эфирную связь между фосфатом и остатком спирта)
Однако белок PPAC_BACSU - пирофосфатаза (утилизирует пирофосфат), то есть предсказание не выполняется. Отмечено, что
этот домен может связывать ионы марганца, что действительно имеет место в белке PPAC_BACSU.
1–147
------------
2.
PF02833 DHHA2 Этот домен разработан на основе белков из второго подсемейства DHH.
Отмечена связь домена с функцией пирофосфатазы. 181–307 -------------
Представленность домена DHHA2 в организмах разных видов
Таксон
Количество белков с доменом DHHA2.
Эукариоты
Зеленые растения
11
Грибы
56
Животные
31
Остальные эукариоты
17
Бактерии
547
Археи
17
DHH встречается в 3650 последовательностях, DHHA2 - в 679. Подавляющее большинство представлено среди бактерий.
Представленность изучаемых доменов в белках Bacillus subtilis
№
PFAM ID
Bacillus subtilis
1.
DHH
5
2.
DHHA2
1
Белков с точно такой же доменной организацией, что и заданный белок нет.
Примеры доменных перестроек.
В DHH-домен часто последовательно (?) встраиваются другие домены.
Q8RI12_FUSNN
 C1UA78_9ACTN
C1UA78_9ACTN
 PPAC_THEMA
PPAC_THEMA
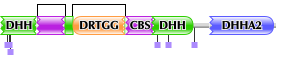 Q748M6_GEOSL
Q748M6_GEOSL
 DHH может и не находится близко от DHHA2, хотя чаще это так.
MGP1_MYCGE
DHH может и не находится близко от DHHA2, хотя чаще это так.
MGP1_MYCGE
 DHHA2 очень редко встречается без DHH.
Q24465_DROME
DHHA2 очень редко встречается без DHH.
Q24465_DROME

Выравнивание-затравка (seed) для N-концевого домена заданного белка.
Сравнение описания мотивов в разных БД.
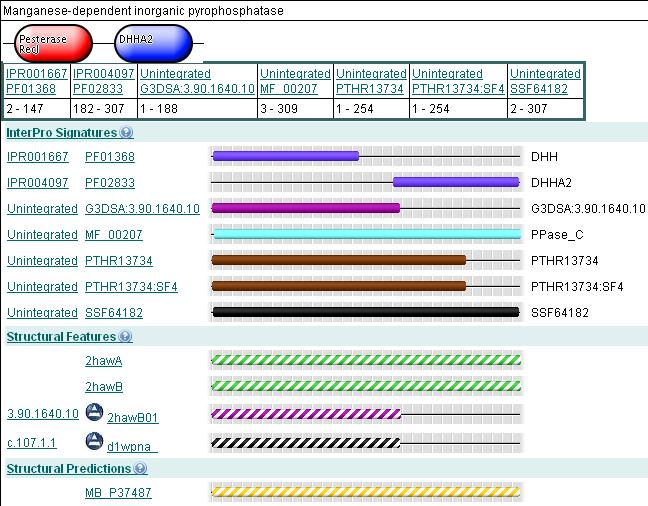
1.Самый короткий мотив - DHHA2. Описан в Pfam. Профиль HMM.
2.Самый длинный мотив - PPase_C. Описан в ExPASy Proteomics Server. HAMAP annotation rule.
3.Много разных структурных подписей интегрировано в InterPro.
4.Границы структурных доменов от границ доменов Pfam отличаются. (из приведенной картинки видно, что они не совпадают).
Cхема из Pfam: |
|||||
| Пояснения к схеме |
|||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов (по-русски! и желательно с кратким пояснением) |
Положение в последовательности белка PPAC_BACSU | Клан |
| 1. | PF01368 | DHH | Это предсказанное семейство пирофосфоэстераз (расщепляют эфирную связь между фосфатом и остатком спирта) Однако белок PPAC_BACSU - пирофосфатаза (утилизирует пирофосфат), то есть предсказание не выполняется. Отмечено, что этот домен может связывать ионы марганца, что действительно имеет место в белке PPAC_BACSU. | 1–147 | ------------ |
| 2. | PF02833 | DHHA2 | Этот домен разработан на основе белков из второго подсемейства DHH. Отмечена связь домена с функцией пирофосфатазы. | 181–307 | ------------- |
|
Таксон
|
Количество белков с доменом DHHA2.
|
|
| Эукариоты | Зеленые растения | 11 |
| Грибы | 56 | |
| Животные | 31 | |
| Остальные эукариоты | 17 | |
| Бактерии | 547 | |
| Археи | 17 | |
DHH встречается в 3650 последовательностях, DHHA2 - в 679. Подавляющее большинство представлено среди бактерий.
| № | PFAM ID | Bacillus subtilis |
| 1. | DHH | 5 |
| 2. | DHHA2 | 1 |
Белков с точно такой же доменной организацией, что и заданный белок нет.
Примеры доменных перестроек.
В DHH-домен часто последовательно (?) встраиваются другие домены. Q8RI12_FUSNNВыравнивание-затравка (seed) для N-концевого домена заданного белка.C1UA78_9ACTN
PPAC_THEMA
Q748M6_GEOSL
DHH может и не находится близко от DHHA2, хотя чаще это так. MGP1_MYCGE
DHHA2 очень редко встречается без DHH. Q24465_DROME

Сравнение описания мотивов в разных БД.

1.Самый короткий мотив - DHHA2. Описан в Pfam. Профиль HMM. 2.Самый длинный мотив - PPase_C. Описан в ExPASy Proteomics Server. HAMAP annotation rule. 3.Много разных структурных подписей интегрировано в InterPro. 4.Границы структурных доменов от границ доменов Pfam отличаются. (из приведенной картинки видно, что они не совпадают).